La IA predice el ritmo de trabajo de las enzimas
"TurNuP supera a los modelos anteriores e incluso puede utilizarse con éxito para enzimas que sólo tienen una baja similitud con las del conjunto de datos de entrenamiento"
Las enzimas desempeñan un papel clave en los procesos metabólicos celulares. Para poder evaluar cuantitativamente estos procesos, los investigadores necesitan conocer el llamado "número de recambio" (abreviado: kcat) de las enzimas. En la revista científica Nature Communications, un equipo de bioinformáticos de la Universidad Heinrich Heine de Düsseldorf (HHU) describe ahora una herramienta para predecir este parámetro para varias enzimas utilizando métodos de IA.
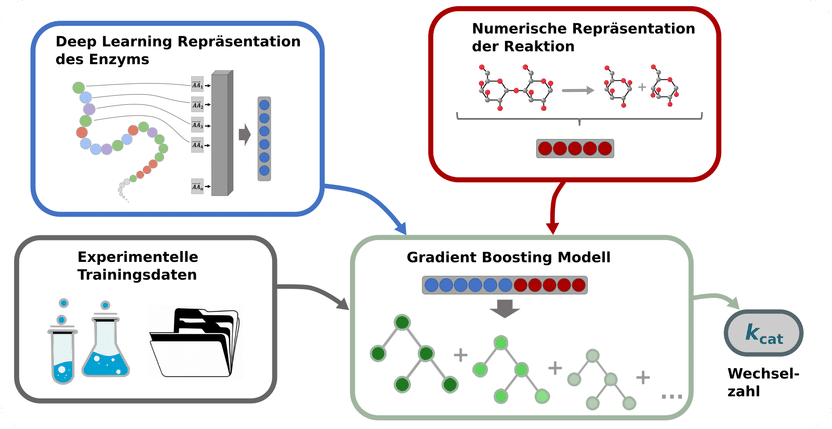
Diagrama esquemático del proceso de predicción de los números de recambio de las reacciones enzimáticas: Las enzimas son secuencias de aminoácidos; estas secuencias se convierten en vectores numéricos, representados como cuadrados grises, que un modelo de aprendizaje profundo transforma en un único vector (arriba a la izquierda). La información sobre las reacciones catalizadas también se convierte en vectores numéricos (arriba a la derecha). Los números de recambio determinados experimentalmente (abajo a la izquierda) se utilizan para entrenar un modelo de refuerzo de gradiente con el fin de predecir el número de recambio kcat (abajo a la derecha). Los modelos de refuerzo de gradiente son un conjunto de múltiples árboles de decisión, representados en diferentes tonos verdes.
HHU / Alexander Kroll
Las enzimas son importantes biocatalizadores en todas las células vivas. Suelen ser proteínas de gran tamaño que se unen a moléculas más pequeñas, los llamados sustratos, y las convierten en otras moléculas, los "productos". Sin enzimas, la reacción que convierte los sustratos en productos no podría llevarse a cabo, o sólo podría hacerlo a una velocidad muy baja. La mayoría de los organismos poseen miles de enzimas diferentes. Las enzimas tienen muchas aplicaciones en una amplia gama de procesos biotecnológicos y en la vida cotidiana, desde la fermentación de la masa del pan hasta los detergentes.
La velocidad máxima a la que una enzima específica puede convertir sus sustratos en productos viene determinada por el llamado número de recambio kcat. Es un parámetro importante para la investigación cuantitativa de las actividades enzimáticas y desempeña un papel clave en la comprensión del metabolismo celular.
Sin embargo, determinar el número de recambio kcat en experimentos lleva mucho tiempo y es caro, por lo que no se conoce para la gran mayoría de las reacciones. El grupo de investigación de Biología Celular Computacional de la HHU, dirigido por el Dr. Martin Lercher, ha desarrollado una nueva herramienta llamada TurNuP para predecir los números de recambio kcat de las enzimas mediante métodos de IA.
Para entrenar un modelo de predicción de kcat, la información sobre las enzimas y las reacciones catalizadas se convirtió en vectores numéricos mediante modelos de aprendizaje profundo. Estos vectores numéricos sirvieron de entrada para un modelo de aprendizaje automático -el llamado modelo de refuerzo de gradiente- que predice los números de recambio kcat.
Autor principal Alexander Kroll: "TurNuP supera a los modelos anteriores e incluso se puede utilizar con éxito para enzimas que solo tienen una baja similitud con las del conjunto de datos de entrenamiento." Los modelos anteriores no han sido capaces de hacer predicciones significativas a menos que al menos el 40% de la secuencia de la enzima sea idéntica a al menos una enzima del conjunto de entrenamiento. En cambio, TurNuP ya puede hacer predicciones significativas para enzimas con una identidad de secuencia máxima de entre el 0 y el 40%.
El profesor Lercher añade: "En nuestro estudio, demostramos que las predicciones realizadas por TurNuP pueden utilizarse para predecir las concentraciones de enzimas en células vivas con mucha más precisión que hasta ahora".
Para que el modelo de predicción sea fácilmente accesible al mayor número posible de usuarios, el equipo de la HHU ha desarrollado un servidor web de fácil manejo, que otros investigadores pueden utilizar para predecir los números de recambio kcat de las enzimas.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.