Escalas moleculares en membranas biológicas: Seguimiento de partículas sensibles a la masa
Nuevo método para determinar la localización y los cambios de tamaño de las proteínas no marcadas en las membranas a través de su masa
Una gran parte de los procesos biológicamente relevantes tienen lugar en las membranas. Estudiar la dinámica de estos procesos en tiempo real y sin perturbar el sistema biológico sigue siendo un gran reto metodológico. Un equipo dirigido por Petra Schwille, directora del MPI de Bioquímica (MPIB), y Nikolas Hundt, de la Ludwig-Maximilians-Universität München (LMU), ha desarrollado ahora un nuevo método para este fin: el seguimiento de partículas sensibles a la masa (MSPT). Con el MSPT se pueden determinar los movimientos y las reacciones de las proteínas individuales no marcadas en las membranas biológicas únicamente por su masa. El método se ha publicado en Nature Methods.
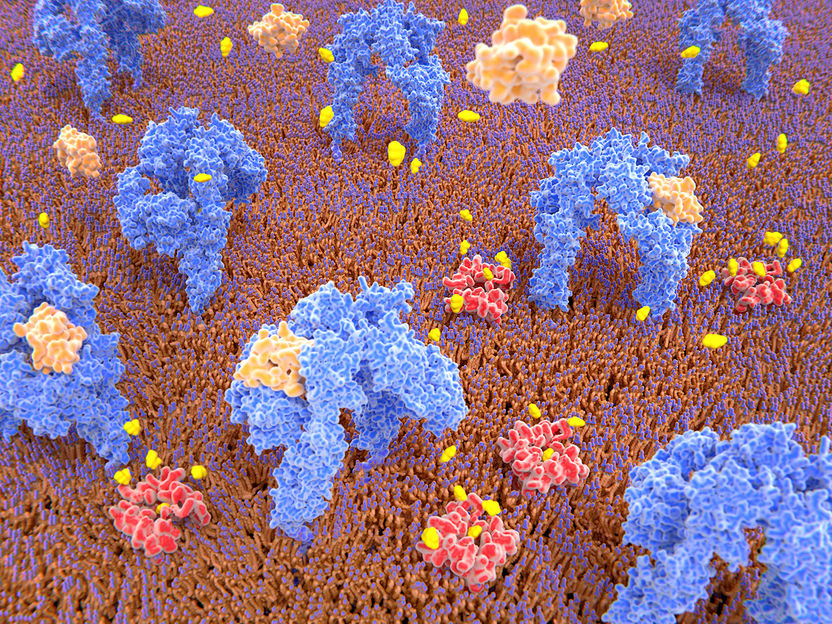
El nuevo método Mass-Sensitive Particle Tracking (MSPT) permite analizar la masa molecular, el movimiento y la interacción de las proteínas en las membranas biológicas.
Juan Gärtner, AdobeStock
Los procesos celulares en las membranas suelen ser rápidos y de corta duración. Las moléculas se ensamblan brevemente, se separan de nuevo, interactúan con diferentes socios y se mueven a lo largo o a través de la membrana. Por ello, es importante no sólo estudiar instantáneas estáticas de estos procesos, sino también comprender su dinámica. Pero, ¿cómo se puede conseguir esto de forma metódica? Petra Schwille, del MPIB, y Nikolas Hundt, de la LMU, junto con su equipo, han desarrollado el método Mass-Sensitive Particle Tracking - MSPT, que permite analizar las proteínas durante los procesos dinámicos en las membranas.
Análisis de procesos dinámicos en membranas biológicas
El punto de partida para los biofísicos fueron los recientes avances en la fotometría de masas, que ya podían utilizarse para determinar la masa molecular de moléculas no marcadas en solución. La novedad de la MSPT es que ahora se puede seguir la dinámica de las proteínas asociadas a la membrana en su entorno biológicamente plausible. En este proceso, las proteínas individuales se identifican por su masa molecular sin necesidad de etiquetarlas. Frederik Steiert, uno de los primeros autores de la publicación, afirma: "Ahora podemos seguir directamente en las membranas biológicas qué masa tienen las proteínas individuales, cómo se mueven y cómo interactúan. Esto nos permite estudiar la dinámica de los sistemas biológicos con mayor detalle". Analizar los procesos dinámicos es especialmente importante en biología, ya que muchos procesos en la membrana son transitorios.
Determinación de la masa mediante dispersión de la luz
¿En qué principios se basa el nuevo método? Cuando la luz incide en una partícula, la luz se dispersa. La intensidad de la luz dispersa depende de la masa de la partícula. Con un microscopio se graban vídeos en los que se hacen visibles directamente las proteínas individuales en las membranas. Con la ayuda de un software de análisis, estas proteínas pueden ser rastreadas y su señal de dispersión, y por lo tanto su masa, puede ser determinada. Esto es posible actualmente para las proteínas con un peso molecular de al menos 50 kDa, es decir, para una gran parte de todas las proteínas conocidas. Otra ventaja del nuevo método MSPT es que no es necesario etiquetar las proteínas. El etiquetado puede lograrse, por ejemplo, uniendo etiquetas fluorescentes a las moléculas. Sin embargo, el etiquetado plantea el riesgo de que las proteínas puedan ver afectada su función o de que las etiquetas fluorescentes se decoloren durante el experimento. En cambio, con el uso de MSPT se evitan los problemas metodológicos que pueden surgir del etiquetado.
Sistema de proteínas MinDE
Para demostrar el potencial del método en cuestiones biológicas, los biofísicos utilizaron un sistema establecido del laboratorio de Schwille: el sistema de proteínas MinDE de la bacteria Escherichia coli (E. coli). Las proteínas MinD y MinE intervienen en la división celular de E. coli. Tamara Heermann, otra de las primeras autoras, afirma: "El método nos permite caracterizar propiedades de sistemas dinámicos que antes no se podían medir. Esto nos permitió no sólo verificar los hallazgos establecidos sobre el sistema Min, sino también obtener nuevos conocimientos". Mediante el uso de MSPT, el equipo pudo demostrar que los complejos de las proteínas MinD son más grandes de lo que se pensaba inicialmente. Además, los experimentos proporcionan los primeros conocimientos de que MinE puede actuar como una pieza de conexión para las proteínas MinD y que, por tanto, puede iniciar la liberación de la membrana de los complejos más grandes.
Tal y como se recoge en el nuevo artículo de Nature Methods, el MSPT proporciona valiosos conocimientos para dilucidar los procesos dinámicos en las membranas biológicas. Sin embargo, los investigadores están trabajando continuamente para mejorar el método aún más. En el futuro, el método también debería ser aplicable a las proteínas integrales de membrana y debería permitir la detección de proteínas incluso más pequeñas.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.