Una herramienta identifica virus específicos para combatir bacterias peligrosas
Un equipo de investigadores acorta la búsqueda de atacantes capaces de acabar con patógenos multirresistentes, con el objetivo de tratar infecciones sin antibióticos
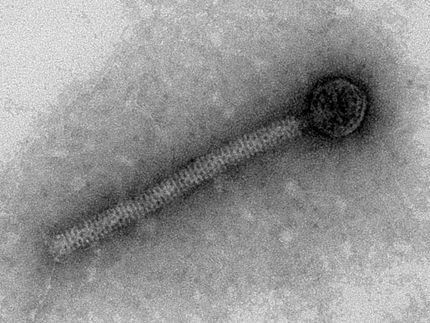
Una herramienta de laboratorio recientemente desarrollada puede ayudar, en cuestión de horas, a identificar virus específicos que pueden utilizarse para destruir variantes de la peligrosa bacteria patógena Staphylococcus aureus. Los virus de las bacterias, conocidos como bacteriófagos, ofrecen un enfoque alternativo a los antibióticos en el tratamiento de patógenos multirresistentes. La nueva herramienta podría suponer una importante contribución a las futuras terapias con fagos, que aún no se utilizan de forma estándar en Alemania. Ha sido desarrollada por un equipo de investigación dirigido por el profesor Andreas Peschel, del Cluster de Excelencia "Controlling Microbes to Fight Infections" (CMFI) de la Universidad de Tubinga, y publicada en la revista Cell Reports.
Las bacterias multirresistentes suponen una amenaza creciente para la salud humana en todo el mundo. Entre ellas se encuentra un patógeno frecuente en los hospitales, el Staphylococcus aureus, que puede causar inflamaciones graves e incluso sepsis. En su forma multirresistente, el SARM sólo puede tratarse con gran dificultad. El director del estudio, Andreas Peschel, afirma: "Los antibióticos son nuestra arma más importante contra las infecciones, pero cada vez con más frecuencia vemos que o son ineficaces o causan demasiados efectos secundarios". Y continúa: "Los fagos, por el contrario, son muy específicos y pueden atacar y eliminar patógenos individuales sin alterar el resto del microbioma beneficioso para la salud de los pacientes."
El enemigo de un enemigo se convierte en amigo
La terapia con fagos aprovecha el hecho de que los bacteriófagos infectan específicamente determinadas variantes de bacterias, se multiplican en su interior y acaban destruyéndolas. Durante este proceso, se liberan nuevos bacteriófagos que pueden combatir otras bacterias. El primer autor del estudio, Janes Krusche, del Clúster de Excelencia CMFI, explica: "Sin embargo, debido a su especificidad, ya no pueden multiplicarse una vez eliminadas todas las bacterias patógenas". Uno de los retos de esta terapia es elegir los bacteriófagos adecuados, añade. Krusche es el principal desarrollador de la nueva herramienta de identificación de fagos (Phage Aureus RBP Identification System; PhARIS). PhARIS identifica proteínas específicas de unión a receptores en el material genético de los fagos para determinar su capacidad de infectar determinadas variantes de Staphylococcus aure-us.
Peschel y Krusche creen que esta herramienta tiene un gran potencial para mejorar las terapias con fagos en el tratamiento de heridas infectadas e infecciones asociadas a implantes. El equipo de investigación tiene previsto seguir desarrollando el sistema para otros patógenos. El objetivo es convertir PhARIS en una herramienta de laboratorio estándar para identificar fagos de forma rápida y eficaz como tratamiento alternativo a los antibióticos para muchas infecciones bacterianas diferentes.
"En todo el mundo, las infecciones causadas por gérmenes multirresistentes son difíciles de tratar y constituyen uno de los mayores retos médicos de nuestro tiempo. Los nuevos resultados de la investigación del Clúster de Excelencia CMFI de la Universidad de Tubinga muestran de manera impresionante lo esencial que es la investigación básica en este campo. En PhARIS, nuestros investigadores han desarrollado una herramienta innovadora que acelera la selección de los fagos adecuados para futuras terapias. El resultado es un beneficio directo para los pacientes. Estos extraordinarios resultados de investigación subrayan una vez más la posición preeminente a nivel internacional de la microbiología y la biología de las infecciones en la Universidad de Tubinga", declaró la Profesora Dra. Dr. h.c. (Dōshisha) Karla Pollmann, Presidenta y Vicerrectora de la Universidad de Tubinga.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.
Publicación original
Janes Krusche, Christian Beck, Esther Lehmann, David Gerlach, Ellen Daiber, Christoph Mayer, Jennifer Müller, Hadil Onallah, Silvia Würstle, Christiane Wolz, Andreas Peschel; "Characterization and host range prediction of Staphylococcus aureus phages through receptor-binding protein analysis"; Cell Reports, Volume 44