Microscopía de fluorescencia de resolución Ångström
Esta innovación está a punto de suponer un cambio de paradigma en nuestra forma de estudiar los sistemas biológicos con un nivel de detalle sin precedentes hasta la fecha.
El grupo de investigación de Ralf Jungmann, del Instituto Max Planck de Bioquímica (MPIB) y la Universidad Ludwig Maximilian (LMU) de Múnich, ha logrado un gran avance en la microscopía de fluorescencia. El equipo ha desarrollado RESI (Resolution Enhancement by Sequential Imaging), una técnica revolucionaria que mejora la resolución de la microscopía de fluorescencia hasta la escala de Ångström. Esta innovación está a punto de marcar el comienzo de un cambio de paradigma en nuestra forma de estudiar los sistemas biológicos con un nivel de detalle sin precedentes hasta la fecha.
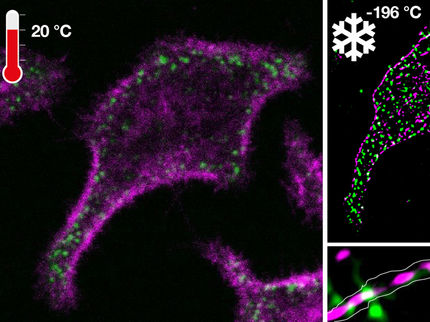
RESI permite la microscopía a través de escalas de longitud con una resolución de Ångström: Desde células enteras, pasando por proteínas individuales, hasta la distancia entre dos bases adyacentes en el ADN.
Illustration: Max Iglesias, MPI für Biochemie
Las células, unidades fundamentales de la vida, contienen una plétora de estructuras, procesos y mecanismos intrincados que sostienen y perpetúan los sistemas vivos. Muchos componentes celulares básicos, como el ADN, el ARN, las proteínas y los lípidos, tienen un tamaño de apenas unos nanómetros. Esto los hace sustancialmente más pequeños que el límite de resolución de la microscopía óptica tradicional. Por ello, a menudo se desconoce la composición y disposición exactas de estas moléculas y estructuras, lo que provoca una falta de comprensión mecanicista de aspectos fundamentales de la biología.
En los últimos años, las técnicas de superresolución han dado pasos de gigante para resolver muchas estructuras subcelulares por debajo del límite clásico de difracción de la luz. La microscopía de localización de moléculas individuales (Single Molecule Localization Microscopy, SMLM) es un método de superresolución que permite resolver estructuras del orden de diez nanómetros de tamaño separando temporalmente su emisión de fluorescencia individual. Como los objetivos individuales se iluminan estocásticamente (parpadean) en un campo de visión que, de otro modo, estaría oscuro, su localización puede determinarse con una precisión inferior a la difracción. DNA-PAINT, inventada por el grupo de Jungmann, es una técnica de SMLM que utiliza la hibridación transitoria de cadenas de ADN marcadas con colorantes "generadoras de imágenes" con sus complementos unidos al objetivo para lograr el parpadeo necesario para la superresolución. Sin embargo, hasta la fecha, ni siquiera DNA-PAINT ha sido capaz de resolver las estructuras celulares más pequeñas.
En el estudio actual, dirigido por los coautores Susanne Reinhardt, Luciano Masullo, Isabelle Baudrexel y Philipp Steen junto con Jungmann, el equipo introduce un novedoso enfoque en microscopía de superresolución que permite una resolución espacial fundamentalmente "ilimitada". La nueva técnica, denominada "Resolution Enhancement by Sequential Imaging" (RESI), aprovecha la capacidad de DNA-PAINT para codificar la identidad de los objetivos mediante secuencias de ADN únicas. Al etiquetar objetivos adyacentes, demasiado cercanos entre sí para ser resueltos incluso mediante microscopía de superresolución, con cadenas de ADN diferentes, se introduce en la muestra un grado adicional de diferenciación (un código de barras). Al visualizar secuencialmente primero una secuencia y luego la otra (y, por tanto, la diana), es posible separarlas sin ambigüedad. Y lo que es más importante, como se obtienen secuencialmente, los objetivos pueden estar arbitrariamente cerca el uno del otro, algo que ninguna otra técnica puede resolver. Además, RESI no requiere instrumentación especializada; de hecho, puede aplicarse con cualquier microscopio de fluorescencia estándar, lo que la hace fácilmente accesible para casi todos los investigadores.
Para demostrar el salto de resolución de RESI, el equipo se propuso el reto de resolver una de las distancias espaciales más pequeñas en un sistema biológico: La separación entre bases individuales a lo largo de una doble hélice de ADN, separadas menos de un nanómetro (la milmillonésima parte de un metro). Mediante el diseño de una nanoestructura de origami de ADN que presenta secuencias de ADN monocatenario que sobresalen de una doble hélice a una distancia de un par de bases y la posterior obtención de imágenes secuenciales de estos monocatenarios, el equipo de investigación resolvió una distancia de 0,85 nm (u 8,5 Ångström) entre bases adyacentes, una hazaña hasta entonces inimaginable. Los investigadores lograron estas mediciones con una precisión de 1 Ångström, o una diezmilmillonésima parte de un metro, lo que subraya las capacidades sin precedentes del método RESI.
Y lo que es más importante, la técnica es universal y no se limita a aplicaciones en nanoestructuras de ADN. Para ello, el equipo investigó el modo de acción molecular del rituximab, un anticuerpo monoclonal anti-CD20 que se aprobó por primera vez en 1997 para el tratamiento del cáncer de sangre CD20-positivo. Sin embargo, la investigación de los efectos de tales moléculas farmacológicas en los patrones de receptores moleculares ha estado más allá de las capacidades de resolución espacial de las técnicas tradicionales de microscopía. Comprender si estos patrones cambian en la salud y la enfermedad, así como en el tratamiento, y cómo lo hacen, no sólo es importante para la investigación mecanicista básica, sino también para el diseño de nuevas terapias dirigidas contra la enfermedad. Gracias a la RESI, Jungmann y su equipo pudieron revelar la disposición natural de los receptores CD20 como dímeros en células no tratadas y cómo el CD20 se reorganizaba en cadenas de dímeros tras el tratamiento farmacológico. Los datos obtenidos a nivel de una sola proteína ayudan ahora a esclarecer el modo de acción molecular del rituximab.
Como la RESI se realiza en células enteras e intactas, la técnica cierra la brecha entre las técnicas puramente estructurales, como la cristalografía de rayos X o la microscopía electrónica criogénica, y los métodos tradicionales de obtención de imágenes de células enteras de menor resolución. Jungmann y su equipo están convencidos de que "esta técnica sin precedentes es un verdadero revulsivo no sólo para la superresolución, sino para la investigación biológica en su conjunto".
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.