Un novedoso software basado en inteligencia artificial permite obtener imágenes rápidas y fiables de proteínas en células
Los investigadores programaron una herramienta que reconoce y selecciona con precisión las proteínas en criotomografía electrónica, sustituyendo a la molesta selección manual
La criotomografía electrónica (crioET) se perfila como una potente técnica para obtener imágenes tridimensionales detalladas de entornos celulares y biomoléculas encerradas. Sin embargo, uno de los retos de la metodología es la identificación de moléculas proteicas en las imágenes para su posterior procesamiento. Un equipo de investigación en torno a Stefan Raunser, Director del MPI de Fisiología Molecular de Dortmund, dirigido por Thorsten Wagner, desarrolló un software para seleccionar proteínas en volúmenes celulares abarrotados. La nueva herramienta de código abierto, denominada TomoTwin, se basa en el aprendizaje métrico profundo y permite a los científicos localizar varias proteínas con gran precisión y rendimiento sin necesidad de crear o reentrenar manualmente la red cada vez.

Imagen simbólica
Computer-generated image
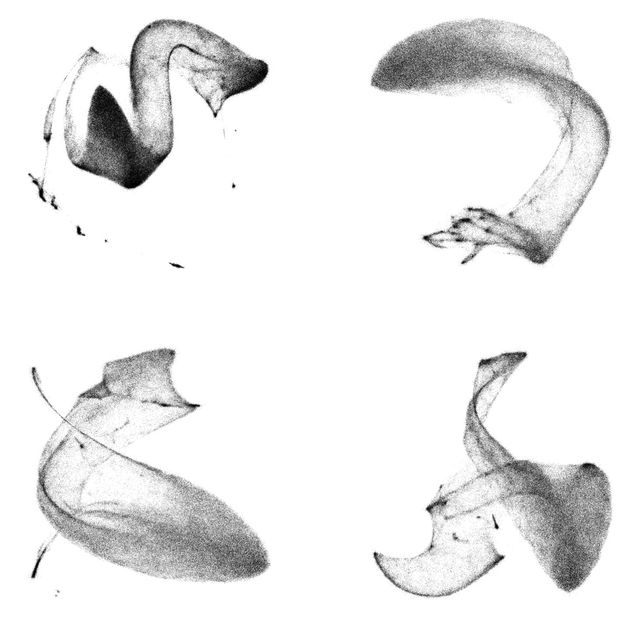
Mapa de procesamiento de TomoTwin para un tomograma aplanado a 2D. Las partículas de diferentes macromoléculas se ordenan en el mapa según su estructura, lo que permite a los usuarios identificar y localizar diferentes macromoléculas dentro de las células.
MPI of Molecular Physiology

Gavin Rice (izquierda) y Thorsten Wagner (derecha), primeros autores.
MPI of Molecular Physiology


Cuantas más, mejor
"TomoTwin allana el camino para la identificación y localización automatizada de proteínas directamente en su entorno celular, ampliando el potencial de la crio-ET", afirma Gavin Rice, co-primer autor de la publicación. La crioET tiene el potencial de descifrar cómo funcionan las biomoléculas dentro de una célula y, con ello, desvelar la base de la vida y el origen de las enfermedades".
En un experimento de crioET, los científicos utilizan un microscopio electrónico de transmisión para obtener imágenes tridimensionales, llamadas tomogramas, del volumen celular que contiene biomoléculas complejas. Para obtener una imagen más detallada de cada proteína diferente, promedian tantas copias de ellas como sea posible - de forma similar a los fotógrafos que capturan la misma foto a distintas exposiciones para combinarlas después en una imagen perfectamente expuesta. Lo más importante es identificar y localizar correctamente las distintas proteínas en la imagen antes de promediarlas. "Los científicos pueden obtener cientos de tomogramas al día, pero carecíamos de herramientas para identificar plenamente las moléculas que contienen", afirma Rice.
Selección manual
Hasta ahora, los investigadores utilizaban algoritmos basados en plantillas de estructuras moleculares ya conocidas para buscar coincidencias en los tomogramas, pero suelen ser propensos a errores. Identificar las moléculas a mano es otra opción que garantiza una selección de alta calidad, pero lleva de días a semanas por conjunto de datos.
Otra posibilidad sería utilizar una forma de aprendizaje automático supervisado. Estas herramientas pueden ser muy precisas, pero actualmente carecen de utilidad, ya que requieren etiquetar manualmente miles de ejemplos para entrenar el software para cada nueva proteína, una tarea casi imposible para pequeñas moléculas biológicas en un entorno celular abarrotado.
TomoTwin
El nuevo software TomoTwin supera muchos de estos obstáculos: Aprende a elegir las moléculas de forma similar dentro de un tomograma y las mapea en un espacio geométrico: el sistema es recompensado por colocar proteínas similares cerca unas de otras y penalizado en caso contrario. En el nuevo mapa, los investigadores pueden aislar e identificar con precisión las distintas proteínas y utilizarlo para localizarlas dentro de la célula. "Una de las ventajas de TomoTwin es que proporcionamos un modelo de selección preentrenado", afirma Rice. Al eliminar el paso de entrenamiento, el software puede ejecutarse incluso en ordenadores locales: mientras que el procesamiento de un tomograma suele requerir entre 60 y 90 minutos, el tiempo de ejecución en el superordenador MPI Raven se reduce a 15 minutos por tomograma.
TomoTwin permite a los investigadores seleccionar docenas de tomogramas en el tiempo que se tarda en seleccionar manualmente uno solo, con lo que aumenta el caudal de datos y la tasa de promediado para obtener una imagen mejor. Actualmente, el software puede localizar proteínas globulares o complejos proteicos de más de 150 kilodaltons en las células; en el futuro, el grupo de Raunser pretende incluir proteínas de membrana, proteínas filamentosas y proteínas de tamaños más pequeños.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.