Descubiertos por cribado virtual unos compuestos naturales que inhiben la proteasa principal del virus SARS-CoV-2
La lucha contra la pandemia de COVID-19
La lucha contra la pandemia de Covid-19 es un desafío científico a nivel internacional para encontrar estrategias con las que combatir la infección viral y prevenir sus consecuencias. Además de las vacunas y los medicamentos aprobados por las autoridades sanitarias, es vital encontrar nuevos tratamientos para luchar contra la infección vírica una vez se ha producido.
Este nuevo avance científico se basa en el uso del cribado virtual (in silico), una técnica de análisis computacional para seleccionar moléculas candidatas a futuros fármacos en una biblioteca química.
Universidad de Barcelona
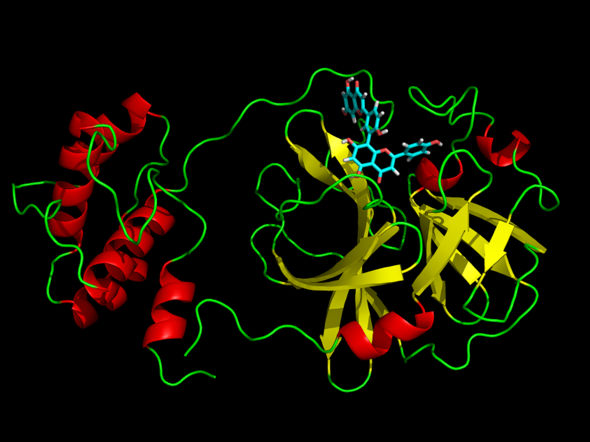
Un estudio publicado en Journal of Chemical Information and Modeling ha identificado una serie de productos naturales que son capaces de inhibir la Mpro, la principal proteasa del virus SARS-CoV-2. La Mpro es una proteína no estructural que tiene un papel esencial en la replicación y la transcripción de este virus, y se considera una potencial diana terapéutica, ya que, si se pudiera inhibir, podría evitar la progresión del virus.
El trabajo está dirigido por el catedrático Jaime Rubio Martínez, de la Facultad de Química y del Instituto de Química Teórica y Computacional (IQTC) de la Universidad de Barcelona, y puede abrir nuevas perspectivas para diseñar nuevas estrategias terapéuticas en la lucha contra la COVID -19. Este nuevo avance científico se basa en el uso del cribado virtual (in silico), una técnica de análisis computacional de moléculas almacenadas en bases de datos para identificar y seleccionar compuestos candidatos de potencial interés farmacológico.
Cribado virtual: en busca de futuros fármacos
Hoy en día, el abordaje clínico de la COVID-19 consiste en el tratamiento sintomático mediante agentes antiinflamatorios (por ejemplo, la dexametasona o los inhibidores de citoquinas) combinados con antibióticos para tratar infecciones secundarias. Más recientemente, también se ha aprobado el antiviral ritonavir, pero todavía hay necesidad de nuevos medicamentos. En el contexto de la salud global, es urgente desarrollar terapias antivirales específicas contra el SARS-CoV-2, lo que ha llevado a poner en marcha diversas estrategias para identificar compuestos bioactivos que puedan utilizarse como agentes terapéuticos (incluidos los fármacos disponibles y los productos naturales). En este contexto, el cribado virtual es un procedimiento fiable, rápido y eficiente para descubrir compuestos bioactivos de grandes colecciones químicas contra un objetivo molecular específico o diana.
En el marco de este trabajo, el equipo ha llevado a cabo un cribado virtual de la base de datos Selleck de productos naturales —una biblioteca química con cerca de 2.000 compuestos— para un conjunto de diversas configuraciones de la proteasa Mpro del virus SARS-CoV-2, caracterizadas a partir de un estudio de dinámica molecular.
Como resultado, el trabajo presenta en primer lugar la caracterización del perfil dinámico de la proteasa Mpro en la forma apo mediante simulaciones convencionales (cMD) y de dinámica molecular acelerada gaussiana (GaMD), y define una serie de estructuras representativas. Estas estructuras se utilizaron posteriormente para llevar a cabo el ensamblaje molecular, un método bioinformático que permite predecir y calcular con técnicas computacionales la posición más favorable de interacción entre un ligando y su proteína diana. A continuación, se desarrolló un proceso iterativo con el que se incrementó la longitud de las cMD de los complejos proteína-ligando y se calculó la energía libre de unión para seleccionar los candidatos más prometedores.
Teniendo en cuenta los resultados, se seleccionaron once compuestos y se probaron in vitro por su capacidad de inhibir la proteasa Mpro. Por último, del conjunto final se identificaron cinco candidatos antivirales SARS-CoV-2, inhibidores de la proteasa Mpro, a partir de la base de datos de productos naturales.




















































