les "dictionnaires" rendent accessibles les données basées sur la fluorescence pour les modèles de structure intégrative et leur dynamique
structures 3D des biomolécules
Une équipe de recherche allemande et américaine dirigée par l'université Heinrich Heine de Düsseldorf (HHU) a mis au point une description de données qui peut fournir des résultats de mesures de fluorescence pour la modélisation structurelle et dynamique de grandes biomolécules. Les auteurs expliquent dans la revue scientifique Nature Methods que, pour la première fois, d'autres chercheurs peuvent accéder à des modèles structurels intégratifs basés sur la fluorescence et à leur dynamique par l'intermédiaire de bases de données. Cela permet d'obtenir des données d'entraînement basées sur des expériences pour la prochaine génération d'outils d'intelligence artificielle destinés à la modélisation dynamique de la structure.
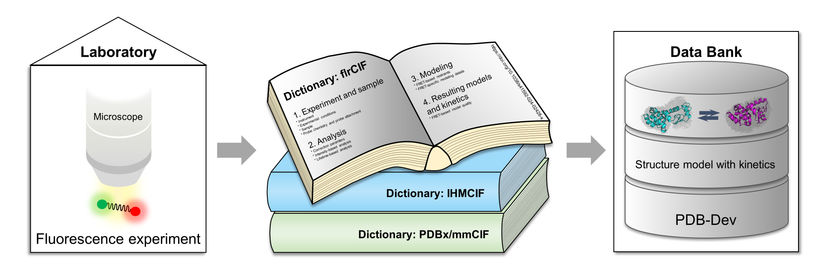
Les données issues des expériences de fluorescence sont traitées à l'aide de "dictionnaires" et mises à disposition dans une base de données avec des modèles de structure intégrative.
HHU / Christian Hanke
Les protéines et les acides nucléiques sont les principaux éléments constitutifs de la vie dans tous les organismes. Ces biomolécules sont constituées de nombreux éléments individuels, tels que les acides aminés dans le cas des protéines. Lorsque les différents éléments sont assemblés dans les cellules, les biomolécules forment des structures tridimensionnelles complexes. Leur forme spécifique est déterminée par la configuration et les forces entre les éléments constitutifs. Cependant, pour comprendre la fonction des biomolécules, il est important de prendre en compte non seulement leur structure tridimensionnelle, mais aussi le nombre d'états structurels différents et la dynamique des échanges entre eux.
Pendant longtemps, la détermination de la structure tridimensionnelle des biomolécules à l'aide des méthodes biophysiques classiques a été très complexe et a pris beaucoup de temps. Afin de simplifier et de systématiser ce travail étape par étape, toutes ces structures tridimensionnelles ont été rassemblées dans la "Protein Data Bank" (PDB) depuis 1971. Ces plus de 220 000 structures sont utilisées par des outils d'intelligence artificielle tels que "AlphaFold" - pour le développement duquel le prix Nobel de chimie a été décerné cette année - comme données d'entraînement pour les réseaux neuronaux. Les systèmes d'IA font déjà de bonnes prédictions sur les structures biomoléculaires. Cependant, ces outils sont actuellement incapables de prédire la dynamique en raison d'un manque de données expérimentales.
Il est donc important d'utiliser des méthodes expérimentales puissantes, telles que la spectroscopie de fluorescence, qui fournissent des informations combinées sur la dynamique et la structure de biomolécules complexes. Dans les expériences de fluorescence, des parties intéressantes spécifiques des biomolécules sont marquées avec de petites molécules de colorant qui s'illuminent lorsqu'elles sont excitées de l'extérieur et révèlent ainsi leur position. Les approches de modélisation intégrative combinent ces données expérimentales avec des méthodes informatiques afin d'obtenir une meilleure résolution structurelle et de prendre en compte la dynamique.
Le Dr Christian Hanke, postdoc à l'Institut de chimie physique moléculaire de la HHU et premier auteur de l'article publié dans Nature Methods, souligne : "Les expériences de fluorescence fournissent des informations détaillées, ce qui en fait une excellente source de données pour la modélisation intégrative. Toutefois, pour exploiter pleinement cette mine d'informations, il faut la rendre accessible et utilisable par une large communauté scientifique."
Dans la publication, l'équipe de recherche de l'HHU, de l'université d'État Rutgers du New Jersey et de l'université de Californie à San Francisco présente une description normalisée des données sous la forme de trois "dictionnaires" liés entre eux et organisés dans une bibliothèque commune. Claus Seidel de la HHU, l'un des deux auteurs correspondants : "Ce principe d'organisation avec des dictionnaires combinés permet pour la première fois aux chercheurs de stocker des modèles structurels intégratifs basés sur des données de fluorescence ainsi que des informations cinétiques. En même temps, les définitions générales peuvent également être utilisées par d'autres méthodes pour documenter les propriétés dynamiques des biomolécules parallèlement à leur structure dans la base de données."
Cette approche est nécessaire pour relier les modèles structurels statiques au paysage énergétique sous-jacent, c'est-à-dire aux différences d'énergie entre les différents arrangements tridimensionnels des éléments constitutifs de la biomolécule. Le professeur Seidel : "Ces informations permettent de développer et de former la prochaine génération de programmes basés sur l'IA pour prédire les structures biomoléculaires dynamiques. Ici, les données obtenues à partir d'expériences de fluorescence sur les dynamiques fonctionnellement pertinentes peuvent apporter une contribution très importante."
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.