Microscopie ONE : de la molécule à la structure 3D avec la microscopie conventionnelle
La méthode, simple et rentable, a été élaborée à partir de quelques astuces simples mais efficaces
Annonces
Des chercheurs du Centre médical universitaire de Göttingen (UMG) ont mis au point une nouvelle méthode qui permet pour la première fois d'imager la forme tridimensionnelle des protéines à l'aide d'un microscope classique. Combinée à l'intelligence artificielle, la microscopie ONE (One-step Nanoscale Expansion) permet de détecter les changements structurels des protéines endommagées ou toxiques dans des échantillons humains. Des maladies telles que la maladie de Parkinson, qui sont basées sur un mauvais repliement des protéines, pourraient ainsi être détectées et traitées à un stade précoce. La microscopie ONE a été désignée comme l'une des "sept technologies à surveiller en 2024" par la revue Nature et a été récemment publiée dans la revue Nature Biotechnology.

Les inventeurs (de gauche à droite), le chef de groupe Dr. Ali H. Shaib et le professeur Silvio O. Rizzoli, directeur du département de physiologie neurologique et sensorielle du centre médical universitaire de Göttingen (UMG).
A. Shaib
L'imagerie de fluorescence est l'un des outils les plus polyvalents et les plus utilisés en biologie pour observer les processus biologiques dans les cellules vivantes. Malgré les progrès technologiques et l'amélioration de la résolution, la visualisation de molécules uniques et de l'organisation de complexes moléculaires à l'aide de la microscopie à fluorescence reste un défi. Jusqu'à présent, cela n'était possible qu'en utilisant des méthodes coûteuses de biologie structurale telles que la microscopie électronique (ME) et, en particulier, la cryomicroscopie électronique, dans laquelle les échantillons sont imagés avec un faisceau d'électrons très puissant à une température extrêmement basse.
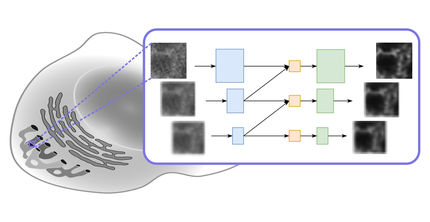
Une équipe de recherche dirigée par le professeur Silvio O. Rizzoli, directeur du département de physiologie neurologique et sensorielle du centre médical universitaire de Göttingen (UMG), porte-parole du Centre d'imagerie biostructurale de la neurodégénérescence (BIN) et membre du pôle d'excellence "Bioimagerie multi-échelle : des machines moléculaires aux réseaux de cellules excitables" (MBExC), et par le Dr. Ali Shaib, chef de groupe au Département de physiologie neurologique et sensorielle de l'UMG, ont mis au point une méthode utilisant quelques astuces simples mais efficaces pour visualiser des molécules individuelles en détail à l'aide de la microscopie optique conventionnelle. Au lieu d'utiliser des microscopes coûteux à haute résolution pour améliorer la résolution, ils ont mis au point la microscopie ONE (One-step Nanoscale Expansion). Dans cette méthode, le volume de l'échantillon est augmenté en liant les cellules et les structures qu'elles contiennent à un gel absorbant l'eau qui pénètre dans les cellules. En absorbant l'eau, le gel augmente jusqu'à 15 fois son volume. Les molécules de l'échantillon s'écartent alors uniformément les unes des autres et deviennent également plus grosses, de sorte qu'elles peuvent être visualisées au microscope optique après avoir été marquées de manière spécifique par des molécules fluorescentes. Grâce à une méthode basée sur l'intelligence artificielle pour évaluer les changements de fluorescence, les scientifiques ont réussi pour la première fois à faire ce qui n'était auparavant possible qu'avec la cryo-microscopie électronique à haute résolution et la technologie des rayons X : "Nous sommes désormais en mesure de reconstruire des structures protéiques en 3D à partir d'images de fluorescence bidimensionnelles", explique le professeur Rizzoli. Cela offre une opportunité sans précédent de visualiser directement les détails structurels fins des protéines individuelles ainsi que des complexes multiprotéiques dans les cellules ou de manière isolée. Les changements dans la structure spatiale des protéines peuvent également être facilement détectés avec la microscopie ONE. Dans le cadre d'une collaboration avec des collègues de Göttingen et de Kassel, des agrégats de protéines moléculaires, typiques de la maladie de Parkinson, ont été imagés et classés dans des échantillons de liquide céphalo-rachidien de patients. Ces résultats sont prometteurs pour améliorer la détection précoce de la maladie de Parkinson, qui touche des millions de personnes dans le monde.
La microscopie ONE est une méthode simple et rentable qui peut être réalisée dans n'importe quel laboratoire avec un microscope conventionnel et qui permet d'atteindre un niveau de résolution d'environ un nanomètre. C'est environ 100 000 fois plus petit que le diamètre d'un cheveu humain. Les auteurs fournissent le logiciel nécessaire sous la forme d'un logiciel libre. La nouvelle méthode a été publiée dans la revue Nature Biotechnology. En raison de son potentiel élevé, Nature a inclus la microscopie ONE dans sa liste des "sept technologies à surveiller en 2024".
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.
Publication originale
Ali H. Shaib, Abed Alrahman Chouaib, Rajdeep Chowdhury, Jonas Altendorf, Daniel Mihaylov, Chi Zhang, Donatus Krah, Vanessa Imani, Russell K. W. Spencer, Svilen Veselinov Georgiev, Nikolaos Mougios, Mehar Monga, Sofiia Reshetniak, Tiago Mimoso, ... Julia Preobraschenski, Ute Becherer, Tobias Moser, Edward S. Boyden, A. Radu Aricescu, Markus Sauer, Felipe Opazo, Silvio O. Rizzoli; "One-step nanoscale expansion microscopy reveals individual protein shapes"; Nature Biotechnology, 2024-10-9