Suivre et tracer les membres du microbiome végétal à l'aide de codes-barres ADN
Une équipe de recherche dirigée par Paul Schulze-Lefert de l'Institut Max Planck de recherche sur l'amélioration des plantes à Cologne, en Allemagne, a mis au point un ensemble d'outils modulaires permettant de suivre les souches bactériennes qui colonisent les tissus végétaux en concurrence avec d'autres membres du microbiome. L'étude est maintenant publiée dans Nature Microbiology.
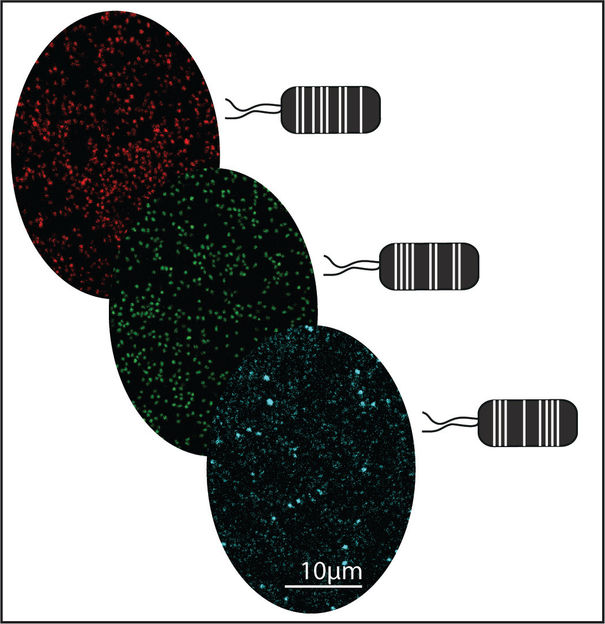
Schéma : bactéries Pseudomonas capeferrum dérivées de plantes marquées avec différents marqueurs fluorescents à l'aide de la boîte à outils MoBacTag.
Jana Ordon
L'énorme diversité de micro-organismes qu'une plante saine héberge en tant que communauté dans la nature est appelée microbiome végétal. Afin d'évaluer la composition du microbiome, invisible à l'œil nu, on détermine généralement la séquence d'ADN d'un gène marqueur microbien universel composé de segments de séquence variables et conservés. De cette manière, les différentes espèces microbiennes du microbiome peuvent être distinguées les unes des autres sur la base de séquences de marqueurs ADN polymorphes. Cependant, les activités bénéfiques des membres du microbiome pour la plante hôte, telles que la mobilisation des nutriments minéraux du sol pour l'absorption par les racines, ne sont souvent réalisées que par des souches microbiennes individuelles au sein d'une espèce bactérienne et dépendent de la présence de gènes microbiens spécialisés. Par conséquent, le profilage actuel du microbiote basé sur les séquences d'ADN est insuffisant pour capturer la véritable diversité génétique de la communauté microbienne sur l'hôte.
Pour surmonter cette limitation, les chercheurs de l'Institut Max Planck ont développé une boîte à outils modulaire qui est utilisée comme un code-barres d'ADN pour les souches bactériennes. Un code-barres ADN est d'abord inséré dans le chromosome d'une seule souche d'un membre du microbiome. Dans les analyses ultérieures des profils du microbiome sur les plantes, le code-barres ADN est considéré comme un gène marqueur microbien synthétique. Outre le code-barres de l'ADN chromosomique, des éléments génétiques pour les protéines fluorescentes ont également été incorporés. Ces dernières permettent d'utiliser des détecteurs de fluorescence très sensibles pour cartographier l'endroit où une souche bactérienne à code-barres colonise le tissu végétal en concurrence avec d'autres membres du microbiome.
Les chercheurs ont ensuite réalisé des expériences avec la bactérie Pseudomonas capeferrum, qui favorise la croissance des plantes et colonise les racines de la plante modèle Arabidopsis, ainsi qu'avec des variantes de la bactérie qui ne se distinguent de la souche de type sauvage que par l'absence de certains gènes. Les gènes Pseudomonas correspondants atténuent les réactions immunitaires de la plante hôte et renforcent ainsi la capacité de la bactérie à coloniser les racines de la plante, ce qui accroît son activité de promotion de la croissance de la plante. Les bactéries Pseudomonas marquées par des codes-barres ADN ont montré la capacité différentielle attendue à coloniser les racines d'Arabidopsis lorsque des plantes sans germe ont été inoculées avec des souches individuelles. Ce qui a été surprenant, cependant, c'est l'apparition de caractéristiques qualitativement nouvelles des bactéries Pseudomonas lorsque des combinaisons de la souche de type sauvage et de ses variantes ont été inoculées sur la plante hôte en même temps qu'un consortium de microbiome composé de différentes bactéries assemblées en laboratoire. En biologie, ce phénomène est également appelé propriété émergente ou propriété du système.
L'utilisation de codes-barres ADN a donc permis non seulement de confirmer les résultats précédents, mais aussi de révéler de nouvelles activités des gènes bactériens qui n'auraient pas pu être identifiées à l'aide de méthodes conventionnelles. La boîte à outils modulaire de codage à barres de l'ADN peut désormais être utilisée pour la recherche sur le microbiome afin d'étudier la contribution des gènes microbiens individuels dans le contexte des communautés microbiennes, non seulement dans les plantes, mais aussi dans les études sur le microbiome des animaux.
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.






















































