Microscopie : dépassement de la limite de résolution traditionnelle pour le co-tracking rapide de molécules
Des chercheurs ont mis au point une méthode innovante pour suivre simultanément les processus dynamiques rapides de plusieurs molécules à l'échelle moléculaire
Les processus à l'intérieur de notre corps sont caractérisés par l'interaction de diverses biomolécules telles que les protéines et l'ADN. Ces processus se déroulent souvent à une échelle de quelques nanomètres. Par conséquent, ils ne peuvent pas être observés avec la microscopie à fluorescence, qui a une limite de résolution d'environ 200 nanomètres en raison de la diffraction. Lorsque deux colorants marquant des positions de biomolécules sont plus proches que cette limite optique, leur fluorescence ne peut être distinguée au microscope. Comme cette fluorescence est utilisée pour les localiser, il devient impossible de déterminer avec précision leur position.
Cette limite de résolution a été traditionnellement surmontée dans les méthodes de microscopie à super-résolution en faisant clignoter les colorants et en activant et désactivant leur fluorescence. Cela sépare temporellement leur fluorescence, ce qui permet de les distinguer et d'obtenir des localisations inférieures à la limite de résolution classique. Cependant, pour les applications impliquant l'étude de processus dynamiques rapides, cette astuce présente un inconvénient important : le clignotement empêche la localisation simultanée de plusieurs colorants. Cela réduit considérablement la résolution temporelle lors de l'étude de processus dynamiques impliquant plusieurs biomolécules.
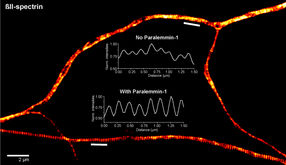
Sous la direction du professeur Philip Tinnefeld, chimiste à LMU, et en coopération avec le professeur Fernando Stefani (Buenos Aires), les chercheurs de LMU ont mis au point le multiplexage pMINFLUX, une approche élégante pour résoudre ce problème. L'équipe a récemment publié un article sur sa méthode dans la revue Nature Photonics. MINFLUX est une méthode de microscopie à super-résolution qui permet des localisations avec une précision de l'ordre du nanomètre. Contrairement au MINFLUX conventionnel, le pMINFLUX enregistre la différence de temps entre l'excitation des colorants par une impulsion laser et la fluorescence qui s'ensuit, avec une résolution inférieure à la nanoseconde. Outre la localisation des colorants, cela permet de comprendre une autre propriété fondamentale de leur fluorescence : leur durée de vie. Celle-ci décrit le temps moyen que met une molécule de colorant à devenir fluorescente après avoir été excitée.
"La durée de vie de la fluorescence dépend du colorant utilisé", explique Fiona Cole, coauteur de la publication. "Nous avons exploité les différences de durée de vie de la fluorescence lors de l'utilisation de différents colorants pour attribuer les photons fluorescents au colorant qui a émis sans qu'il soit nécessaire de cligner des yeux et sans la séparation temporelle qui en résulte. À cette fin, les chercheurs ont adapté l'algorithme de localisation et ont inclus un modèle d'ajustement multiexponentiel pour obtenir la séparation requise. "Cela nous a permis de déterminer la position de plusieurs colorants simultanément et d'étudier les processus dynamiques rapides entre plusieurs molécules avec une précision de l'ordre du nanomètre", ajoute Jonas Zähringer, également coauteur. Les chercheurs ont démontré leur méthode en suivant avec précision deux brins d'ADN qui sautaient entre différentes positions sur une nanostructure d'origami d'ADN, ainsi qu'en séparant les mouvements de translation et de rotation d'une nanostructure d'origami d'ADN et en mesurant la distance entre les sites de fixation d'antigènes des anticorps. "Mais ce n'est que le début", déclare Philip Tinnefeld. "Je suis certain que le multiplexage pMINFLUX, avec sa haute résolution temporelle et spatiale, fournira à l'avenir de nouvelles informations sur les interactions entre les protéines et d'autres phénomènes biologiques.
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.