L'IA prédit le taux de travail des enzymes
"TurNuP surpasse les modèles précédents et peut même être utilisé avec succès pour des enzymes qui n'ont qu'une faible similarité avec celles de l'ensemble de données d'apprentissage"
Les enzymes jouent un rôle clé dans les processus métaboliques cellulaires. Pour permettre l'évaluation quantitative de ces processus, les chercheurs ont besoin de connaître ce que l'on appelle le "taux de renouvellement" (en abrégé : kcat) des enzymes. Dans la revue scientifique Nature Communications, une équipe de bioinformaticiens de l'université Heinrich Heine de Düsseldorf (HHU) décrit maintenant un outil permettant de prédire ce paramètre pour diverses enzymes à l'aide de méthodes d'intelligence artificielle.
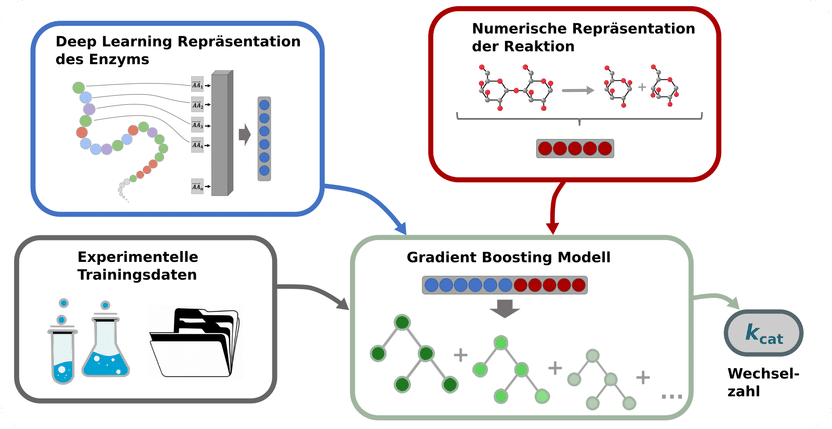
Schéma du processus de prédiction des nombres de renouvellement des réactions enzymatiques : Les enzymes sont des séquences d'acides aminés ; ces séquences sont converties en vecteurs numériques, représentés par des carrés gris, qui sont ensuite transformés en un seul vecteur par un modèle d'apprentissage profond (en haut à gauche). Les informations sur les réactions catalysées sont également converties en vecteurs numériques (en haut à droite). Les nombres de rotation déterminés expérimentalement (en bas à gauche) sont utilisés pour entraîner un modèle de boosting de gradient afin de prédire le nombre de rotation kcat (en bas à droite). Les modèles de gradient boosting sont un ensemble d'arbres de décision multiples, représentés dans différentes teintes de vert.
HHU / Alexander Kroll
Les enzymes sont des biocatalyseurs importants dans toutes les cellules vivantes. Il s'agit normalement de protéines de grande taille qui lient des molécules plus petites - appelées substrats - et les transforment ensuite en d'autres molécules, les "produits". Sans enzymes, la réaction qui convertit les substrats en produits ne pourrait pas avoir lieu, ou ne pourrait avoir lieu qu'à un rythme très faible. La plupart des organismes possèdent des milliers d'enzymes différentes. Les enzymes ont de nombreuses applications dans un large éventail de processus biotechnologiques et dans la vie quotidienne - de la fermentation de la pâte à pain aux détergents.
La vitesse maximale à laquelle une enzyme spécifique peut convertir ses substrats en produits est déterminée par ce que l'on appelle le taux de renouvellement kcat. Il s'agit d'un paramètre important pour la recherche quantitative sur les activités enzymatiques et il joue un rôle clé dans la compréhension du métabolisme cellulaire.
Cependant, il est long et coûteux de déterminer les nombres de renouvellement kcat dans les expériences, c'est pourquoi ils ne sont pas connus pour la grande majorité des réactions. Le groupe de recherche en biologie cellulaire computationnelle de l'HHU, dirigé par le professeur Martin Lercher, a mis au point un nouvel outil appelé TurNuP, qui permet de prédire les nombres de renouvellement kcat des enzymes à l'aide de méthodes d'intelligence artificielle.
Pour entraîner un modèle de prédiction kcat, les informations sur les enzymes et les réactions catalysées ont été converties en vecteurs numériques à l'aide de modèles d'apprentissage profond. Ces vecteurs numériques ont servi d'entrée à un modèle d'apprentissage automatique - un modèle dit de gradient boosting - qui prédit les nombres de renouvellement kcat.
Alexander Kroll, auteur principal de l'étude : "TurNuP surpasse les modèles précédents et peut même être utilisé avec succès pour des enzymes qui n'ont qu'une faible similarité avec celles de l'ensemble de données d'apprentissage." Les modèles précédents n'étaient pas en mesure de faire des prédictions significatives à moins qu'au moins 40 % de la séquence de l'enzyme ne soit identique à au moins une enzyme de l'ensemble de données d'apprentissage. En revanche, TurNuP peut déjà faire des prédictions significatives pour des enzymes dont l'identité de séquence maximale est comprise entre 0 et 40 %.
Le professeur Lercher ajoute : "Dans notre étude, nous montrons que les prédictions faites par TurNuP peuvent être utilisées pour prédire les concentrations d'enzymes dans les cellules vivantes avec beaucoup plus de précision que ce n'était le cas jusqu'à présent".
Afin de rendre le modèle de prédiction facilement accessible au plus grand nombre d'utilisateurs possible, l'équipe de l'HHU a développé un serveur web convivial, que d'autres chercheurs peuvent utiliser pour prédire les nombres de renouvellement kcat des enzymes.
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.