Une résistance aux antibiotiques jusqu'alors inconnue est répandue parmi les bactéries
"Nous avons identifié de nouveaux gènes de résistance dans des endroits où ils n'avaient pas été détectés jusqu'à présent.
Les gènes qui rendent les bactéries résistantes aux antibiotiques sont beaucoup plus répandus dans notre environnement qu'on ne le pensait auparavant. Une nouvelle étude, menée par l'université technologique de Chalmers et l'université de Göteborg en Suède, montre que les bactéries sont porteuses de gènes de résistance dans presque tous les environnements, ce qui risque de les propager et d'aggraver le problème des infections bactériennes qui ne peuvent être traitées par des antibiotiques.
"Nous avons identifié de nouveaux gènes de résistance dans des endroits où ils n'avaient pas été détectés jusqu'à présent. Ces gènes peuvent constituer une menace négligée pour la santé humaine", explique Erik Kristiansson, professeur au département des sciences mathématiques.
Selon l'Organisation mondiale de la santé (OMS), la résistance aux antibiotiques est l'une des plus grandes menaces pour la santé mondiale. Lorsque les bactéries deviennent résistantes aux antibiotiques, il devient difficile, voire impossible, de traiter des maladies telles que la pneumonie, les infections de plaies, la tuberculose et les infections des voies urinaires. Selon le groupe de coordination interagences des Nations unies sur la résistance aux antimicrobiens (IACG), 700 000 personnes meurent chaque année d'infections causées par des bactéries résistantes aux antibiotiques.
À la recherche de gènes de résistance dans de nouveaux environnements
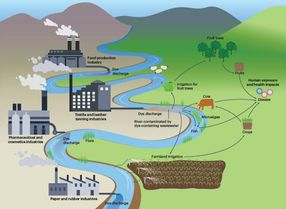
Les gènes qui rendent les bactéries résistantes sont étudiés depuis longtemps, mais l'accent a toujours été mis sur l'identification des gènes de résistance qui sont déjà répandus dans les bactéries pathogènes. Dans cette nouvelle étude suédoise, les chercheurs ont examiné de grandes quantités de séquences d'ADN de bactéries pour analyser de nouvelles formes de gènes de résistance et comprendre leur fréquence. Ils ont tracé les gènes dans des milliers d'échantillons bactériens provenant de différents environnements, sur et dans les personnes, dans le sol et dans les stations d'épuration. L'étude a analysé 630 milliards de séquences d'ADN au total.
"Les données nécessitent un traitement important avant de pouvoir obtenir des informations. Nous avons utilisé la métagénomique, une méthodologie qui permet d'analyser de grandes quantités de données", explique Juan Inda Díaz, doctorant au département des sciences mathématiques et auteur principal de l'article.
L'étude a montré que les nouveaux gènes de résistance aux antibiotiques sont présents dans les bactéries de presque tous les environnements. Cela inclut également nos microbiomes - les gènes des bactéries présentes dans et sur les personnes - et, plus inquiétant, les bactéries pathogènes, qui peuvent entraîner davantage d'infections difficiles à traiter. Les chercheurs ont découvert que les gènes de résistance dans les bactéries qui vivent sur et dans les humains et dans l'environnement étaient dix fois plus abondants que ceux qui étaient connus jusqu'à présent. Et parmi les gènes de résistance trouvés dans les bactéries du microbiome humain, 75 % n'étaient pas du tout connus auparavant.
Les chercheurs soulignent la nécessité d'approfondir les connaissances sur le problème de la résistance aux antibiotiques.
"Avant cette étude, on ne savait rien de l'incidence de ces nouveaux gènes de résistance. La résistance aux antibiotiques est un problème complexe, et notre étude montre que nous devons améliorer notre compréhension du développement de la résistance chez les bactéries et des gènes de résistance qui pourraient constituer une menace à l'avenir", déclare Kristiansson.
L'espoir de prévenir les épidémies bactériennes dans le secteur des soins de santé
L'équipe de recherche travaille actuellement à l'intégration des nouvelles données dans le projet international EMBARK (Establishing a Monitoring Baseline for Antibiotic Resistance in Key environments). Ce projet, coordonné par Johan Bengtsson-Palme, professeur assistant au département des sciences de la vie de Chalmers, vise à prélever des échantillons à partir de sources telles que les eaux usées, le sol et les animaux, afin de se faire une idée de la manière dont la résistance aux antibiotiques se propage entre l'homme et l'environnement.
"Il est essentiel que les nouvelles formes de gènes de résistance soient prises en compte dans l'évaluation des risques liés à la résistance aux antibiotiques. Les techniques que nous avons mises au point nous permettent de surveiller ces nouveaux gènes de résistance dans l'environnement, dans l'espoir de les détecter chez les bactéries pathogènes avant qu'elles ne puissent provoquer des épidémies dans un établissement de soins de santé", explique Mme Bengtsson-Palme.
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.