Structure atomique d'un bactériophage staphylococcique par cryo-microscopie électronique exposée
La cryo-microscopie électronique réalisée par des chercheurs de l'Université d'Alabama à Birmingham a permis d'exposer la structure d'un virus bactérien avec des détails sans précédent. Il s'agit de la première structure d'un virus capable d'infecter Staphylococcus epidermidis. La connaissance à haute résolution de la structure est un lien essentiel entre la biologie virale et l'utilisation thérapeutique potentielle du virus pour étouffer les infections bactériennes.
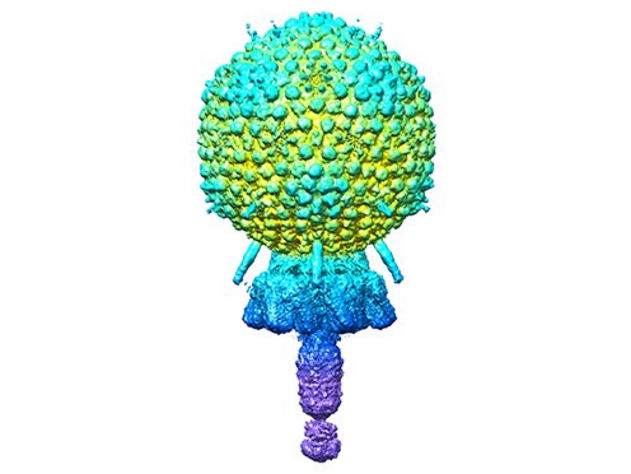
Le phage d'Andhra
UAB, Dokland lab
Les bactériophages ou "phages" sont les termes utilisés pour désigner les virus qui infectent les bactéries. Les chercheurs de l'UAB, dirigés par Terje Dokland, Ph.D., en collaboration avec Asma Hatoum-Aslan, Ph.D., de l'Université de l'Illinois Urbana-Champaign, ont décrit des modèles atomiques pour tout ou partie de 11 protéines structurelles différentes du phage Andhra. L'étude est publiée dans Science Advances.
Andhra est un membre du groupe des picovirus. Sa gamme d'hôtes est limitée à S. epidermidis. Cette bactérie de la peau est généralement bénigne, mais elle est aussi l'une des principales causes d'infection des dispositifs médicaux à demeure. "Les picovirus sont rarement présents dans les collections de phages et restent sous-étudiés et sous-utilisés pour des applications thérapeutiques", a déclaré M. Hatoum-Aslan, biologiste spécialiste des phages à l'université de l'Illinois.
Avec l'émergence de la résistance aux antibiotiques chez S. epidermidis et l'agent pathogène connexe Staphylococcus aureus, les chercheurs s'intéressent à nouveau à l'utilisation potentielle des bactériophages pour traiter les infections bactériennes. Les picovirus tuent toujours les cellules qu'ils infectent, après s'être liés à la paroi cellulaire bactérienne, avoir brisé cette paroi par voie enzymatique, pénétré la membrane cellulaire et injecté l'ADN viral dans la cellule. Ils présentent également d'autres caractéristiques qui en font des candidats intéressants pour une utilisation thérapeutique, notamment un petit génome et une incapacité à transférer des gènes bactériens entre bactéries.
La connaissance de la structure des protéines d'Andhra et la compréhension de la manière dont ces structures permettent au virus d'infecter une bactérie permettront de produire des phages sur mesure, adaptés à un objectif spécifique, en utilisant la manipulation génétique.
"La base structurelle de la spécificité d'hôte entre les phages qui infectent S. aureus et S. epidermidis est encore mal comprise", a déclaré Dokland, professeur de microbiologie à l'UAB et directeur du centre de microscopie cryo-électronique de l'UAB. "Grâce à la présente étude, nous avons acquis une meilleure compréhension des structures et des fonctions des produits du gène Andhra et des déterminants de la spécificité de l'hôte, ouvrant la voie à une conception plus rationnelle de phages personnalisés pour des applications thérapeutiques. Nos résultats élucident les caractéristiques critiques pour l'assemblage du virion, la reconnaissance de l'hôte et la pénétration."
Les phages staphylococciques ont généralement une gamme étroite de bactéries qu'ils peuvent infecter, en fonction des polymères variables de l'acide teichoïque de la paroi à la surface des différentes souches bactériennes. "Cette gamme d'hôtes étroite est une arme à double tranchant : D'une part, elle permet aux phages de cibler uniquement l'agent pathogène spécifique à l'origine de la maladie ; d'autre part, elle signifie que le phage doit être adapté au patient dans chaque cas spécifique", a déclaré M. Dokland.
La structure générale d'Andhra est une tête de capside icosaédrique arrondie à 20 faces qui contient le génome viral. La capside est attachée à une courte queue. Cette queue est en grande partie responsable de la liaison avec S. epidermidis et de la rupture enzymatique de la paroi cellulaire. L'ADN viral est injecté dans la bactérie par la queue. Les segments de la queue comprennent le portail de la capside à la queue, ainsi que la tige, les appendices, le bouton et l'extrémité de la queue.
Les 11 protéines différentes qui composent chaque particule virale se trouvent en plusieurs exemplaires qui s'assemblent ensemble. Par exemple, la capside est constituée de 235 copies de deux protéines chacune, et les neuf autres protéines du virion ont des nombres de copies allant de deux à 72. Au total, le virion est constitué de 645 morceaux de protéines, dont deux copies d'une 12e protéine, dont la structure a été prédite à l'aide du programme de prédiction de la structure des protéines AlphaFold.
Les modèles atomiques décrits par Dokland, Hatoum-Aslan et les co-premiers auteurs N'Toia C. Hawkins, Ph.D., et James L. Kizziah, Ph.D., du département de microbiologie de l'UAB, montrent les structures de chaque protéine - telles qu'elles sont décrites dans le langage moléculaire comme l'hélice alpha, l'hélice bêta, le brin bêta, le barillet bêta ou le prisme bêta. Les chercheurs ont décrit comment chaque protéine se lie à d'autres copies de ce même type de protéine, par exemple pour constituer les faces hexamériques et pentamériques de la capside, ainsi que la manière dont chaque protéine interagit avec les différents types de protéines adjacentes.
Les microscopes électroniques utilisent un faisceau d'électrons accélérés pour éclairer un objet, ce qui permet d'obtenir une résolution beaucoup plus élevée qu'avec un microscope optique. La cryo-microscopie électronique ajoute l'élément de températures super-froides, ce qui la rend particulièrement utile pour la résolution de la structure quasi atomique de protéines plus grandes, de protéines membranaires ou d'échantillons contenant des lipides comme les récepteurs liés à la membrane, et de complexes de plusieurs biomolécules ensemble.
Au cours des huit dernières années, de nouveaux détecteurs d'électrons ont permis à la cryo-microscopie électronique d'améliorer considérablement la résolution par rapport à la microscopie électronique normale. Les éléments clés de cette "révolution de la résolution" pour la microscopie cryo-électronique sont les suivants :
- La congélation instantanée d'échantillons aqueux dans de l'éthane liquide refroidi à moins de -256 degrés F. Au lieu de cristaux de glace qui perturbent les échantillons et dispersent le faisceau d'électrons, l'eau gèle en une "glace vitreuse" semblable à une fenêtre.
- L'échantillon est maintenu à des températures super froides dans le microscope, et une faible dose d'électrons est utilisée pour éviter d'endommager les protéines.
- Des détecteurs d'électrons directs extrêmement rapides sont capables de compter les atomes individuels à des centaines d'images par seconde, ce qui permet de corriger le mouvement de l'échantillon à la volée.
- L'informatique avancée fusionne des milliers d'images pour générer des structures tridimensionnelles à haute résolution. Des unités de traitement graphique sont utilisées pour traiter des téraoctets de données.
- La platine du microscope qui tient l'échantillon peut également être inclinée au fur et à mesure que les images sont prises, ce qui permet de construire une image tomographique tridimensionnelle, similaire à un scanner à l'hôpital.
L'analyse de la structure du virion Andhra par les chercheurs de l'UAB a commencé par 230 714 images de particules. La reconstruction moléculaire de la capside, de la queue, de la queue distale et de l'extrémité de la queue a commencé avec 186 542, 159 489, 159 489 et 159 489 images, respectivement. La résolution était comprise entre 3,50 et 4,90 angströms.
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.