L'intelligence artificielle plie les molécules d'ARN
De nouvelles structures fiables et prévisibles
La structure spatiale des molécules d'ARN est cruciale pour leur fonction. Mais la prédire est un défi.
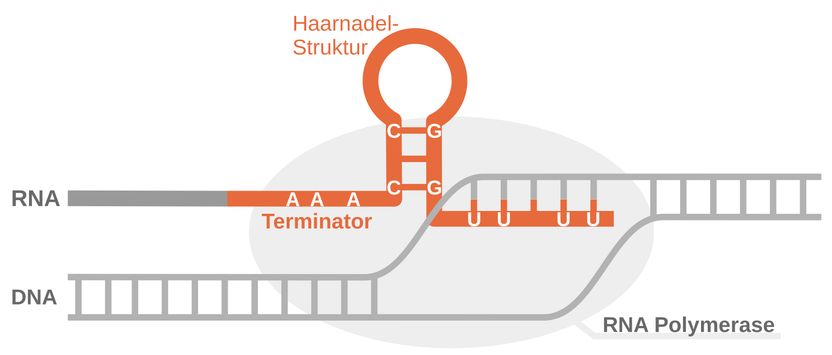
Certains segments d'une molécule d'ARN (orange) forment des structures dites en épingle à cheveux.
© Vivian Brandenburg
Pour la fonction de nombreuses biomolécules, leur structure tridimensionnelle est cruciale. Les chercheurs ne s'intéressent donc pas seulement à la séquence des différents éléments constitutifs des biomolécules, mais aussi à leur structure spatiale. Grâce à l'intelligence artificielle (IA), les bioinformaticiens peuvent déjà prédire de manière fiable la structure tridimensionnelle d'une protéine à partir de sa séquence d'acides aminés. Pour les molécules d'ARN, en revanche, cette technologie n'en est encore qu'à ses débuts. Des chercheurs de la Ruhr-Universität Bochum (RUB) décrivent dans la revue PLOS Computational Biology du 7 juillet 2022 un moyen d'utiliser l'IA pour prédire de manière fiable la structure de certaines molécules d'ARN à partir de leur séquence de nucléotides.
Pour ces travaux, les équipes dirigées par Vivian Brandenburg et le professeur Franz Narberhaus, de la chaire de biologie des micro-organismes de la RUB, ont coopéré avec le professeur Axel Mosig, du domaine de compétence en bioinformatique du centre de diagnostic des protéines de Bochum.
L'environnement cellulaire doit être pris en compte
"L'ARN est souvent considéré uniquement comme un messager entre l'ADN génomique et les protéines", explique Axel Mosig. "Mais de nombreuses molécules d'ARN prennent en charge des fonctions cellulaires". Leur structure spatiale est importante pour cela. Des régions similaires dans une séquence de nucléotides peuvent se regrouper pour former des arrangements tridimensionnels.
"Identifier ces autosimilarités dans une séquence d'ARN est comme un puzzle mathématique", explique Vivian Brandenburg. Il existe un modèle biophysique pour ce puzzle, avec les algorithmes de prédiction correspondants. Cependant, le modèle ne peut pas prendre en compte l'environnement cellulaire de l'ARN, qui influence également le processus de repliement. "Si l'ARN était isolé et flottait dans une solution aqueuse, le modèle pourrait prédire la structure avec une grande précision", explique M. Brandenburg. Mais une cellule vivante contient de nombreux autres composants.
C'est là que l'intelligence artificielle entre en jeu. L'IA peut apprendre des modèles subtils de l'environnement cellulaire sur la base de structures connues. Elle pourrait ensuite intégrer ces découvertes dans ses prédictions structurelles. Pour le processus d'apprentissage, l'IA a toutefois besoin de suffisamment de données d'entraînement, ce qui fait défaut dans la pratique.
Obtenir des données d'entraînement avec une astuce
Pour résoudre le problème des données d'entraînement manquantes, l'équipe de Bochum a eu recours à une astuce : les chercheurs ont travaillé avec des motifs structurels d'ARN connus. En utilisant une sorte de mécanisme inverse, ils ont pu générer un nombre presque illimité de séquences nucléotidiques à partir des modèles énergétiques de ces structures qui se plieraient dans ces structures spatiales. À l'aide de ce pliage dit inverse, les chercheurs ont généré de nombreuses paires de séquences nucléotidiques et de structures avec lesquelles ils ont pu entraîner l'IA.
De nouvelles structures prévisibles de manière fiable
Les chercheurs ont ensuite confronté l'IA à une nouvelle tâche : elle devait prédire la structure de certaines molécules d'ARN bactériennes. Ces molécules - appelées terminateurs de transcription - sont des signaux d'arrêt importants dans la traduction de l'ADN génomique chez les bactéries. Souvent, comme de nombreuses autres molécules d'ARN ayant des fonctions cellulaires importantes, elles sont cachées dans le génome et difficiles à distinguer des zones ayant d'autres fonctions.
L'intelligence artificielle a pu reconnaître et prédire de manière fiable la structure typique des terminateurs de transcription, qui rappelle une épingle à cheveux. L'équipe de recherche a pu le prouver en utilisant des données expérimentales accessibles au public.
"Alors que les approches de l'IA sont désormais presque inévitables dans la prédiction des structures des protéines, le développement des structures des ARN ne fait que commencer", résume Axel Mosig.
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.























































