Les structures considérées comme essentielles à l'expression des gènes sont étonnamment fugaces.
Une étude révèle que les boucles génomiques ne durent pas longtemps dans les cellules ; les théories sur la façon dont les boucles contrôlent l'expression des gènes doivent peut-être être révisées.
Dans les chromosomes humains, l'ADN est recouvert de protéines pour former un fil perlé extrêmement long. Cette "corde" est repliée en de nombreuses boucles, dont on pense qu'elles aident les cellules à contrôler l'expression des gènes et facilitent la réparation de l'ADN, entre autres fonctions. Une nouvelle étude menée par le Massachusetts Institute of Technology (MIT) en collaboration avec le Max Planck Institute of Molecular Cell Biology and Genetics (MPI-CBG) de Dresde, en Allemagne, et le Center for Systems Biology Dresden (CSBD) suggère que ces boucles sont très dynamiques et ont une durée de vie plus courte qu'on ne le pensait auparavant. Dans cette nouvelle étude, les chercheurs ont pu suivre le mouvement d'une portion du génome dans une cellule vivante pendant environ deux heures. Ils ont constaté que ce segment n'était entièrement bouclé que 3 à 6 % du temps, et que la boucle ne durait que 10 à 30 minutes. Selon les chercheurs, ces résultats suggèrent que la compréhension actuelle par les scientifiques de la manière dont les boucles influencent l'expression des gènes doit être révisée.
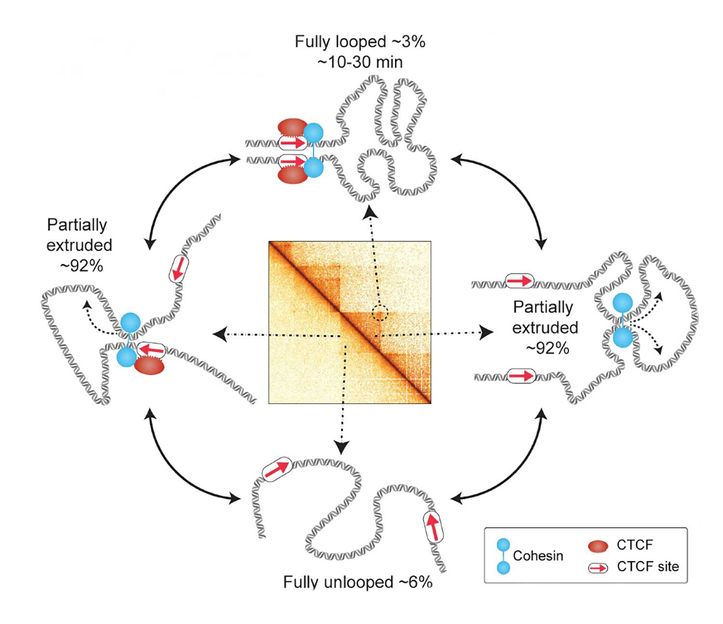
L'imagerie à super-résolution de cellules vivantes des boucles de chromatine médiées par CTCF/Cohesin révèle que ces boucles sont à la fois rares et dynamiques.
© Gabriele et al., Science
"De nombreux modèles dans le domaine ont été ces images de boucles statiques régulant ces processus. Ce que notre nouvel article montre, c'est que cette image n'est pas vraiment correcte", déclare Anders Sejr Hansen, professeur adjoint de développement de carrière Underwood-Prescott en génie biologique au MIT. "Nous suggérons que l'état fonctionnel de ces domaines est beaucoup plus dynamique". Hansen est l'un des auteurs principaux de la nouvelle étude, avec Leonid Mirny, professeur à l'Institute for Medical Engineering and Science et au département de physique du MIT, et Christoph Zechner, chef de groupe au MPI-CBG et au CSBD. Michele Gabriele, post-doc du MIT, Hugo Brandão, récemment titulaire d'un doctorat de l'université de Harvard, et Simon Grosse-Holz, étudiant diplômé du MIT, sont les principaux auteurs de l'article, qui paraît aujourd'hui dans Science.
Hors du circuit
À l'aide de simulations informatiques et de données expérimentales, les scientifiques du groupe de Mirny au MIT ont montré que les boucles du génome sont formées par un processus appelé extrusion, dans lequel un moteur moléculaire favorise la croissance de boucles de plus en plus grandes. Le moteur s'arrête chaque fois qu'il rencontre un "panneau d'arrêt" sur l'ADN. Le moteur qui extrude ces boucles est un complexe protéique appelé cohésine, tandis que la protéine CTCF liée à l'ADN fait office de panneau d'arrêt. Ces boucles médiées par la cohésine entre les sites CTCF ont été observées lors d'expériences précédentes. Cependant, ces expériences n'offraient qu'un instantané d'un moment donné, sans aucune information sur la façon dont les boucles évoluent dans le temps. Dans leur nouvelle étude, les chercheurs ont mis au point des techniques qui leur ont permis de marquer par fluorescence les sites d'ADN CTCF afin de pouvoir visualiser les boucles d'ADN sur plusieurs heures. Ils ont également créé une nouvelle méthode de calcul qui permet de déduire les événements de bouclage à partir des données d'imagerie. "Cette méthode était cruciale pour nous permettre de distinguer le signal du bruit dans nos données expérimentales et de quantifier les boucles", explique M. Zechner. "Nous pensons que de telles approches deviendront de plus en plus importantes pour la biologie, car nous continuons à repousser les limites de la détection avec des expériences."
Les chercheurs ont utilisé leur méthode pour imager une portion du génome dans des cellules souches embryonnaires de souris. "Si nous plaçons nos données dans le contexte d'un cycle de division cellulaire, qui dure environ 12 heures, la boucle entièrement formée n'existe en fait que pendant environ 20 à 45 minutes, soit environ 3 à 6 % du temps", explique Grosse-Holz.
"Si la boucle n'est présente que pendant une période aussi minuscule du cycle cellulaire et qu'elle est de très courte durée, nous ne devrions pas penser que cet état de boucle complète est le principal régulateur de l'expression génétique", déclare Hansen. "Nous pensons que nous avons besoin de nouveaux modèles pour comprendre comment la structure 3D du génome régule l'expression des gènes, la réparation de l'ADN et d'autres processus fonctionnels en aval." Si les boucles entièrement formées étaient rares, les chercheurs ont constaté que les boucles partiellement extrudées étaient présentes environ 92 % du temps. Ces boucles plus petites ont été difficiles à observer avec les méthodes précédentes de détection des boucles dans le génome. "Dans cette étude, en intégrant nos données expérimentales aux simulations de polymères, nous avons pu quantifier l'étendue relative des états non bouclé, partiellement extrudé et complètement bouclé", explique Brandão.
"Comme ces interactions sont très courtes, mais très fréquentes, les méthodologies précédentes n'étaient pas en mesure de saisir pleinement leur dynamique", ajoute Gabriele. "Avec notre nouvelle technique, nous pouvons commencer à résoudre les transitions entre les états entièrement bouclés et non bouclés".
Les chercheurs émettent l'hypothèse que ces boucles partielles pourraient jouer un rôle plus important dans la régulation des gènes que les boucles entièrement formées. Les brins d'ADN se déplacent les uns le long des autres lorsque les boucles commencent à se former puis se désagrègent, et ces interactions peuvent aider les éléments régulateurs tels que les exhausteurs et les promoteurs de gènes à se trouver les uns les autres. "Dans plus de 90 % des cas, il y a des boucles transitoires, et l'important est probablement d'avoir ces boucles qui sont perpétuellement extrudées", explique Mirny. "Le processus d'extrusion lui-même est peut-être plus important que l'état de boucle complète qui ne se produit que pendant une courte période de temps."
D'autres boucles à étudier
Étant donné que la plupart des autres boucles du génome sont plus faibles que celle que les chercheurs ont étudiée dans cet article, ils soupçonnent que de nombreuses autres boucles se révéleront également très transitoires. Ils prévoient maintenant d'utiliser leur nouvelle technique pour étudier certaines de ces autres boucles, dans divers types de cellules. "Il existe environ 10 000 de ces boucles, et nous n'en avons étudié qu'une seule", déclare Hansen. "Nous avons beaucoup de preuves indirectes qui suggèrent que les résultats pourraient être généralisés, mais nous ne l'avons pas démontré. En utilisant la plateforme technologique que nous avons mise en place, qui combine de nouvelles méthodes expérimentales et informatiques, nous pouvons commencer à aborder d'autres boucles dans le génome." Les chercheurs prévoient également d'étudier le rôle de boucles spécifiques dans les maladies. De nombreuses maladies, dont un trouble neurodéveloppemental appelé syndrome FOXG1, pourraient être liées à une dynamique de boucle défectueuse. Les chercheurs étudient actuellement comment la forme normale et mutée du gène FOXG1, ainsi que le gène cancérigène MYC, sont affectés par la formation de boucles dans le génome.
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.