Un nuevo método hace visibles las proteínas enmascaradas en las células nerviosas
"Este método abre nuevas puertas al estudio de proteínas difíciles de etiquetar en neuronas vivas"
Los grupos de investigación del Prof. Dr. Markus Sauer, del Biocentro, y del Dr. Gerti Beliu, del Centro Rudolf Virchow de Bioimagen Integrativa y Traslacional de la Universidad de Würzburg, así como el grupo del Dr. Daniel Choquet, del departamento de Neurocampus de la Universidad de Burdeos, desarrollaron un nuevo método que permitió visualizar por primera vez un complejo de proteína receptora y proteína auxiliar en neuronas vivas. "Este método abre nuevas puertas al estudio de proteínas difíciles de etiquetar en neuronas vivas", explica Sauer.
Los investigadores pueden ahora estudiar las interacciones de proteínas difíciles de etiquetar con una precisión inigualable. Imágenes de fluorescencia de una neurona viva que coexpresa una proteína fluorescente (cian) y la proteína auxiliar TARP marcada con un clic (magenta).
Choquet group and Beliu group.
Combinación de métodos innovadores y experiencia en neurociencia
Para visualizar las proteínas, los científicos combinaron el método de microscopía de superresolución dSTORM desarrollado por Sauer con el método de etiquetado de la química de clic. En esta técnica, un aminoácido artificial, los componentes básicos de las proteínas, se incorpora directamente a la proteína de interés. Un diminuto colorante se une a este aminoácido artificial, como una segunda pieza de un rompecabezas, y así se etiqueta directamente la proteína. Con este nuevo método, los investigadores pueden ver exactamente lo que ocurre en una célula, hasta el nivel molecular.
"En las células nerviosas, es especialmente importante qué proteína se encuentra en qué lugar de la célula y con qué otras proteínas está conectada. Porque esta organización molecular regula la transducción de señales de las neuronas y, por tanto, procesos tan importantes como el aprendizaje", explica Choquet. Los métodos de etiquetado convencionales, como el etiquetado con anticuerpos o la unión de proteínas fluorescentes, tienen un uso limitado, sobre todo en las neuronas vivas. Estos métodos pueden alterar el metabolismo normal de la célula debido al tamaño de las moléculas de etiquetado. Por lo tanto, también ha sido muy difícil estudiar los procesos moleculares exactos y la regulación de las proteínas individuales en las células vivas.
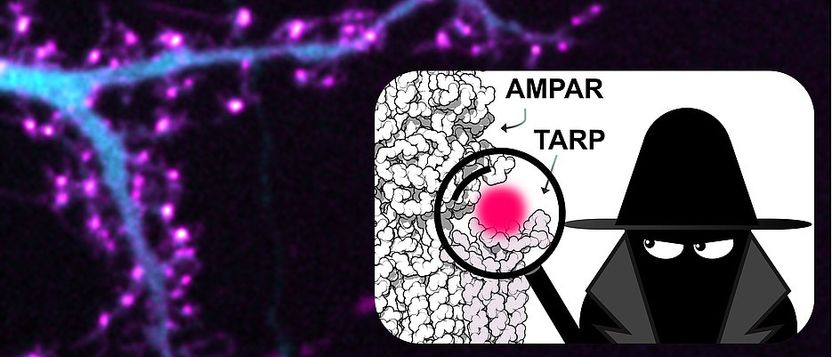
El sitio de unión oculto enmascara los receptores
El receptor AMPA es una proteína que ha despertado un gran interés en la neurociencia. Entre otras cosas, el receptor es importante para la transmisión sináptica. Pequeñas proteínas auxiliares (TARP) unidas al receptor regulan la actividad y la localización del mismo. Por estudios de biología estructural se sabía que el receptor y la proteína auxiliar podían formar un complejo. Aunque los científicos sospechaban que este complejo proteico debía estar presente en las sinapsis, aún no se había podido etiquetar las proteínas y visualizarlas al microscopio.
Ahora está claro: los receptores estaban literalmente enmascarados. La unión de las proteínas auxiliares a los receptores también cubría el sitio de unión del anticuerpo. Al no poder unirse, el etiquetado no era posible y los receptores quedaban así "invisibles". Con el nuevo método, optimizado especialmente para las neuronas, ahora es posible mostrar el complejo proteico de los receptores y la proteína auxiliar. La alta resolución espacial de dSTORM permite contar los receptores individualmente y estudiar específicamente su distribución en las células nerviosas. Los grupos de investigación ya están trabajando en el desenmascaramiento de otras proteínas para permitir su visualización mediante imágenes de fluorescencia de superresolución.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.
Publicación original
Diogo Bessa-Neto, Gerti Beliu, Alexander Kuhlemann, Valeria Pecoraro, Sören Doose, Natacha Retailleau, Nicolas Chevrier, David Perrais, Markus Sauer & Daniel Choquet; "Bioorthogonal labeling of transmembrane proteins with non-canonical amino acids unveils masked epitopes in live neurons"; Nature Communications; November 2021





















































