Seguimiento del ARN en el espacio y el tiempo
Nuevas vías para el estudio de aspectos como el mecanismo de acción de las terapias farmacológicas
Un equipo de investigación del MDC ha conseguido rastrear los genes en el espacio y el tiempo dentro de un embrión de pez cebra de una célula, incluso antes de que se produzca la división celular. Ahora han descrito en la revista "Nature Communications" un método que podría permitir algún día a los científicos medir la respuesta celular a los fármacos, por ejemplo, en los organoides.
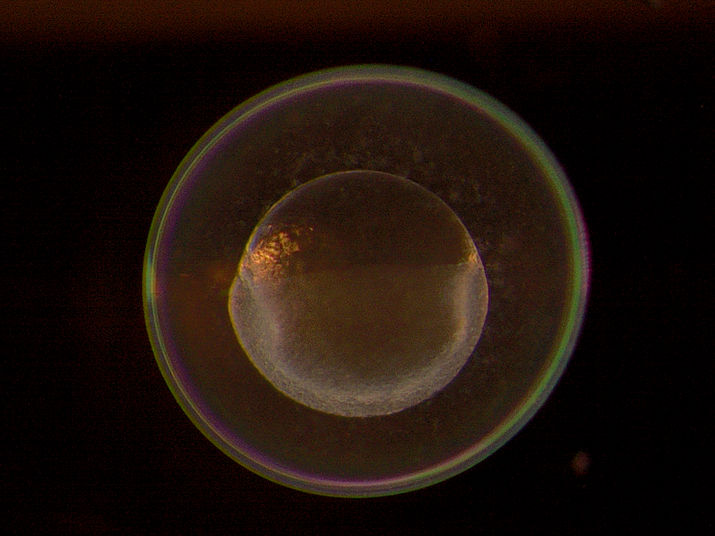
Embrión de pez cebra de una célula: El laboratorio de investigación del MDC encontró numerosos genes localizados en esta etapa temprana del desarrollo. Gran parte de su información genética fluye hacia las células precursoras de las posteriores células germinales
© AG Junker, MDC
El "milagro de la vida" es más evidente al principio: Cuando el óvulo fecundado se divide mediante surcos en blastómeros, se envuelve en un saco amniótico y se despliega para formar capas germinales. Cuando los blastómeros comienzan a diferenciarse en diferentes células, y cuando acaban por desarrollarse en un organismo completo.
"Queríamos averiguar si las diferencias posteriores entre las distintas células ya están en parte incorporadas en el óvulo fecundado", explica el Dr. Jan Philipp Junker, que dirige el Laboratorio de Biología Cuantitativa del Desarrollo en el Instituto de Biología de Sistemas de Berlín (BIMSB) del Centro Max Delbrück de Medicina Molecular de la Asociación Helmholtz (MDC). Junker y su equipo investigan cómo las células toman decisiones y qué dicta que se conviertan en células nerviosas, musculares o de la piel. Para ello, crean árboles de linaje celular que les permiten determinar el linaje y el tipo de célula de miles de células individuales de un organismo. Gracias a estos árboles de linaje, pueden entender cómo y por qué mecanismos se unen las células para formar un organismo funcional o cómo responden a las perturbaciones.
En el embrión unicelular ya existen los planos de los distintos tipos de células
Sin embargo, esta búsqueda de pistas mediante árboles de linaje celular comienza en una fase posterior, es decir, cuando la división y la diferenciación celular ya están en marcha. Además, las observaciones abarcan largos periodos de tiempo. En su estudio actual, Junker y su equipo se centran en un periodo de tiempo muy corto: las primeras horas después de la fecundación, desde el estadio unicelular hasta el proceso de gastrulación -la formación de las capas germinales- del embrión. Los científicos querían saber si el embrión unicelular ya contiene partes del modelo para la multitud de tipos de células diferentes que posteriormente se desarrollan a partir de él. Para ello, estudiaron embriones de pez cebra y de rana con garras. Anteriormente, los investigadores habían logrado encontrar genes individuales cuyo ARN se localiza en lugares específicos dentro de los embriones de pez cebra de una célula. Los científicos de Berlín han demostrado ahora que hay muchos más genes de este tipo. "Hemos descubierto diez veces más genes cuyo ARN se localiza espacialmente en el óvulo fecundado de lo que se conocía hasta ahora", explica Karoline Holler, autora principal del estudio. "Muchas de estas moléculas de ARN son transportadas posteriormente a las células germinales primordiales. Esto significa que el programa para la posterior diferenciación celular está conectado en el óvulo fecundado".
Nuevos enfoques en transcriptómica
Los métodos más modernos de transcriptómica unicelular permiten comprender bien la diferenciación celular. Los científicos ordenan las células individuales según la similitud de su transcriptoma -la colección completa de moléculas de ARN presentes en una célula- y pueden utilizar los patrones que surgen para descifrar cómo las células se convirtieron en lo que son. Sin embargo, no pueden utilizar este método para reconstruir las primeras etapas del desarrollo embrionario, porque aquí la disposición espacial de las moléculas de ARN
de las moléculas de ARN es crucial. En su lugar, su equipo utilizó una técnica especializada llamada tomo-seq, que Junker desarrolló en el Instituto Hubrecht de los Países Bajos en 2014. Permite a los científicos rastrear espacialmente las moléculas de ARN dentro de la célula. Esto se consigue cortando embriones de los organismos modelo en finas láminas. Entonces es posible leer los perfiles de ARN en las superficies cortadas y convertirlos en patrones de expresión espacial. Holler refinó la técnica de tomo-seq para medir ahora la distribución espacial del transcriptoma dentro del óvulo fecundado.
Los científicos utilizaron otra técnica nueva para estudiar qué genes localizados contribuyen posteriormente a qué células. "Etiquetamos las moléculas de ARN para poder seguirlas a lo largo de diferentes etapas de desarrollo. Esto nos permite observar el ARN no sólo en el espacio sino también en el tiempo", explica Junker. De este modo, los científicos pueden distinguir el ARN transferido al embrión por la madre del ARN producido por el propio embrión. Este método de etiquetado de ARN, denominado scSLAM-seq, se puso a punto en el BIMSB en los laboratorios del profesor Markus Landthaler y del profesor Nikolaus Rajewsky, lo que permitió aplicarlo en peces cebra vivos. "El etiquetado de moléculas de ARN nos permite medir con gran precisión cómo cambia la expresión génica en células individuales, por ejemplo, tras una intervención experimental", explica Junker.
¿Cómo afectan los fármacos a la diferenciación celular?
El etiquetado de ARN abre vías completamente nuevas para estudiar aspectos como el mecanismo de acción de las terapias farmacológicas. "Podemos utilizarlo en organoides para investigar cómo responden los distintos tipos de células a las sustancias", explica el físico. El método, dice Junker, no es adecuado para procesos de cambio a largo plazo. "Pero podemos ver qué genes cambian en las cinco o seis horas siguientes al tratamiento, lo que proporciona una vía para entender cómo podríamos influir en la diferenciación celular".
El análisis espacial también tiene relevancia médica: De cara al futuro, podría ser útil para estudiar aquellas enfermedades que son consecuencia de una mala localización del ARN, como el cáncer o las enfermedades neurodegenerativas. En estas enfermedades se transporta un gran número de moléculas a través de la célula. "Si entendemos estos procesos de transporte, tal vez podamos identificar factores de riesgo para estas enfermedades", explica Holler. Pero, por ahora, eso está muy lejos. "Todavía queda mucho trabajo por hacer antes de que el embrión de pez cebra de una célula pueda utilizarse como sistema modelo para estudiar las enfermedades neurodegenerativas humanas", subraya Junker.
Los científicos quieren descubrir a continuación los mecanismos implicados en la localización del ARN: ¿Cómo difiere el ARN detectado de otros transcritos en la célula? El equipo de Junker tiene previsto trabajar con el laboratorio de la profesora Irmtraud Meyer en el BIMSB para caracterizar las características de la secuencia del ARN localizado. Con la ayuda de algoritmos, esperan predecir si los genes localizados comparten un pliegue bidimensional o tridimensional. También están trabajando en el desarrollo de su método para que pueda utilizarse en otros sistemas además del embrión de pez cebra de una célula.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.