Desactivación de la "proteína de supervivencia" de las células cancerosas
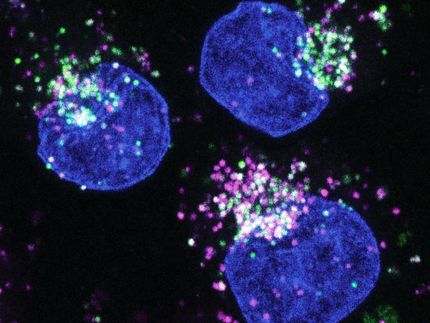
Se la denomina "proteína de la supervivencia" porque desempeña un papel central en el crecimiento de las células cancerosas: la survivina influye en dos procesos importantes de las células del organismo al mismo tiempo: la muerte y la división celular. Químicos y biólogos de la Universidad de Duisburgo-Essen (UDE) han logrado desarrollar una molécula precisa que puede unirse a la superficie de la proteína en un lugar definido y desactivarla.
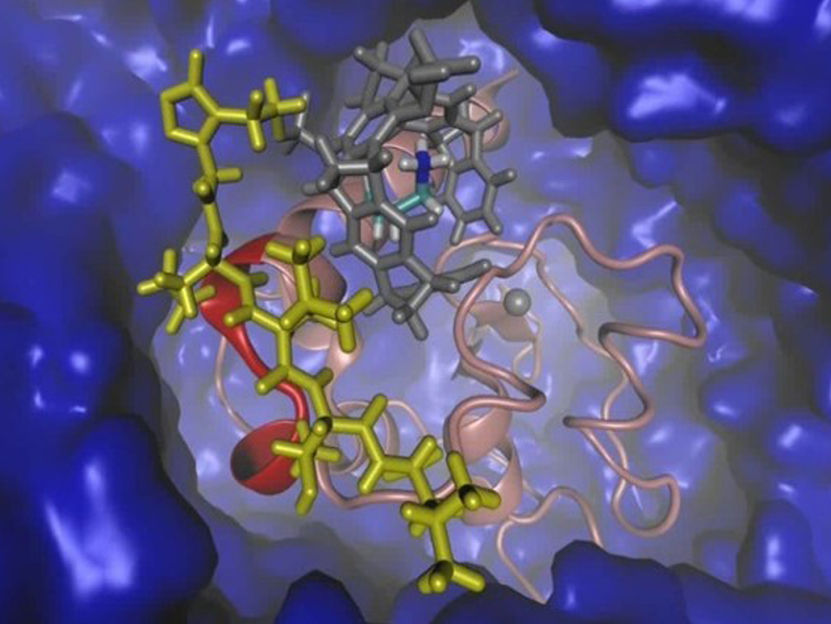
Representación gráfica de la proteína "Survivin" (rojo, rosa) y el ligando adaptado (gris, amarillo) en agua (azul).
© UDE/Sanchez-Garcia
Las proteínas controlan casi todos los procesos vitales de nuestras células. Si no funcionan correctamente, si hay demasiada o muy poca cantidad de ellas, esto puede conducir al desarrollo de una variedad de enfermedades, incluido el cáncer. Por ello, las proteínas asociadas son también objetivos importantes para el descubrimiento de fármacos en la investigación biomédica.
Sin embargo, hay un gran número de proteínas que sencillamente no ofrecen dianas adecuadas para que se acople un principio activo convencional. Por ello, los científicos del Centro de Investigación Colaborativa 1093 de la UDE están desarrollando pequeñas moléculas inusuales, denominadas ligandos supramoleculares, que pueden unirse con precisión a su superficie.
Las moléculas bloquean el sitio pivote de la proteína
Recientemente, el equipo de científicos dirigido por la Prof. Shirley Knauer, la Prof. Elsa-Sánchez-García y el Prof. Thomas Schrader ha conseguido dirigir una interfaz crítica que es importante para la supervivencia de las células cancerosas con dichas moléculas personalizadas. "La proteína survivin apenas se encuentra en los organismos adultos sanos", explica la profesora Shirley Knauer. "En las células cancerosas, sin embargo, su producción se dispara". Utilizando un ligando artificial a medida, los científicos pudieron cubrir el sitio exacto de la survivina, que es responsable de su activación y transporte fuera del núcleo celular.
Cada proteína tiene una estructura tridimensional única con una superficie fisurada que puede formar bucles y nichos. Sánchez-García y su equipo realizaron análisis computacionales de la superficie de la proteína y descubrieron que la interfaz importante se encuentra en un bucle ordenado pero algo dinámico. Con esta información y tras otros análisis estructurales, los químicos dirigidos por Schrader pudieron diseñar el ligando para esta superficie especialmente difícil.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.
Publicación original
A. Meiners, S. Bäcker, I. Hadrovic, C. Heid, C. Beuck, Y. B. Ruiz-Blanco, J. Mieres-Perez, M. Pörschke, J.-N. Grad, C. Vallet, D. Hoffmann, P. Bayer, E. Sanchez-Garcia, T. Schrader, S. K. Knauer; "Targeting a protein epitope: Specific inhibition of the Survivin-CRM1 interaction by peptide-modified molecular tweezers"; Nature Communications; 2021
Noticias más leídas
Publicación original
A. Meiners, S. Bäcker, I. Hadrovic, C. Heid, C. Beuck, Y. B. Ruiz-Blanco, J. Mieres-Perez, M. Pörschke, J.-N. Grad, C. Vallet, D. Hoffmann, P. Bayer, E. Sanchez-Garcia, T. Schrader, S. K. Knauer; "Targeting a protein epitope: Specific inhibition of the Survivin-CRM1 interaction by peptide-modified molecular tweezers"; Nature Communications; 2021
Organizaciones
Más noticias del departamento ciencias

Reciba la industria de las ciencias biológicas en su bandeja de entrada
Al enviar el formulario, se muestra usted de acuerdo con que LUMITOS AG le envíe por correo electrónico el boletín o boletines seleccionados anteriormente. Sus datos no se facilitarán a terceros. El almacenamiento y el procesamiento de sus datos se realiza sobre la base de nuestra política de protección de datos. LUMITOS puede ponerse en contacto con usted por correo electrónico a efectos publicitarios o de investigación de mercado y opinión. Puede revocar en todo momento su consentimiento sin efecto retroactivo y sin necesidad de indicar los motivos informando por correo postal a LUMITOS AG, Ernst-Augustin-Str. 2, 12489 Berlín (Alemania) o por correo electrónico a revoke@lumitos.com. Además, en cada correo electrónico se incluye un enlace para anular la suscripción al boletín informativo correspondiente.