El nuevo enfoque explica cómo los complejos de proteínas regulan los genes del cáncer
Nuevo punto de partida para mejorar las terapias contra el cáncer
Aunque se ha demostrado que las proteínas de fusión NUP98 causan leucemia, hasta ahora se desconocen los mecanismos moleculares. En un estudio recientemente publicado que fue dirigido por Vetmeduni Viena, los investigadores han sido capaces de descifrar una parte importante de estos mecanismos. Según el estudio, un posible impulsor de la activación de los genes de la leucemia es la formación de un complejo muy dinámico de proteínas de fusión NUP89 con otras proteínas en un proceso conocido como condensación biomolecular. Este hallazgo podría contribuir al desarrollo de terapias contra el cáncer más eficaces y específicas que serían especialmente beneficiosas para los niños que sufren de leucemia mieloide aguda (LMA).
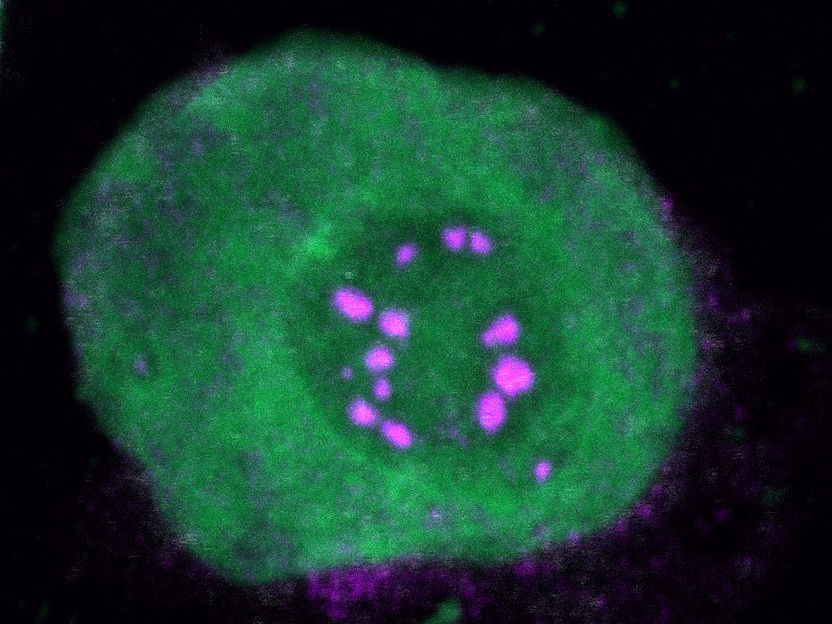
Las proteínas de fusión NUP89
S. Terlecki-Zaniewicz/CCRI
Los reordenamientos cromosómicos asociados al cáncer suelen dar lugar a la expresión de proteínas de fusión patógenas. La leucemia presenta una frecuencia particularmente alta de oncogenes de fusión, y la investigación funcional de las proteínas de fusión asociadas a la leucemia ha proporcionado conocimientos inestimables sobre los mecanismos moleculares del desarrollo del cáncer. Los complejos de proteínas en torno a las proteínas de fusión desempeñan un papel determinante en la configuración de los patrones de expresión génica oncogénica, por lo que la investigación de las interacciones entre las proteínas es fundamental para identificar nuevas dianas sobre cuya base se puedan desarrollar terapias oncológicas más eficaces.
Pronóstico extremadamente malo para los niños con LMA
Si bien las proteínas de fusión de la leucemia más comunes han sido ampliamente caracterizadas, todavía falta la comprensión funcional de las fusiones raras que afectan a un número significativo de pacientes y tienen opciones de tratamiento limitadas. En la leucemia mieloide aguda (LMA), la parte N-terminal del gen de la Nucleoporina 98 (NUP98) (N-NUP98) se fusiona con más de 30 loci de la pareja C-terminal diferentes. Aunque los reordenamientos del NUP98 son raros (representan alrededor del 2% de toda la LMA), están sobrerrepresentados en la LMA pediátrica, donde se asocian con un pronóstico particularmente malo.
Cómo las proteínas de fusión NUP98 impulsan la LMA
Todas las proteínas de fusión NUP98 comparten una región intrínsecamente desordenada (IDR) en el NUP98 N-terminal que presenta repeticiones de fenilalanina-glicina (FG), mientras que los compañeros de fusión C-terminal a menudo funcionan en el control de los genes. Los investigadores investigaron si los interacomas de las proteínas de fusión NUP98 proporcionaban algún tipo de conocimiento sobre los mecanismos de transformación oncogénica. "Los análisis de purificación de afinidad acoplados a la espectrometría de masas y a las imágenes confocales de cinco proteínas diferentes de fusión NUP98 revelaron que los interactores compartidos estaban enriquecidos para las proteínas implicadas en la condensación biomolecular. Estos factores se colocaron con las proteínas de fusión NUP98 en condensados característicos en el núcleo de la célula", dice el director del estudio Florian Grebien, Jefe del Instituto de Bioquímica Médica de la Universidad de Medicina Veterinaria de Viena. Una proteína artificial de fusión FG fenocopió los patrones de localización nuclear de las proteínas de fusión NUP98 y su capacidad para impulsar programas de expresión génica oncogénica.
Nuevo punto de partida para mejorar las terapias contra el cáncer
Los investigadores utilizaron un método recientemente desarrollado llamado espectrometría de masas por condensado mediado por isoxazol biotinilado (biCon-MS) para demostrar que las proteínas de fusión NUP98 alteran la composición global de los condensados biomoleculares para impulsar programas de expresión de genes oncogénicos. "Sobre la base de los datos recogidos en este estudio, proponemos que las proteínas de fusión que contienen IDR combinen la condensación biomolecular con el control transcripcional para inducir el cáncer", explica el primer autor Stefan Terlecki-Zaniewicz del Instituto de Bioquímica Médica de Vetmeduni Viena y St. La alteración de la condensación biomolecular podría ser, por lo tanto, un principio fundamental del desarrollo del cáncer impulsado por las proteínas de fusión oncogénica. Los investigadores creen que esto podría ser un nuevo enfoque para desarrollar terapias de cáncer más efectivas y específicas.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.