Evolución en tiempo real: Cómo se adaptan las bacterias a sus huéspedes
Algunas bacterias se vuelven cada vez más infecciosas cuando tienen que pasar de una célula a otra para sobrevivir
Las bacterias que invaden las células animales para multiplicarse están muy extendidas en la naturaleza. Algunas de ellas son patógenos de humanos y animales. En el medio ambiente, a menudo se encuentran dentro de organismos unicelulares. Un equipo de investigación dirigido por Matthias Horn en el Centro de Microbiología y Ciencia de Sistemas Ambientales de la Universidad de Viena ha hecho uso de experimentos de laboratorio para comprender mejor cómo estas bacterias se adaptan a su célula huésped con el tiempo y se vuelven cada vez más infecciosas en determinadas condiciones. Esto se debe a los cambios en el genoma y en la expresión de los genes, en particular en los genes que controlan la interacción de las bacterias con sus huéspedes y los responsables del metabolismo bacteriano.
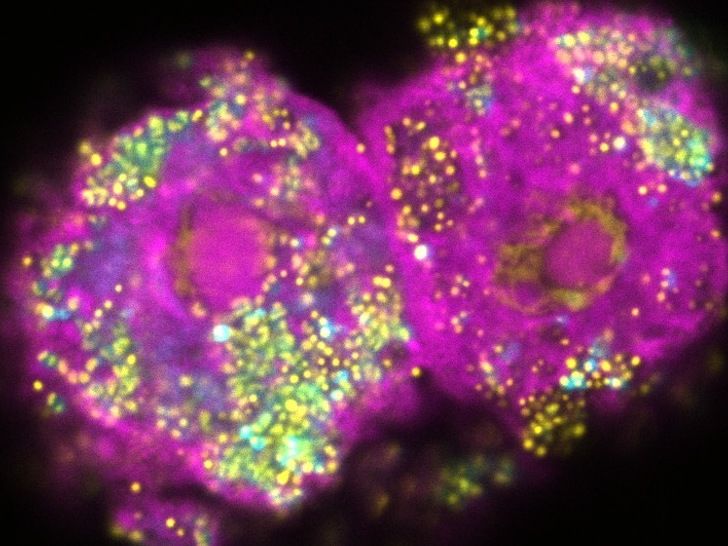
La paraclamidia está muy extendida en el entorno donde viven en las amebas. Las bacterias sirven como un sistema modelo para estudiar la evolución de la infectividad. La imagen muestra dos células de ameba en rosa que están infectadas con Parachlamydia (amarillo/azul).
© Patrick Arthofer
En el laboratorio del Centro de Microbiología y Ciencia de Sistemas Ambientales de la Universidad de Viena, se observó la llamada "Paraclamidia" después de un período de 14 meses de evolución dentro de su huésped unicelular. La paraclamidia es un género de clamidia ambiental que se encuentra en el agua o el suelo. A diferencia de sus parientes patógenos para los humanos, no son infecciosas para los humanos. Viven en organismos unicelulares y dependen de los nutrientes de sus huéspedes. Por lo tanto, con el paso del tiempo han perfeccionado los mecanismos para invadir las células huéspedes y reproducirse dentro de ellas.
Las predicciones teóricas confirmadas en el laboratorio
La paraclamidia sirve como un sistema modelo para estudiar la adaptación de las bacterias dependientes del huésped en el laboratorio. En el experimento evolutivo realizado con este fin participaron más de 500 generaciones de bacterias, lo que corresponde a un período de unos 15.000 años en el marco temporal humano.
Para verificar las predicciones teóricas sobre el desarrollo de la infecciosidad, las bacterias se mantuvieron en el laboratorio bajo dos condiciones experimentales diferentes. En una parte del experimento, las bacterias dependían de la infección frecuente de nuevas células huésped para sobrevivir. En la otra parte, fueron capaces de multiplicarse permanentemente dentro de una misma célula huésped. "Nuestros resultados revelan que si las bacterias son capaces de permanecer dentro de una célula anfitriona y asegurarse de que siguen viviendo en las células hijas del anfitrión cuando la célula anfitriona se divide, su infectividad no cambia. Sin embargo, las bacterias se vuelven cada vez más infecciosas cuando tienen que pasar de una célula anfitriona a otra para sobrevivir", explica Paul Herrera, primer autor del estudio.
La adaptación de la bacteria a nivel molecular
Los investigadores fueron un paso más allá en sus experimentos. Examinaron los genes de las bacterias al principio del proceso evolutivo y los compararon con los genes después de 500 generaciones de evolución. Encontraron que los genes de los dos grupos de bacterias diferían significativamente en 1.161 sitios.
Sin embargo, esta información genética por sí sola no era suficiente para explicar las diferencias en la infectividad. Sólo el análisis posterior de la expresión de los genes, es decir, el uso de los casi 2.500 genes durante la infección, reveló que las bacterias infecciosas, que tienen que cambiar entre las células huéspedes, mostraron cambios en la expresión de genes cruciales para el mecanismo de infección y para ciertas vías metabólicas que son importantes para la supervivencia fuera de las células huéspedes.
La vía de transmisión desempeña un papel crucial en el desarrollo de la infectividad en las bacterias dependientes del huésped. El aumento observado de la infecciosidad se basa en una variedad de diferencias genéticas y cambios importantes en la expresión de los genes. Estas modificaciones hacen que las células huéspedes se infecten más fácilmente y dan a la bacteria una mejor oportunidad de sobrevivir fuera de la célula huésped", resume Matthias Horn.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.
Publicación original
Paul Herrera, Lisa Schuster, Cecilia Wentrup, Lena König, Thomas Kempinger, Hyunsoo Naa, Jasmin Schwarz, Stephan Köstlbacher, Florian Wascher, Markus Zojer, Thomas Rattei, Matthias Horn; "Molecular causes of an evolutionary shift along the parasitism-mutualism continuum in a bacterial symbiont"; PNAS; 2020