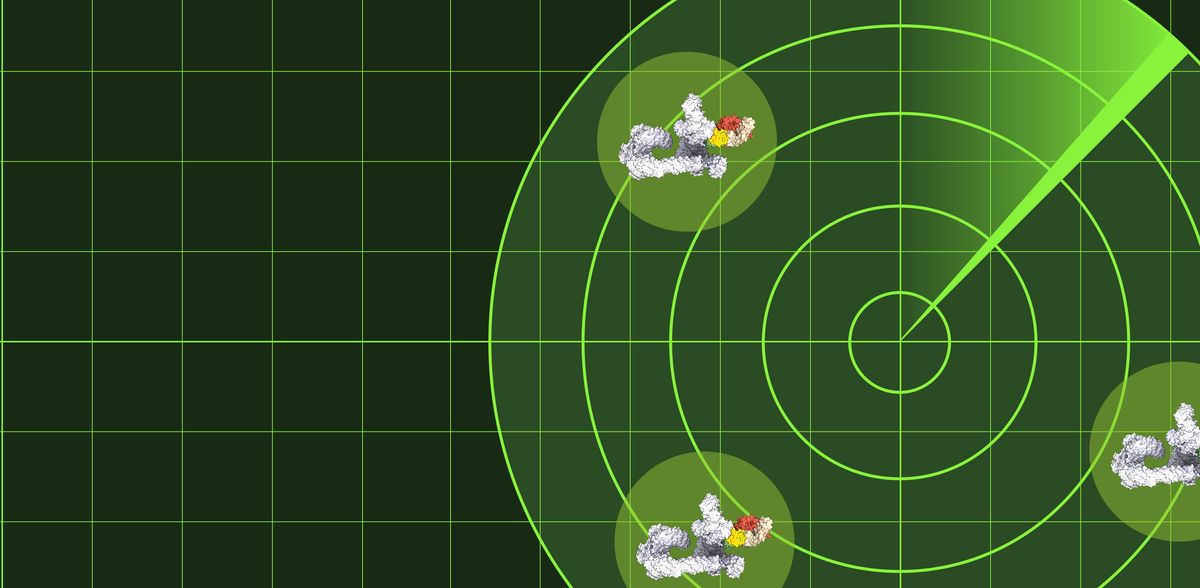
Un nouveau "radar" détecte les destructeurs cellulaires actifs
L'étude fournit un aperçu sans précédent des acteurs impliqués dans les changements dynamiques de notre équilibre protéique
Nos cellules disposent d'une flotte de 300 machines moléculaires, appelées cullin-RING ligases, ou "CRLs", qui sont toutes capables de déclencher la destruction de protéines spécifiques pour le bien-être de nos cellules. Toutefois, la plupart des protéines étant nécessaires, seules celles qui bloquent les voies cellulaires nouvellement requises ou qui sont toxiques doivent être détruites. Le potentiel destructeur des LCR est étroitement contrôlé. Les LCR ne sont activées que temporairement, en cas de besoin. Des chercheurs du MPI de biochimie et de l'université de Waterloo ont mis au point une nouvelle méthode pour détecter les LCR de la flotte qui sont "allumés", ce qui révèle les LCR déployés pour résoudre les stress cellulaires et pour effectuer les actions de certains médicaments anticancéreux.
Les cellules du corps humain doivent adapter leur équilibre protéique à certaines situations, telles que la disponibilité du fer ou une infection. Ces adaptations se produisent par le biais d'un processus complexe, dans lequel les protéines qui ne sont plus nécessaires ou qui sont toxiques sont marquées pour être détruites en y attachant une petite protéine appelée ubiquitine. Ce marquage d'une protéine en vue de sa destruction par l'ubiquitine est effectué par les ligases Cullin-RING, ou "CRL" en abrégé. Les LCR peuvent donc être considérées comme des "destructeurs" de molécules protéiques spécifiques.
Dans l'ensemble, les CRLs constituent la plus grande collection de machines de marquage de l'ubiquitine, avec plus de 300 membres. Le premier auteur, Lukas Henneberg, explique : "On peut imaginer que les 300 LCR différents à l'intérieur d'une cellule sont comme une flotte de destroyers, chacun d'entre eux étant capable de se verrouiller sur une cible différente. Si une cellule est exposée à une augmentation de fer ou à une bactérie infectieuse, les protéines qui augmenteraient encore le fer à des niveaux toxiques ou qui empêcheraient la réponse immunitaire de guérir l'infection doivent être détruites. Ces protéines sont ciblées par les destructeurs de LCR". Mais on ne sait pas lesquels des 300 LCR différents sont impliqués dans l'adaptation à de nombreuses conditions cellulaires différentes, car jusqu'à présent, il était difficile de détecter quels destructeurs spécifiques de la flotte étaient activés à un moment donné.
Quelles machines moléculaires Cullin-RING ligase sont actives et à quel moment ?
La flotte des LCR navigue essentiellement dans la mer qu'est l'intérieur de la cellule, chacune d'entre elles attendant individuellement un signal indiquant qu'elle est nécessaire. Lorsque le signal se produit, la LCR nécessaire est temporairement activée par la fixation d'une autre protéine, appelée NEDD8. Dès que l'action destructrice de la LCR n'est plus nécessaire, la LCR est désactivée par l'élimination de NEDD8.
Les chercheurs des laboratoires de Brenda Schulman, de l'Institut Max Planck (MPI) de biochimie, et de Sachdev Sidhu, de l'Université de Waterloo, ont mis au point la première d'une méthode en deux étapes pour détecter quelles LCR sont attachées à NEDD8, et donc activées.
Ils ont produit un anticorps synthétique reconnaissant les machines moléculaires CRL attachées à NEDD8. Les chercheurs ont déterminé une structure cristalline, c'est-à-dire une photographie moléculaire en 3D, qui montre comment l'anticorps peut capturer NEDD8 attaché à presque toutes les LCR, uniquement lorsqu'une LCR est activée pour permettre la destruction des protéines marquées à l'ubiquitine. L'anticorps synthétique est donc une sonde basée sur l'activité, ou un "radar moléculaire", qui peut détecter quelles LCR sont activées pour marquer leurs protéines cibles en vue de leur destruction.
Les scientifiques des départements de Brenda Schulman et de Matthias Mann au MPI de biochimie ont ensuite mis au point la deuxième étape de la nouvelle méthode afin de découvrir, parmi toute la flotte de LCR, celles qui sont activées dans des conditions cellulaires normales et celles qui sont activées pour s'adapter à l'évolution des besoins cellulaires. Les machines moléculaires CRL liées à l'anticorps, c'est-à-dire celles qui sont actives, ont été retirées des cellules et collectées afin d'utiliser une spectrométrie de masse de pointe pour mesurer quelles et combien de CRL étaient actives dans les cellules à un moment précis.
Les Cullin-RING ligases en médecine pharmaceutique
Dans l'étude actuelle, les auteurs ont pu identifier les LCR qui sont activées en réponse au fer et celles qui sont activées par des signes cellulaires d'inflammation. Les auteurs ont également étudié les LCR activées par l'action de médicaments dits "dégradeurs". Les médicaments dégradeurs sont des thérapies qui amènent un LCR à cibler une protéine pathogène pour la détruire. Pour l'instant, les médicaments dégradeurs sont utilisés pour traiter certains cancers, mais le concept est à l'étude pour d'autres maladies. La nouvelle méthode a montré que la quantité disponible de certaines LCR varie selon les types de cellules, ce qui influe sur l'efficacité des médicaments dégradeurs. Plus la quantité de "destructeur" de LCR déjà activée dans une cellule est importante, plus une molécule de dégradeur peut accélérer l'élimination de la protéine responsable de la maladie.
Les ligases Cullin-RING et notre système immunitaire
Les chercheurs ont également collaboré avec le laboratoire de Peter Murray au MPI de biochimie pour étudier les LCR actives dans des cellules appelées macrophages. Les macrophages sont des types de cellules spécifiques sur lesquelles notre système immunitaire s'appuie pour différentes fonctions de guérison. La comparaison des molécules de LCR actives des macrophages spécialisés dans la lutte contre les bactéries avec celles des macrophages spécialisés dans la cicatrisation des plaies a révélé des différences évidentes qui suggèrent les types d'adaptations nécessaires pour que les cellules macrophages puissent remplir ces fonctions très différentes.
Les résultats de cette étude donnent un aperçu sans précédent des acteurs impliqués dans les changements dynamiques de notre équilibre protéique et de leur implication dans les états pathophysiologiques, ce qui pourrait guider l'utilisation des LCR dans le développement de nouvelles thérapies à l'avenir.
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.
Publication originale
Lukas T. Henneberg, Jaspal Singh, David M. Duda, Kheewoong Baek, David Yanishevski, Peter J. Murray, Matthias Mann, Sachdev S. Sidhu & Brenda A. Schulman, Activity-based profiling of cullin–RING E3 networks by conformation-specific probes. Nature Chemical Biology, August 2023