Comment les moteurs moléculaires convertissent-ils l'énergie chimique en travail mécanique ?
Des chercheurs révèlent la structure et la fonction d'une micro-machine complexe
Les moteurs moléculaires sont des dispositifs complexes composés de nombreuses pièces différentes qui consomment de l'énergie pour réaliser diverses activités cellulaires. En bref, les moteurs moléculaires transforment l'énergie en travail utile. La compréhension des aspects mécaniques qui sous-tendent ces moteurs commence par une description détaillée de leur architecture globale et de leur organisation atomique. Cependant, pour découvrir les mécanismes fondamentaux qui alimentent ces moteurs, il est essentiel de décoder toute la dynamique moléculaire dans les moindres détails.
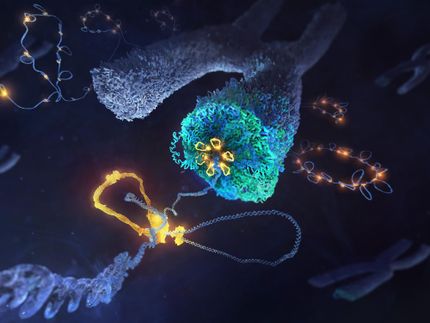
Représentation artistique de la jonction Holliday et des moteurs RuvB.
CSSB, Nicola Graf
Aujourd'hui, l'équipe de recherche de Thomas C. Marlovits, du Centre de biologie des systèmes structuraux CSSB de DESY et du Centre médical universitaire de Hambourg-Eppendorf (UKE), à Hambourg, révèle l'architecture, le cycle fonctionnel complet et le mécanisme d'un tel moteur moléculaire : Ils rapportent dans la revue Nature, comment un "complexe de migration de branche RuvAB" convertit l'énergie chimique en travail mécanique pour effectuer la recombinaison et la réparation de l'ADN.
La recombinaison de l'ADN est l'un des processus biologiques les plus fondamentaux des organismes vivants. C'est le processus par lequel les chromosomes "échangent" de l'ADN, soit pour générer de la diversité génétique, en créant une nouvelle progéniture, soit pour maintenir l'intégrité génétique, en réparant les cassures des chromosomes existants. Lors de la recombinaison de l'ADN, quatre bras d'ADN se séparent de leur formation en double hélice et se rejoignent à une intersection connue sous le nom de jonction de Holliday. C'est là que les bras d'ADN échangent leurs brins dans un processus appelé migration active des branches.
L'énergie essentielle nécessaire à cette migration des branches provient d'une machinerie moléculaire que les scientifiques ont baptisée complexe de migration des branches RuvAB. Ce complexe s'assemble autour de la jonction de Holliday et se compose de deux moteurs étiquetés RuvB AAA+ ATPases, qui alimentent la réaction, et d'un stator RuvA. L'équipe de recherche a maintenant fourni un plan détaillé qui explique comment les moteurs RuvB AAA+ fonctionnent sous la régulation de la protéine RuvA pour effectuer un mouvement synchronisé de l'ADN.
Les migrations des branches actives activées par la molécule du moteur RuvB AAA+ sont très rapides et très dynamiques. Pour déterminer les différentes étapes de ce processus, les scientifiques ont utilisé la cryo-microscopie électronique à résolution temporelle pour observer la machinerie du moteur au ralenti. "Nous avons essentiellement fourni au moteur AAA+ de la RuvB un carburant à combustion plus lente, ce qui nous a permis de saisir les réactions biochimiques au moment où elles se produisent", explique Marlovits.
Le scientifique a capturé plus de dix millions d'images de la machinerie du moteur interagissant avec la jonction de Holliday. Jiri Wald (CSSB, UKE et membre du programme de doctorat du BioCenter de Vienne), premier auteur de l'article, a passé au peigne fin l'immense quantité de données et a soigneusement classé les changements subtils survenant dans chaque image. Grâce à l'installation de calcul haute performance du DESY, les scientifiques ont ensuite pu rassembler toutes les pièces du puzzle pour générer un film haute résolution détaillant le fonctionnement du complexe RuvAB à l'échelle moléculaire.
"Nous avons pu visualiser sept états distincts du moteur et démontrer comment les éléments interconnectés fonctionnent ensemble de manière cyclique", explique Wald. "Nous avons également démontré que le moteur RuvB convertit l'énergie en un mouvement de levier qui génère la force qui entraîne la migration des branches. Nous avons été stupéfaits de découvrir que les moteurs utilisent un mécanisme de levier de base pour déplacer le substrat d'ADN. Globalement, le mécanisme séquentiel, la coordination et la manière de générer la force du moteur RuvAB présentent des similitudes conceptuelles avec les moteurs à combustion."
Les moteurs AAA+ sont souvent utilisés dans d'autres systèmes biologiques, tels que le transport des protéines. Par conséquent, ce modèle détaillé du moteur AAA+ de RuvB peut être utilisé comme modèle pour des moteurs moléculaires similaires. "Nous comprenons comment le moteur fonctionne et maintenant nous pouvons mettre ce moteur dans un autre système avec quelques adaptations mineures", explique Marlovits. "Nous présentons essentiellement les principes de base des moteurs AAA+."
Les futurs travaux du groupe de Marlovits exploreront les moyens d'interférer avec la fonction des moteurs AAA+. Cela pourrait servir de base au développement d'une nouvelle génération de médicaments, qui perturberaient les mécanismes de ce type de moteur chez les agents pathogènes et stopperaient ainsi la propagation de l'infection. "Nous sommes impatients d'explorer les possibilités qui existent maintenant que nous disposons d'un plan du moteur AAA+ de RuvB", note Wald.
Des scientifiques du CSSB, de l'UKE, de l'Institut de biotechnologie moléculaire, de l'Institut de recherche en pathologie moléculaire, tous deux situés à Vienne, en Autriche, et du DESY ont contribué à cette recherche.
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.