Descifrados los compuestos antibióticos del ribosoma
Lucha contra las bacterias multirresistentes mediante imágenes estructurales de alta resolución
Un equipo europeo de investigación dirigido por el Departamento de Química de la Universidad de Hamburgo presenta en un estudio las estructuras de 17 compuestos antibióticos ribosomales diferentes con una resolución de gran detalle. Estos conocimientos podrían allanar el camino hacia el desarrollo de nuevos antibióticos para combatir las bacterias multirresistentes.
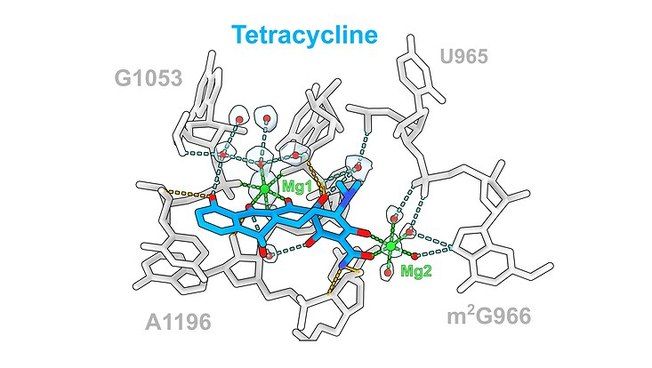
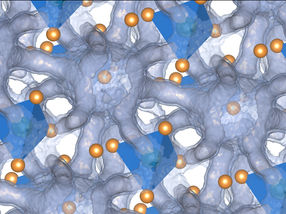
Antibiótico tetraciclina (azul) y sus interacciones con el ARNr ribosómico Foto: UHH/Paternoga El antibiótico tetraciclina (azul) y sus interacciones con el ARNr ribosómico (gris), que están mediadas por interacciones directas (naranja) o indirectas (cian) a través de Mg1 (verde) o moléculas de agua (rojo).
UHH/Paternoga
Los ribosomas son grandes complejos moleculares que se encuentran en las células de plantas, animales, humanos y bacterias y que producen proteínas. Por tanto, son dianas importantes para los antibióticos que se acoplan a una subunidad de los ribosomas en las bacterias y provocan errores de lectura o incluso paradas de lectura. El resultado es la formación de proteínas defectuosas que pierden su función biológica y la bacteria muere. Sin embargo, cada vez hay más bacterias multirresistentes que impiden la acción de los antibióticos.
Para eludir esta resistencia, es esencial conocer en detalle los antibióticos, especialmente sus estructuras ribosómicas. En las dos últimas décadas, se han publicado estructuras de ribosomas antibióticos para cada una de las principales clases de antibióticos dirigidos por ribosomas, lo que ha permitido comprender mejor sus sitios de unión y mecanismos de acción.
El equipo dirigido por el Prof. Dr. Daniel N. Wilson, del Departamento de Química de la Universidad de Hamburgo, ha visualizado ahora estas estructuras mediante criomicroscopía electrónica con una resolución de 1,6 a 2,2 Ångström (Å), donde un Å corresponde a la diezmillonésima parte de un milímetro. "En el pasado, las estructuras de los ribosomas antibióticos se presentaban principalmente con resoluciones de 2,5 a 3,5 Å", afirma el profesor Daniel N. Wilson. "La mejora de la resolución nos permite ahora observar incluso moléculas de agua que favorecen las interacciones entre fármacos". Los investigadores descubrieron que, en general, entre diez y veinte enlaces de hidrógeno interactuaban con el ribosoma, y que la contribución de las moléculas de agua variaba según las distintas clases de antibióticos.
Los antibióticos estudiados incluyen las seis familias de antibióticos clínicamente relevantes, entre ellas las tetraciclinas utilizadas para infecciones respiratorias, los aminoglucósidos con un amplio espectro de actividad, incluso para la tuberculosis o la meningitis, y las pleuromutilinas para infecciones de piel y tejidos blandos.
Para el estudio, el equipo de investigación aplicó estos antibióticos a los llamados ribosomas 70S de la bacteria "Escherichia coli", una bacteria coliforme que también se encuentra en el intestino humano. "Prevemos que esta información pueda utilizarse en el futuro para el diseño basado en estructuras de nuevos derivados de antibióticos. Al identificar las regiones que pueden alterarse, el agua ligada puede desplazarse específicamente para interactuar con el objetivo, que es la bacteria", explica Wilson.
La obtención de imágenes de alta resolución es un gran paso, según Wilson: "Las antiguas estructuras de ribosomas de antibióticos a menor resolución muestran que los sitios de unión para cada clase de antibiótico son similares, sin embargo, en muchos casos hay profundas diferencias en cuanto a la posición exacta de los fármacos, así como el sitio de unión del fármaco". Por tanto, los datos experimentales de alta resolución eran necesarios para obtener una descripción más precisa de las interacciones de los antibióticos con el ribosoma.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Alemán se puede encontrar aquí.