Seguimiento molecular de la regulación del ARN
Nueva herramienta biológica para el seguimiento de los procesos celulares
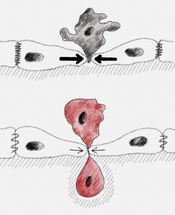
Cuanto mejor comprendamos procesos celulares como la regulación del ARN, mejor se podrán desarrollar las terapias moleculares. Hasta ahora, ha sido especialmente difícil seguir la regulación del ARN no codificante, que es el que no se convierte en proteínas. Un equipo de investigación de la Universidad Técnica de Múnich (TUM) y del Helmholtz de Múnich ha desarrollado ahora un sistema reportero mínimamente invasivo que permite un seguimiento muy sensible de la producción de ARN tanto codificante como no codificante.

Los intrones de ingeniería insertados en el circuito genético arrojan luz sobre la regulación del ARN (no) codificante
Barth van Rossum
En los procesos celulares, nuestra información genética de ADN se transcribe en ARN, que luego se somete a un procesamiento posterior antes de que sirva como un plano para las proteínas o realice una función celular en sí misma. Qué tipos de ARN se producen y en qué cantidades revela mucho sobre el estado de nuestras células. En caso de infección, por ejemplo, las células producen mayores cantidades de moléculas de ARN que codifican las proteínas que intervienen en la respuesta inmunitaria.
Cuando las moléculas de ADN se traducen en proteínas a través del ARN, los investigadores pueden seguir el proceso con los sistemas informadores existentes. Sin embargo, no todos los genes humanos codifican proteínas. La mayoría de los genes humanos son no codificantes, incluidos los genes de los ARN no codificantes largos (lncRNA). Se trata de moléculas de ARN con más de 200 bloques de construcción que no actúan como planos para las proteínas. En cambio, controlan procesos importantes en las células. Las primeras investigaciones muestran que los lncRNA participan en procesos como la regulación de la producción de ARN, la organización de estructuras en el núcleo celular o la activación y desactivación de determinadas enzimas.
A pesar de su importancia para los procesos celulares, ha sido difícil investigar los lncRNAs con los métodos existentes. Hasta ahora, esto sólo era posible parcialmente, por ejemplo en células fijadas en puntos temporales específicos, porque no se pueden utilizar los sistemas reporteros clásicos basados en la traducción a proteínas.
INSPECT permite el seguimiento de los ARN no codificantes
Ahora se ha encontrado una solución en forma de un nuevo sistema reportero INSPECT. Un equipo que trabaja con Gil Westmeyer, catedrático de Ingeniería Neurobiológica de la TUM y director del Instituto de Biomedicina Sintética del Helmholtz de Múnich, ha publicado el nuevo sistema reportero en la revista Nature Cell Biology.
"A diferencia de los métodos anteriores, INSPECT codifica secuencias para las proteínas reporteras en intrones modificados. Se trata de secuencias en la molécula de ARN prematuro que se eliminan de forma natural por la célula durante el procesamiento. INSPECT estabiliza los intrones de forma que, en lugar de degradarse tras su eliminación, son transportados al citoplasma celular donde se traducen en proteínas informadoras", explica el primer autor, Dong-Jiunn Jeffery Truong. Los investigadores pueden entonces utilizar métodos convencionales para detectar las señales de las proteínas informadoras, como la fluorescencia.
INSPECT no modifica ni el ARN terminado ni las proteínas
Así, la nueva herramienta de biología molecular no sólo resuelve el problema del seguimiento de la generación de ARN no codificante, sino que también ofrece ventajas para el estudio del ARN codificante. Los sistemas informadores actuales a menudo corren el riesgo de dañar el ARN o las proteínas que se investigan, por ejemplo, porque deben fusionarse directamente con el ARN que se estudia para que se cotraduzca en proteínas. En lugar de modificar el ARN completo o las proteínas, INSPECT modifica los intrones.
El equipo ha demostrado la función de INSPECT utilizando varios ejemplos de ARN codificante y no codificante. Han seguido la producción de ARN para la interleucina 2, una proteína que se produce en mayores cantidades en respuesta a las infecciones. También han logrado un seguimiento muy sensible de la producción de dos lncRNAs y han rastreado los cambios en la regulación durante el periodo de investigación.
"INSPECT añade una importante herramienta de biología molecular a la caja de herramientas biomédicas. Facilita el estudio del papel de ciertas moléculas de ARN no codificante en el desarrollo celular y la exploración de cómo puede modularse su regulación, por ejemplo, para evitar que se conviertan en células cancerosas", afirma el profesor Westmeyer. "En combinación con el sistema reportero mínimamente invasivo EXSISERS, que desarrollamos previamente para estudiar las isoformas de las proteínas, puede ser posible en el futuro estudiar todo un proceso de regulación genética desde el procesamiento del ARN hasta la producción de variantes proteicas específicas en células vivas."
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.
Publicación original
Dong-Jiunn Jeffery Truong*, Niklas Armbrust*, Julian Geilenkeuser, Eva-Maria Lederer, Tobias Heinrich Santl, Maren Beyer, Sebastian Ittermann, Emily Steinmaßl, Mariya Dyka, Gerald Raffl, Teeradon Phlairaharn, Tobias Greisle, Milica Živanić, Markus Grosch, Micha Drukker, Gil Gregor Westmeyer: Intron-encoded cistronic transcripts for minimally-invasive monitoring of coding and non-coding RNAs. Nature Cell Biology (2022). *contributed equally
DJ.J. Truong et al.: Non-invasive and high-throughput interrogation of exon-specific isoform expression. Nature Cell Biology 23, 652–663 (2021).