El complejo proteico ClpX-ClpP podría ser el punto de partida para nuevos antibióticos
Punto débil en bacterias patógenas
Los antibióticos siguen siendo el arma más importante para combatir las infecciones bacterianas. Pero la ciencia médica se está quedando sin "munición" debido a las resistencias cada vez más frecuentes. Un equipo de investigación ha aclarado la estructura del complejo proteolítico ClpX-ClpP. Esta es una clave para el desarrollo de antibióticos innovadores que se dirigen al proceso de degradación de proteínas defectuosas en bacterias.
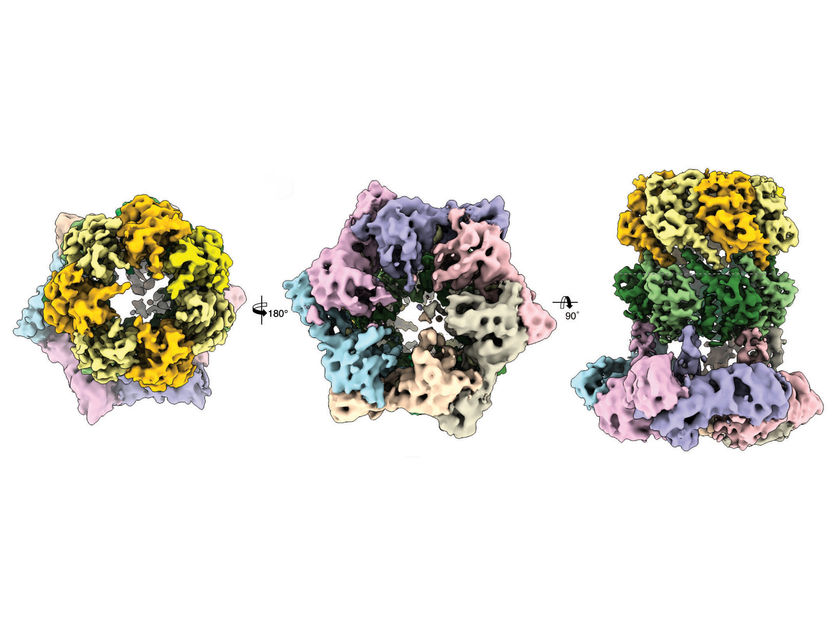
Tres vistas microscópicas crioelectrónicas del complejo proteico ClpX-ClpP.
C. Gatsogiannis / MPI für molekulare Physiologie
Casi 700.000 personas en Europa sufren infecciones cada año a través de patógenos resistentes a los antibióticos; aproximadamente 33.000 de ellas mueren. A pesar de este enorme y creciente peligro a nivel mundial, muy pocos antibióticos nuevos han sido desarrollados y aprobados en las últimas décadas.
No hay ninguna mejora a la vista. Por eso es urgente encontrar nuevos puntos de ataque en las bacterias patógenas y desarrollar nuevos antibióticos que aprovechen estos puntos débiles.
Un nuevo mecanismo de acción destruye las bacterias
Un punto de ataque especialmente prometedor para las terapias antibacterianas es la enzima proteolítica ClpP: por un lado, desempeña un papel importante en el metabolismo bacteriano y, por otro, garantiza la degradación controlada de las proteínas defectuosas.
Pero para ello necesita la proteína ClpX como ayuda de partida. En el complejo con ClpP, el ClpX identifica las proteínas que deben ser degradadas, las despliega y las guía hacia su cámara de degradación en forma de barril.
Los científicos de los grupos dirigidos por el Prof. Stephan Sieber, de la Universidad Técnica de Munich (TUM) y el Prof. Stefan Raunser, director del Instituto Max Planck de Fisiología Molecular de Dortmund, han esclarecido por primera vez la estructura tridimensional del complejo proteolítico ClpX-ClpP y han establecido así una base importante para futuras aplicaciones farmacológicas.
Una nueva clase de antibióticos potenciales - los llamados antibióticos acildepsipéptidos (ADEP) - también provocan una degradación incontrolada a través del ClpP sin el apoyo del ClpX. Como resultado, también se destruyen las proteínas vitales, con consecuencias letales para las bacterias.
Este mecanismo de acción único tiene un considerable potencial de innovación en la lucha contra las bacterias patógenas. Mientras que los antibióticos comunes actúan mediante la inhibición de procesos vitales, en este caso el efecto antibacteriano se consigue mediante la activación de un proceso.
Desarmar las bacterias
Además de la degradación de proteínas defectuosas, el ClpP es también un regulador decisivo en la producción de un arsenal de toxinas bacterianas que son las principales responsables del efecto patógeno de muchos patógenos.
En el TUM, el grupo dirigido por el Prof. Stephan Sieber ha estado investigando con éxito la proteasa ClpP durante años, y ya ha desarrollado un gran número de inhibidores potentes contra ClpP y ClpX que detienen la producción de toxinas bacterianas y por lo tanto pueden más o menos desarmarlas. Dóra Balogh ha conseguido producir y estabilizar el complejo ClpX-ClpP.
Nuevas posibilidades a través de la elucidación de la estructura de ClpX-ClpPP
Sin embargo, hasta hace poco, la estructura del complejo ClpX-ClpP no se ha podido aclarar en detalle. El Dr. Christos Gatsogiannis, investigador del grupo dirigido por el Prof. Stefan Raunser en el MPI de Fisiología Molecular, lo ha conseguido ahora mediante microscopía electrónica criogénica.
Con esta tecnología se pudo demostrar que ADEP y ClpX se acoplan a ClpP en el mismo lugar, pero controlan el proceso de degradación de proteínas de una manera diferente: Mientras que el ClpX no provoca una alteración en la estructura del ClpP, el ADEP provoca una apertura involuntaria del complejo. Como resultado, las proteínas intactas también se degradan de forma incontrolada y sin el apoyo del ClpX.
La clarificación de este mecanismo por parte de los equipos de investigación de Dortmund y Munich es un hito en el camino hacia el desarrollo de sustancias antibióticas innovadoras dirigidas al ClpP.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.