Los investigadores visualizan la batalla entre el virus y las células huéspedes a nivel unicelular
Un método innovador identifica genes recién activados dentro de células individuales
Las células están equipadas con múltiples mecanismos de defensa para combatir a los intrusos. Los genes codifican los bloques de construcción de la respuesta que deben ser activados durante un ataque enemigo de manera ordenada. Científicos del Instituto Helmholtz para la Investigación de Infecciones Basada en ARN (HIRI) en Würzburg, uno de los sitios del Centro Helmholtz para la Investigación de Infecciones (HZI), y la Universidad Julius-Maximilians-Universität de Würzburg han desarrollado un nuevo método, el scSLAM-seq, que les permite rastrear la actividad de miles de genes en células individuales durante varias horas. Por primera vez, los investigadores pudieron explicar por qué algunas células están infectadas con éxito por un virus, mientras que otras no. Además, también obtuvieron nuevos conocimientos fundamentales sobre la regulación de los genes.
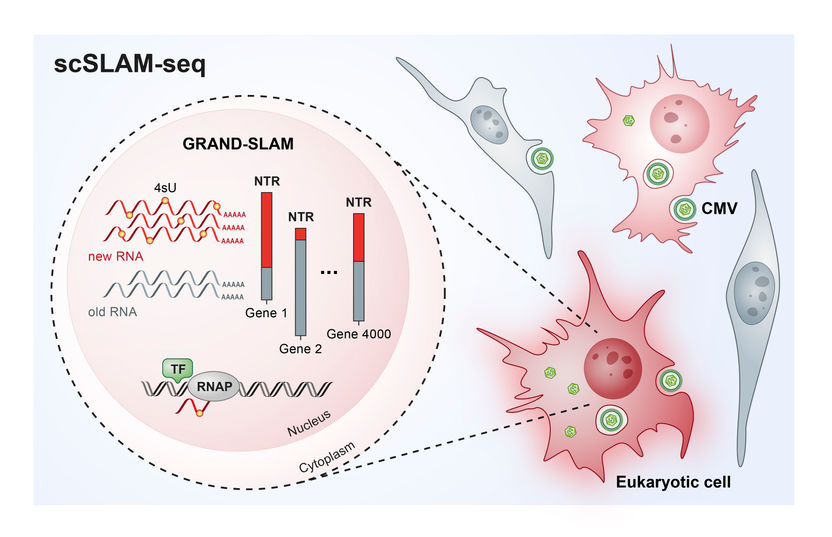
Ilustración del análisis unicelular con scSLAM-seq.
©HZI/HIRI/S. Pernitzsch.
Cuando los virus entran en nuestro cuerpo, como durante una gripe o una infección gastrointestinal, los procesos dentro de las células infectadas cambian: En el peor de los casos, el virus toma el timón y reprograma la célula a su favor. Luego produce componentes virales a escala masiva, permitiendo que el intruso se multiplique exponencialmente. En otras células, sin embargo, el virus puede ser eliminado con éxito mediante la activación de los mecanismos de defensa celular. Pero, ¿cómo puede ser que una célula sea invadida y otra logre controlar el virus? ¿Qué tan rápido reaccionan las células individuales a un ataque viral y qué genes protectores se activan? "Anteriormente, se sabía muy poco sobre estos temas a nivel unicelular", dice el Dr. Antoine-Emmanuel Saliba del Instituto Helmholtz para la Investigación de Infecciones Basadas en ARN (HIRI, por sus siglas en inglés). "Pero nuestro estudio actual nos ha permitido avanzar considerablemente en la respuesta a estas preguntas."
El Prof. Florian Erhard, del Instituto de Virología e Inmunobiología de la Universidad Julius-Maximilians-Universität (JMU) de Würzburg, Saliba del HIRI y el Prof. Lars Dölken, que trabaja tanto en el JMU como en el HIRI, se unieron para investigar cómo la actividad génica -que refleja la identidad y el estado fisiológico de una célula- cambia en las células individuales de los ratones infectados después de una infección con citomegalovirus. Los citomegalovirus están muy extendidos, con más del 80 por ciento de las personas infectadas. Generalmente, las personas sanas no son dañadas por el virus, sin embargo, una infección en recién nacidos o pacientes de trasplante puede llegar a ser peligrosa y tener consecuencias neurológicas graves. Un método experimental, la secuenciación de ARN unicelular (scRNAseq), que surgió hace sólo unos años, puede utilizarse para determinar qué genes de una célula están actualmente activos. Sin embargo, los cambios a corto plazo en la actividad de los genes, como los que ocurren durante una infección viral, apenas se pueden detectar. Además, cada célula individual sólo puede examinarse una vez. Con las tecnologías actuales, por lo tanto, no estaba claro cómo reaccionaban las células individuales a los estímulos externos, por ejemplo, una infección viral. Para investigar los procesos moleculares que tienen lugar en las células individuales infectadas, los investigadores desarrollaron un nuevo método, llamado scSLAM-seq, que, por primera vez, permite visualizar qué genes se activan en las células individuales en el plazo de unas pocas horas. Cuando se activa un gen, se produce una copia de ARN (ácido ribonucleico) del ADN. El ARN consta de cuatro componentes diferentes, a saber, las bases orgánicas adenina, citosina, guanina y uracilo. Éstas están dispuestas en una cadena y en una combinación que es característica del gen original. Esto sirve como plantilla para la producción de una proteína que realiza una función específica dentro de la célula.
scSLAM-seq clasifica el ARN en viejo y nuevo
Para distinguir entre el ARN que ya estaba presente antes de la infección viral y el nuevo ARN que se hizo después, los investigadores utilizaron un truco de etiquetado: añadieron una forma del uracilo, componente del ARN, que fue modificado químicamente en comparación con su variante natural, al medio de cultivo de las células al mismo tiempo que el virus infectante. Las células entonces incorporaron el uracil etiquetado en su ARN recién producido. Después de extraer el ARN de las células dos horas más tarde, el uracilo etiquetado se convirtió en otro componente del ARN, la citosina, a través de una reacción química. "La secuencia de ARN contiene citosina, mientras que el uracilo debería haber sido incorporado", explica Dölken. "La idea detrás de esto es que el ARN producido después de la infección ahora tiene una etiqueta que puede ser usada para identificarlo como'nuevo' durante la subsiguiente secuenciación de ARN." Utilizando un nuevo método bioinformático patentado, los investigadores investigaron el ARN de cada célula individual. De esta manera, fueron capaces de evaluar comúnmente la expresión de más de 4000 genes por célula y cuantificar con precisión las contribuciones relativas de los ARNs nuevos y antiguos. "Los datos que podemos recopilar usando scSLAM-seq son espectaculares", dice Erhard. "Para cada célula individual podemos determinar qué porcentaje de su ARN se produce de nuevo en un plazo de dos horas después de un desafío, en este caso, una infección viral. Por primera vez, esto permite el análisis dosis-respuesta a nivel de célula única". El equipo de investigación investigó el ARN de 100 células individuales en total. "Esto fue suficiente para obtener una visión completamente nueva de la activación de los genes celulares", dice Saliba. "Por primera vez, el scSLAM-seq nos permite analizar con precisión cómo reacciona una sola célula ante una alteración como una infección viral o bacteriana en un corto período de tiempo y qué genes se leen posteriormente con mayor o menor frecuencia, lo que nos permite comprender qué plan de batalla ha preparado la célula para la lucha contra un intruso".
Cada célula trabaja de forma diferente
Los científicos también pudieron demostrar que la producción de ARN a partir de un gen, llamado transcripción, no es continua, sino que tiene lugar en ráfagas: Durante la infección viral se activan cientos de genes que por lo demás son transcriptivamente silenciosos. En particular, esto incluye genes que ayudan a nuestras células a combatir la infección. "En nuestros análisis, muchos genes celulares exhibían un pronunciado comportamiento de ruptura. Dentro de una célula, casi todo el ARN de un gen era nuevo o antiguo", dice Erhard, "y pudimos demostrar que el comportamiento de la explosión depende de la estructura de ciertas regiones de la secuencia genética -los promotores- que controlan la transcripción de los genes". Por lo tanto, la transcripción no sólo es más fuerte o más débil en las células infectadas, sino que se produce según un principio de encendido/apagado. Saliba: "Esto también explica por qué las células tienen a menudo perfiles de ARN tan diferentes y por qué algunas células son capaces de luchar contra los virus inmediatamente después del primer contacto y por qué otras necesitan primero la ayuda de las células vecinas". Cada célula trabaja según su propio ritmo: Las células con perfiles de ARN inicialmente idénticos mostrarán ARN completamente diferentes después de sólo unos pocos días. Dölken: "Nuestras investigaciones nos permitieron obtener información innovadora sobre el momento de la activación de los genes celulares, lo que de hecho ha cambiado completamente mi comprensión de esta área."
Protección contra enfermedades autoinmunes
El principio de encendido/apagado de la activación de genes celulares probablemente tiene una función muy importante para nosotros: Si todos los genes utilizados para combatir los virus fueran producidos continuamente por cada célula del cuerpo, esto podría conducir a reacciones no deseadas y enfermedades autoinmunes. "Al encenderse en el momento adecuado, pero sólo cuando es absolutamente necesario, nuestro sistema inmunológico puede crear un entorno celular protector sin riesgo de reacciones falsas perjudiciales que puedan conducir a la autoinmunidad", dice Dölken. Por esta razón, ciertos mecanismos son completamente funcionales sólo en un pequeño número de células del cuerpo. Estas células "centinela" son capaces de detectar un virus invasor y combatirlo eficientemente, por ejemplo. Informan a las otras células, que a su vez ponen en marcha todo el arsenal de defensa y activan los genes adecuados para controlar la infección y evitar el peligro.
Con el método de análisis bioinformático patentado "GRAND-SLAM", necesario para la evaluación de los datos experimentales altamente complejos, los investigadores de Würzburg obtuvieron nuevos conocimientos sobre los procesos celulares ocultos. "Por primera vez, podemos ver realmente cómo reacciona una sola célula", dice Dölken. Puede utilizarse en investigaciones futuras para responder a una amplia gama de preguntas a nivel de célula única. "El scSLAM-seq es ideal para estudios de knock-out con el fin de descubrir qué genes juegan un papel clave en la lucha contra los patógenos o en el desarrollo de enfermedades", dice Saliba. "Es metodológicamente simple, la calidad de los datos es alta y es ideal para estudios de dosis-respuesta, también a lo largo del tiempo", por lo que scSLAM-seq y GRAND-SLAM tendrán implicaciones importantes en otros campos de las ciencias naturales como el cáncer y el desarrollo de fármacos.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.