Siguiente paso en la mejora de imágenes de microscopía óptica
La nueva arquitectura de aprendizaje profundo permite una mayor eficiencia en comparación con los métodos más utilizados
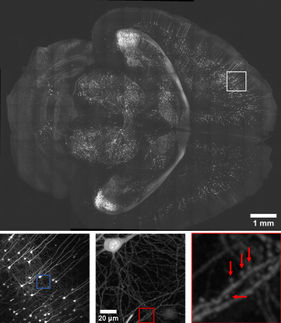
Es el procesamiento computacional de imágenes que revela los detalles más sutiles de una muestra colocada bajo todo tipo de microscopios ópticos diferentes. Aunque este procesamiento ha avanzado mucho, todavía hay margen para aumentar, por ejemplo, el contraste y la resolución de las imágenes. Basado en una arquitectura única de aprendizaje profundo, un nuevo modelo computacional desarrollado por investigadores del Center for Advanced Systems Understanding (CASUS) del Helmholtz-Zentrum Dresden-Rossendorf (HZDR) y el Max Delbrück Center for Molecular Medicine es más rápido que los modelos tradicionales, al tiempo que iguala o incluso supera la calidad de sus imágenes. El modelo, denominado Multi-Stage Residual-BCR Net (m-rBCR), se desarrolló específicamente para imágenes de microscopía. Presentado por primera vez en la conferencia bienal European Conference on Computer Vision (ECCV), el principal evento en el campo de la visión por ordenador y el aprendizaje automático, ya está disponible el correspondiente artículo de conferencia revisado por pares.
El nuevo modelo da un nuevo giro a una técnica de tratamiento de imágenes llamada deconvolución. Este método de cálculo intensivo mejora el contraste y la resolución de las imágenes digitales captadas con microscopios ópticos de campo amplio, confocales o de transmisión. La deconvolución pretende reducir el desenfoque, un cierto tipo de degradación de la imagen introducida por el sistema microscópico utilizado. Las dos estrategias principales son la deconvolución explícita y la deconvolución basada en el aprendizaje profundo.
Los enfoques de deconvolución explícita se basan en el concepto de función de dispersión de puntos (PSF). Una PSF describe básicamente cómo una fuente de luz puntual infinitamente pequeña que se origina en la muestra es ampliada y propagada en un patrón de difracción tridimensional por el sistema óptico. Es decir: En una imagen grabada (bidimensional) siempre hay algo de luz procedente de estructuras desenfocadas que producen el desenfoque. Si se conoce la PSF de un sistema microscópico, se puede calcular el desenfoque para obtener una imagen que se parezca mucho más a la realidad que la imagen grabada sin procesar.
"El gran problema de las técnicas de deconvolución basadas en la PSF es que a menudo la PSF de un sistema microscópico determinado no está disponible o es imprecisa", afirma el Dr. Artur Yakimovich, líder de un grupo de jóvenes investigadores de CASUS y autor correspondiente del artículo de ECCV. "Durante décadas se ha trabajado en la llamada deconvolución ciega, en la que la PSF se estima a partir de la imagen o del conjunto de imágenes. Sin embargo, la deconvolución ciega sigue siendo un problema muy difícil y los avances conseguidos son modestos."
Como ya demostró en el pasado el equipo de Yakimovich, el uso de la caja de herramientas de "resolución inversa de problemas" funciona bien en microscopía. Los problemas inversos tratan de recuperar los factores causales que conducen a determinadas observaciones. Normalmente, se necesitan muchos datos y algoritmos de aprendizaje profundo para abordar este tipo de problemas con éxito. Al igual que con los métodos de deconvolución explícita, los resultados son imágenes de mayor resolución o mejor calidad. Para el enfoque presentado en el ECCV, los científicos utilizaron una red neuronal informada por la física llamada Multi-Stage Residual-BCR Net (m-rBCR).
Aprendizaje profundo desplegado de forma diferente
En general, existen dos variantes básicas para el procesamiento de imágenes. Se puede partir de la representación espacial clásica de una imagen o de su representación en frecuencia (lo que requiere un paso de transformación a partir de la representación espacial). En esta última, cada imagen se representa como una colección de ondas. Ambas representaciones son valiosas. Algunas operaciones de procesamiento son más fáciles de realizar en una forma y otras en la otra. La gran mayoría de las arquitecturas de aprendizaje profundo operan en el dominio espacial . Es muy adecuado para las fotografías. Sin embargo, las imágenes de microscopía son diferentes. En su mayoría son monocromáticas. En el caso de técnicas como la microscopía de fluorescencia, se trata de fuentes de luz específicas sobre un fondo negro. De ahí que m-rBCR utilice la representación de frecuencias como punto de partida.
"Utilizar el dominio de la frecuencia en estos casos puede ayudar a crear representaciones de datos ópticamente significativas, una noción que permite a m-rBCR resolver la tarea de deconvolución con sorprendentemente pocos parámetros en comparación con otras arquitecturas de aprendizaje profundo actuales", explica Rui Li, primer autor y presentador en el ECCV. Li sugirió para avanzar la arquitectura de red neuronal de un modelo llamado BCR-Net que a su vez se inspiraba en un esquema de compresión de señales basado en la representación de frecuencias introducido en la década de 1990 por Gregory Beylkin, Ronald Coifman y Vladimir Rokhlin (lo que explica el nombre de la transformada BCR).
El equipo ha validado el modelo m-rBCR en cuatro conjuntos de datos diferentes, dos conjuntos de datos de imágenes de microscopía simuladas y dos conjuntos de datos de microscopía reales. Demuestra un alto rendimiento con un número significativamente menor de parámetros de entrenamiento y un tiempo de ejecución más corto en comparación con los últimos modelos basados en aprendizaje profundo y, por supuesto, también supera a los métodos de deconvolución explícita.
Un modelo adaptado a las imágenes de microscopía
"Esta nueva arquitectura está aprovechando una forma descuidada de aprender representaciones más allá de los enfoques clásicos de redes neuronales convolucionales", resume el coautor Prof. Misha Kudryashev, Líder del Grupo de "Biología Estructural In Situ" de Max-Delbrück-Centrum für Molekulare Medizin en Berlín. "Nuestro modelo reduce significativamente los parámetros potencialmente redundantes. Como muestran los resultados, esto no va acompañado de una pérdida de rendimiento. El modelo es explícitamente adecuado para imágenes de microscopía y, al tener una arquitectura ligera, está desafiando la tendencia de modelos cada vez más grandes que requieren cada vez más potencia de cálculo."
El grupo de Yakimovich ha publicado recientemente un modelo de mejora de la calidad de imagen basado en inteligencia artificial generativa. Este modelo de difusión variacional condicional produce resultados de última generación que también superan al modelo m-rBCR presentado aquí. "Sin embargo, se necesitan datos de entrenamiento y recursos computacionales que incluyan suficientes unidades de procesamiento gráfico, muy solicitadas hoy en día", recuerda Yakimovich. "El modelo ligero m-rBCR no tiene estas limitaciones y sigue ofreciendo muy buenos resultados. Por tanto, confío en que ganaremos adeptos en la comunidad de la imagen. Para facilitarlo, ya hemos empezado a mejorar la facilidad de uso."
El grupo de Yakimovich "Machine Learning for Infection and Disease" pretende comprender la compleja red de interacciones moleculares que se activa tras la infección del organismo por un patógeno. El uso de las nuevas posibilidades del aprendizaje automático es aquí clave. Las áreas de interés incluyen la mejora de la resolución de las imágenes, la reconstrucción de imágenes en 3D, el diagnóstico automatizado de enfermedades y la evaluación de la calidad de la reconstrucción de imágenes.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.