De miles de horas a unos minutos: el software acelera el desarrollo de fármacos
Los investigadores desarrollan un método novedoso para predecir en cuestión de minutos la morfología de las capas de azúcar de las proteínas de interés clínico
Los azúcares cubren casi todas las proteínas presentes en la superficie de las células de nuestro cuerpo, formando un escudo alrededor de las proteínas. Así, estos azúcares influyen en el modo en que las células interactúan con su entorno, incluidos los patógenos, desempeñando un importante papel en el desarrollo de fármacos médicos. GlycoSHIELD, un nuevo enfoque computacional para estudiar los escudos de azúcares de las proteínas, reduce los recursos, ahorra tiempo y es fácil de usar. Se desarrolló en el marco del Programa Dioscuri de la Sociedad Max Planck en un proyecto de cooperación polaco-alemán mediante colaboraciones internacionales con investigadores de Francia, Taiwán y Alemania.
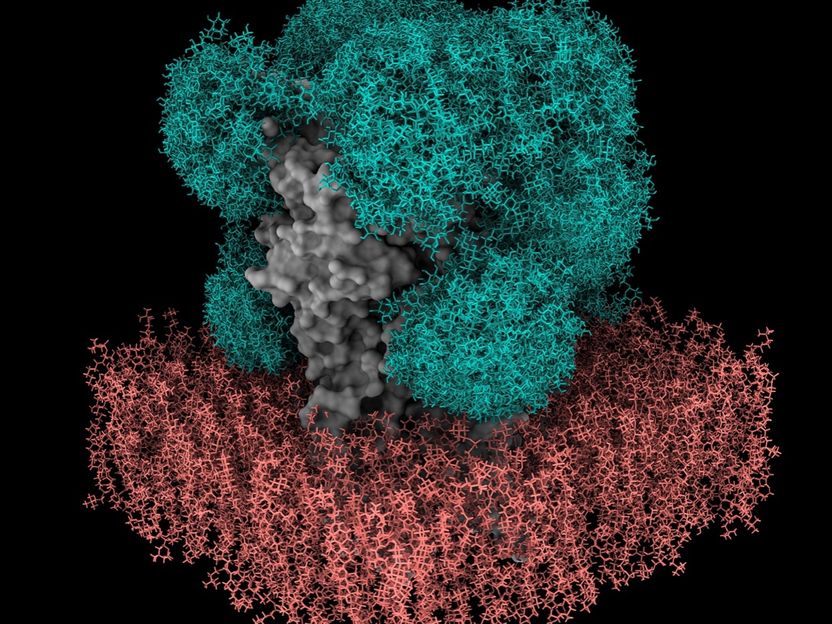
Modelo del escudo de azúcar (verde) en el receptor GABAA (gris) en una membrana (rojo) generado por GlycoSHIELD.
© Dr. Cyril Hanus, Inserm, University Paris-Cité.
Las proteínas no sólo desempeñan funciones críticas para la supervivencia de las células, sino que también influyen en el desarrollo y la progresión de las enfermedades. Para comprender su papel en la salud y la enfermedad, los investigadores estudian la estructura atómica tridimensional de las proteínas utilizando métodos tanto experimentales como computacionales. Más del 75% de las proteínas presentes en la superficie de nuestras células están recubiertas de glicanos. Estas moléculas similares al azúcar forman escudos protectores muy dinámicos alrededor de las proteínas. Sin embargo, la movilidad y variabilidad de los azúcares dificultan determinar cómo se comportan estos escudos o cómo influyen en la unión de moléculas de fármacos. Mateusz Sikora, director del proyecto y jefe del Centro Dioscuri de Modelización de Modificaciones Post-traduccionales, y su equipo de Cracovia, junto con sus socios del Instituto Max Planck de Biofísica de Fráncfort del Meno (Alemania), han abordado este reto utilizando ordenadores, en colaboración con científicos del Inserm de París, la Academia Sinica de Tapei y la Universidad de Bremen. Su nuevo y potente algoritmo GlycoSHIELD permite modelizar de forma rápida pero realista las cadenas de azúcar presentes en las superficies de las proteínas. Al reducir las horas de cálculo y, por tanto, el consumo de energía en varios órdenes de magnitud en comparación con las herramientas de simulación convencionales, GlycoSHIELD allana el camino hacia la informática verde. Los científicos publicaron su nuevo método y su validación en la revista Cell.
De miles de horas a unos minutos
Los escudos protectores de glicanos influyen enormemente en el modo en que las proteínas interactúan con otras moléculas, como los fármacos terapéuticos. Por ejemplo, la capa de azúcar sobre la proteína espiga del coronavirus oculta el virus al sistema inmunitario dificultando que los anticuerpos naturales o inducidos por vacunas reconozcan el virus. Por tanto, los escudos de azúcar desempeñan un papel importante en el desarrollo de fármacos y vacunas. La investigación farmacéutica podría beneficiarse de la predicción rutinaria de su morfología y dinámica. Hasta ahora, sin embargo, predecir la estructura de las capas de azúcar mediante simulaciones informáticas sólo era posible con conocimientos expertos en superordenadores especiales. En muchos casos, se necesitaban miles o incluso millones de horas de cálculo. Con GlycoSHIELD, el equipo de Sikora ofrece una alternativa rápida, ecológica y de código abierto. "Nuestro planteamiento reduce los recursos, el tiempo de cálculo y los conocimientos técnicos necesarios", afirma Sikora. "Cualquiera puede ahora calcular la disposición y la dinámica de las moléculas de azúcar sobre las proteínas en su ordenador personal en cuestión de minutos, sin necesidad de conocimientos expertos ni ordenadores de alto rendimiento. Además, esta nueva forma de hacer cálculos es muy eficiente desde el punto de vista energético". El software no sólo puede utilizarse en investigación, sino que también podría ser útil para el desarrollo de fármacos o vacunas, por ejemplo en inmunoterapia contra el cáncer.
Un rompecabezas hecho de azúcar
¿Cómo consiguió el equipo aumentar tanto la eficacia? Los autores crearon y analizaron una biblioteca de miles de poses tridimensionales más probables de las formas más comunes de cadenas de azúcar en proteínas encontradas en humanos y microorganismos. Mediante largas simulaciones y experimentos, descubrieron que para una predicción fiable de los escudos de glicanos basta con que los azúcares unidos no choquen con membranas o partes de la proteína. El algoritmo se basa en estos hallazgos. "Los usuarios de GlyoSHIELD sólo tienen que especificar la proteína y los lugares donde están unidos los azúcares. A continuación, nuestro software los coloca en la superficie de la proteína en la disposición más probable", explica Sikora. "Pudimos reproducir con precisión los escudos de azúcares de la proteína de la espiga: ¡tienen exactamente el mismo aspecto que lo que vemos en los experimentos!". Con GlycoSHIELD ahora es posible complementar con información sobre los azúcares tanto las estructuras de proteínas nuevas como las ya existentes. Los científicos también utilizaron GlycoSHIELD para revelar el patrón de los azúcares en el receptor GABAA, un objetivo importante para sedantes y anestésicos.
Un éxito para el Centro Dioscuri
Los Centros Dioscuri iniciados por la Sociedad Max Planck tienen por objeto contribuir a reforzar y ampliar la investigación de excelencia en Europa Central y Oriental. Desde mayo de 2023, Mateusz Sikora, antiguo investigador postdoctoral en el Instituto Max Planck de Biofísica, recibe apoyo financiero en el marco del programa financiado bilateralmente como director del Centro Dioscuri para la Modelización de Modificaciones Post-traduccionales creado en la Universidad Jagellónica de Cracovia, Polonia. Gerhard Hummer, Jefe del Departamento de Biofísica Teórica del Instituto Max Planck de Biofísica, le apoya como socio desde Alemania y también ha contribuido a este trabajo. Después de menos de un año, Sikora ya se ha apuntado un gran éxito con su algoritmo verde y está contribuyendo a promover Polonia como lugar de investigación atractivo y competitivo.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.
Publicación original
Yu-Xi Tsai, Ning-En Chang, Klaus Reuter, Hao-Ting Chang, Tzu-Jing Yang, Sören von Bülow, Vidhi Sehrawat, Noémie Zerrouki, Matthieu Tuffery, Michael Gecht, Isabell Louise Grothaus, Lucio Colombi Ciacchi, Yong-Sheng Wang, Min-Feng Hsu, Kay-Hooi Khoo, Gerhard Hummer, Shang-Te Danny Hsu, Cyril Hanus, Mateusz Sikora; "Rapid simulation of glycoprotein structures by grafting and steric exclusion of glycan conformer libraries"; Cell, Volume 187