ADN manipulado por ordenador para estudiar la identidad de las células
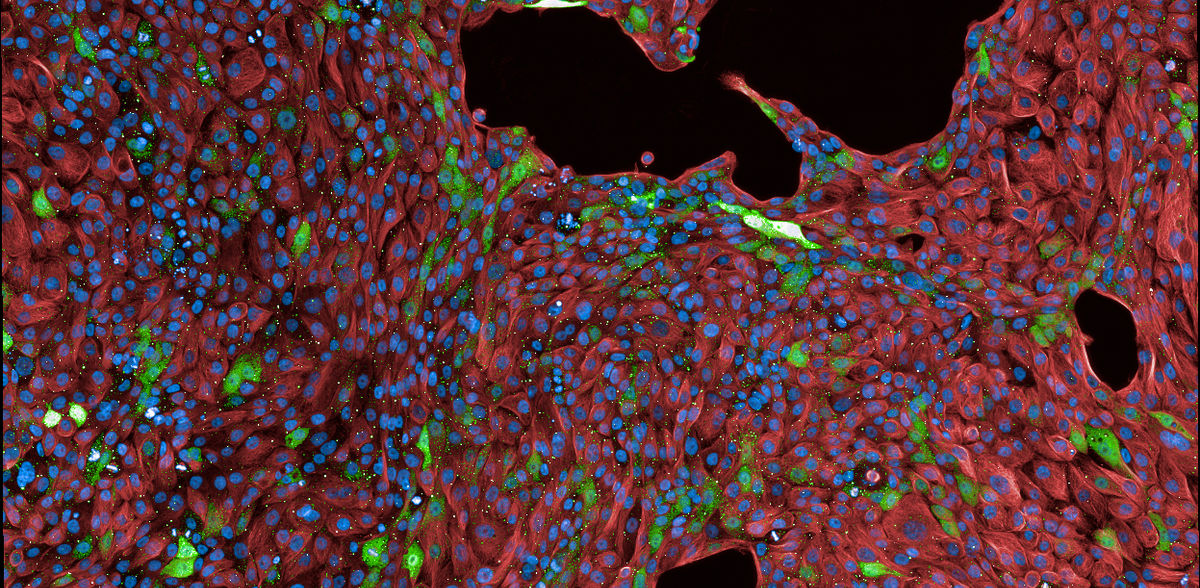
"Podemos seguir con la mirada el color en una placa de Petri cuando damos un tratamiento"
Un nuevo programa informático permite a los científicos diseñar segmentos sintéticos de ADN que indican, en tiempo real, el estado de las células. Según publica el laboratorio Gargiulo en "Nature Communications", servirá para buscar fármacos contra el cáncer o las infecciones víricas, o para mejorar las inmunoterapias génicas y celulares.
Todas las células de nuestro cuerpo tienen el mismo código genético, pero pueden diferir en su identidad, funciones y estados patológicos. Diferenciar una célula de otra de forma sencilla y en tiempo real sería de gran valor para los científicos que tratan de comprender la inflamación, las infecciones o los cánceres. Ahora, científicos del Centro Max Delbrück han creado un algoritmo capaz de diseñar herramientas de este tipo que revelan la identidad y el estado de las células mediante segmentos de ADN denominados "regiones sintéticas de control de locus" (sLCR). Pueden utilizarse en diversos sistemas biológicos. Los hallazgos, del laboratorio del Dr. Gaetano Gargiulo, jefe del Laboratorio de Oncología Molecular, se recogen en "Nature Communications".
"Este algoritmo nos permite crear herramientas de ADN precisas para marcar y estudiar células, ofreciendo nuevos conocimientos sobre los comportamientos celulares", afirma Gargiulo, autor principal del estudio. "Esperamos que esta investigación abra las puertas a una forma más sencilla y escalable de entender y manipular las células".
Este esfuerzo comenzó cuando el Dr. Carlos Company, antiguo estudiante de posgrado en el laboratorio de Gargiulo y coprimer autor del estudio, empezó a invertir energía en automatizar el diseño de las herramientas de ADN y ponerlas al alcance de otros científicos. Codificó un algoritmo capaz de generar herramientas para comprender procesos celulares básicos y procesos patológicos como el cáncer, la inflamación y las infecciones.
"Esta herramienta permite a los investigadores examinar el modo en que las células se transforman de un tipo a otro. Es especialmente innovadora porque recopila todas las instrucciones cruciales que dirigen estos cambios en una sencilla secuencia sintética de ADN. A su vez, esto simplifica el estudio de comportamientos celulares complejos en áreas importantes como la investigación del cáncer y el desarrollo humano", afirma Company.
Algoritmo para fabricar una herramienta de ADN a medida
El programa informático se denomina "diseño lógico de ADN cis-regulador sintético" (LSD). Los investigadores introducen los genes conocidos y los factores de transcripción asociados a los estados celulares específicos que quieren estudiar, y el programa los utiliza para identificar segmentos de ADN (promotores y potenciadores) que controlan la actividad en la célula de interés. Esta información es suficiente para descubrir secuencias funcionales, y los científicos no tienen que conocer la razón genética o molecular precisa del comportamiento de una célula; sólo tienen que construir el sLCR.
Según Yuliia Dramaretska, estudiante de posgrado del laboratorio de Gargiulo y coautora del estudio, el programa busca en los genomas de humanos o ratones los lugares donde es más probable que se unan los factores de transcripción. El programa escupe una lista de secuencias de 150 pares de bases que son relevantes y que probablemente actúan como promotores y potenciadores activos de la enfermedad estudiada.
"Obviamente, no ofrece una lista aleatoria de esas regiones", afirma. "En realidad, el algoritmo las clasifica y encuentra los segmentos que representarán con mayor eficacia el fenotipo que se quiere estudiar".
Como una lámpara dentro de las células
A continuación, los científicos pueden fabricar una herramienta, denominada "región sintética de control de locus" (sLCR), que incluye la secuencia generada seguida de un segmento de ADN que codifica una proteína fluorescente. "Las sLCR son como una lámpara automatizada que se puede poner dentro de las células. Esta lámpara se enciende sólo en las condiciones que se quieren estudiar", explica la Dra. Michela Serresi, investigadora del laboratorio Gargiulo y coautora. El color de la "lámpara" puede variar para adaptarse a los distintos estados de interés, de modo que los científicos pueden mirar con un microscopio de fluorescencia y conocer inmediatamente el estado de cada célula a partir de su color. "Podemos seguir con la vista el color en una placa de Petri cuando administramos un tratamiento", afirma Serresi.
Los científicos han validado la utilidad del programa informático utilizándolo para buscar fármacos en células infectadas por el SARS-CoV-2, como se publicó el año pasado en "Science Advances". También lo utilizaron para encontrar mecanismos implicados en los cánceres cerebrales llamados glioblastomas, en los que ningún tratamiento funciona por sí solo. "Para hallar combinaciones de tratamientos que funcionen en estados celulares específicos de los glioblastomas, no sólo hay que entender qué define esos estados celulares, sino también ver cómo surgen", explica el Dr. Matthias Jürgen Schmitt, investigador del laboratorio de Gargiulo y coprimer autor, que utilizó las herramientas en el laboratorio para demostrar su valor.
Imaginemos unas células inmunitarias diseñadas en el laboratorio como terapia génica para eliminar un tipo de cáncer. Cuando se infunden en el paciente, no todas estas células funcionarán según lo previsto. Algunas serán potentes y otras estarán en un estado disfuncional. Financiado por una beca del Consejo Europeo de Investigación, el laboratorio de Gargiulo utilizará este sistema para estudiar el comportamiento de estas delicadas terapias celulares contra el cáncer durante su fabricación. "Con las colaboraciones adecuadas, este método tiene potencial para hacer avanzar tratamientos en áreas como el cáncer, las infecciones víricas y las inmunoterapias", afirma Gargiulo.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.
Publicación original
Carlos Company, Matthias Jürgen Schmitt, Yuliia Dramaretska, Michela Serresi, Sonia Kertalli, Ben Jiang, Jiang-An Yin, Adriano Aguzzi, Iros Barozzi, Gaetano Gargiulo; "Logical design of synthetic cis-regulatory DNA for genetic tracing of cell identities and state changes"; Nature Communications, Volume 15, 2024-2-5