Una nueva herramienta integra el análisis de la secuenciación genética del microbioma y del huésped
Una nueva herramienta informática facilita el estudio de las relaciones entre un huésped, su microbioma y patógenos como el VIH o el SARS-CoV-2
Investigadores del Instituto de Investigación Biomédica de Texas y de la Universidad de Tulane han desarrollado una nueva herramienta informática que facilita, acelera y rentabiliza el análisis de la información genética de un huésped y su microbioma al mismo tiempo. El software, denominado "detector del metatranscriptoma" (MTD), puede ser utilizado por un amplio abanico de microbiólogos y desarrolladores de fármacos, incluidos los que investigan enfermedades como ciertos tipos de cáncer, el COVID-19, el VIH/SIDA, la malaria y muchas otras afecciones de la salud humana relacionadas con los microorganismos. La herramienta se ha publicado recientemente en la revista Briefings in Bioinformatics.
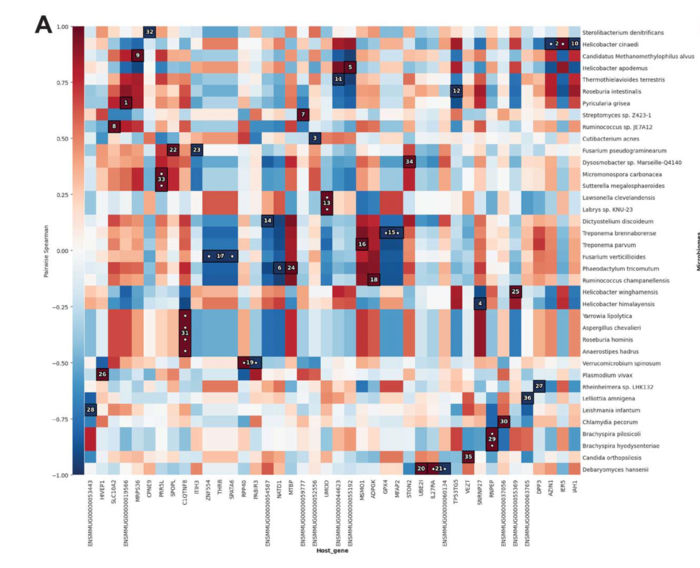
El MTD analiza la actividad de la expresión génica tanto en el huésped como en el microbioma al mismo tiempo, y genera automáticamente un gráfico que muestra las correlaciones entre ambos. El rojo más oscuro muestra una correlación positiva más fuerte, lo que significa que la regulación ascendente o descendente de un determinado gen o vía del huésped está relacionada con la regulación ascendente o descendente de un determinado tipo de microbio en la misma dirección. El azul más oscuro muestra una correlación negativa más fuerte, lo que significa que un gen o vía del huésped es menos activo pero la especie de microbio es más activa. Aunque el análisis no indica que una causa sea la otra, puede poner de relieve relaciones que los investigadores pueden investigar más a fondo y que podrían ayudar a comprender la relación causa-efecto para el posible desarrollo de tratamientos.
Texas Biomed
"Es muy fácil de usar, especialmente para los investigadores con poca o ninguna experiencia en bioinformática", afirma la profesora asociada Binhua "Julie" Ling, doctora que codirige el Programa de Investigación de Interacciones Anfitrión-Patógeno de Texas Biomed y es la autora principal del artículo. "Sólo hay que escribir una línea de código para establecer ciertos parámetros y el software hace el resto automáticamente".
El MTD permite a los investigadores obtener una instantánea completa de los microbios presentes en el huésped, incluida la enorme variedad de "bacterias buenas" que normalmente viven en el interior de las personas y los animales, así como las dañinas, como los virus que causan enfermedades graves. Y lo que es más importante, la MTD analiza la actividad de la expresión génica, es decir, qué genes se activan o desactivan, tanto en los microbios como en el huésped de forma simultánea, lo que permite a los investigadores detectar fácilmente las relaciones entre ellos. Por ejemplo, ver qué genes están activos en un microbio y en el huésped podría indicar que la actividad de uno está influenciada por el otro, y podría sugerir un objetivo potencial para el tratamiento más adelante, explica Ling.
"No podemos decir en este momento si es causa y efecto, pero podemos usar este análisis para señalar qué genes o vías deberíamos investigar, quizás algunos que nunca antes habíamos considerado como relacionados", dice Ling. "MTD puede ayudar a acelerar ese proceso y abrir potencialmente nuevas vías de investigación y desarrollo de fármacos".
MTD enlaza varios paquetes de software existentes y extrae de bases de datos internacionales que contienen secuencias de ARN de más de 100.000 especies de bacterias, virus, hongos, arqueas y protozoos, así como secuencias de vectores y plásmidos. Los usuarios también pueden actualizar la base de datos con las secuencias específicas que les interesen.
La primera autora del artículo, la doctora Fei Wu, trabajó con Ling para estudiar cómo cambia el microbioma con la edad en monos jóvenes y mayores con el virus de la inmunodeficiencia de los simios (VIS), la versión en mono del VIH.
"Teníamos que analizar la expresión génica del huésped mediante un flujo de trabajo, y la expresión génica del microbioma mediante otro flujo de trabajo", dice Wu. "Nos preguntamos por qué no podemos hacer ambas cosas al mismo tiempo".
Como su laboratorio se trasladó durante la pandemia de COVID-19, tuvieron tiempo para centrarse en el trabajo informático y se propusieron resolver esta cuestión. Wu trabajó con Ling y su colaborador Yao-Zhong Liu, doctor y profesor asociado de la Facultad de Salud Pública y Medicina Tropical de la Universidad de Tulane, para crear y probar el nuevo software.
"Normalmente, utilizamos el software bioinformático para analizar nuestros datos, no para construirlos", dice Wu. "Fue un reto, pero emocionante, ramificarse y tener ahora algo que no sólo nos ayudará a nosotros, sino también a cualquier otro investigador que realice la secuenciación de ARN de huéspedes y microbiomas; desde humanos y monos, hasta mosquitos portadores de parásitos de la malaria y caracoles portadores de parásitos de esquistosomas".
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.