ADNzimas: cómo funcionan las moléculas de ADN activas con potencial terapéutico
Las ADNzimas son biocatalizadores de precisión que destruyen moléculas de ARN no deseadas. Sin embargo, siguen existiendo grandes obstáculos para su uso en medicina. Junto con el Centro de Investigación de Jülich (FZJ) y la Universidad de Bonn, un equipo de investigación de la Universidad Heinrich Heine de Düsseldorf (HHU) ha investigado con resolución atómica cómo funcionan las ADNzimas en tiempo real. Ahora han presentado estos importantes hallazgos fundamentales y su aplicación en la revista "Nature".
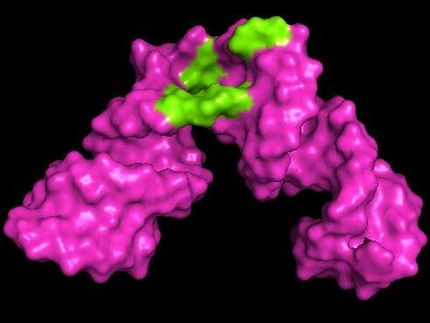
Una enzima de ADN (rojo) utiliza sus brazos de unión para acoplarse a un lugar específico de una cadena de ARN (amarillo) y luego la escinde en su núcleo. Para identificar la estructura y los mecanismos catalíticos de la ADNzima se utilizaron la RMN de alta resolución, la resonancia paramagnética de electrones y la espectroscopia de fluorescencia, así como simulaciones de dinámica molecular.
HHU / Manuel Etzkorn
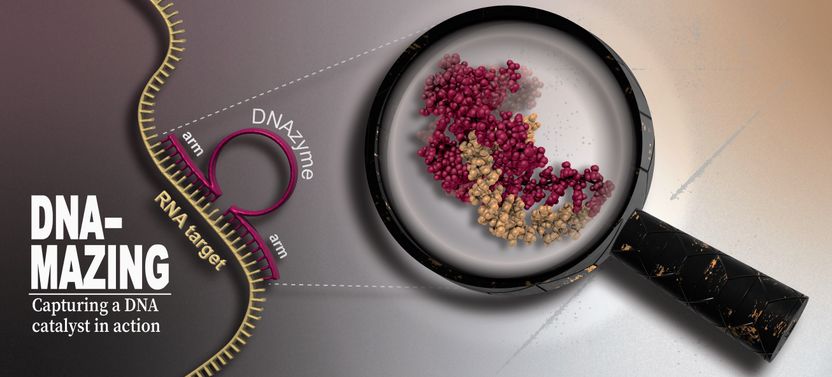
Las ADNzimas -palabra formada por ADN y enzima- son secuencias de ADN catalíticamente activas. Constan de un núcleo catalítico compuesto por unos 15 ácidos nucleicos flanqueados por brazos de unión cortos en los lados derecho e izquierdo, cada uno con unos diez ácidos nucleicos. Mientras que la secuencia del núcleo es fija, los brazos de unión pueden modificarse para adaptarse específicamente a prácticamente cualquier secuencia de ARN.
El objetivo es dirigirse a las moléculas de ARN no deseadas de los virus, el cáncer o las células nerviosas dañadas, utilizando las ADNzimas para atacarlas y destruirlas. Esto se consigue mediante secuencias de unión que coinciden con una secuencia de nucleótidos en la molécula de ARN objetivo. La DNAzima se acopla con precisión a la posición correspondiente y el núcleo escinde la molécula de ARN, cuyos fragmentos se degradan rápidamente en la célula. Los brazos de unión pueden intercambiarse rápida y fácilmente.
Las ventajas terapéuticas son evidentes: el ARN no deseado puede ser destruido con precisión, mientras que otras cadenas de ARN útiles en una célula permanecen intactas. En algunos virus como el SARS-CoV2 y el Ébola, el material genético está codificado en una molécula de ARN. Al igual que las células sanas, las células cancerosas utilizan el llamado ARN mensajero (ARNm) para copiar los planos de las proteínas de su ADN y transferirlos a las fábricas de moléculas. La secuencia de ARNm de las células cancerosas suele ser ligeramente diferente a la de las células sanas o estar presente en cantidades diferentes, lo que significa que las ADNzimas pueden atacar específicamente a las células cancerosas y evitar otras.
"Lo que en teoría suena muy bien y ya se propuso hace 20 años, desgraciadamente no funciona así en la práctica médica", dice el Dr. Manuel Etzkorn, jefe del grupo de trabajo del Instituto de Biología Física de la HHU y último autor del estudio, que se ha publicado ahora en Nature. "En un tubo de ensayo, las ADNzimas son muy eficaces para destruir las moléculas de ARN, pero esto rara vez ocurre en una célula. Debe haber un proceso competitivo que bloquee las DNAzimas. Sin embargo, sin una comprensión fundamental de su funcionamiento, es muy difícil desarrollar variantes mejoradas de las DNAzimas que puedan cumplir su labor en las células. Nuestros conocimientos han puesto en movimiento esta situación de bloqueo".
En su estudio, los autores de la HHU y un equipo del Centro de Investigación de Jülich (FZJ), la Universidad de Bonn y una empresa suiza trataron de entender cómo funciona dinámicamente el sistema en su conjunto, qué pasos se dan en el proceso de unión y escisión y qué cofactores apoyan la reacción.
Los investigadores observaron los procesos con resolución atómica y, en parte, en tiempo real mediante espectroscopia de resonancia magnética nuclear (RMN) de alta resolución. Esto les permitió representar la disposición atómica tridimensional que adopta la ADNzima para unirse al ARN y escindirlo: El núcleo envuelve la cadena de ARN de forma muy eficaz, dividiéndola en dos partes en varios pasos intermedios. Después de escindirla, la ADNzima libera los fragmentos y puede volver a unirse a otra parte.
El Dr. Holger Gohlke, de la Cátedra de Química Farmacéutica y Medicinal de la HHU y del Instituto de Bio y Geociencias de la FZJ, cuyo equipo realizó simulaciones de dinámica molecular del complejo ADN-zima/ARN, añade: "En el mejor sentido de la modelización integradora, pudimos proponer un mecanismo plausible de escisión del ARN a nivel atómico y suministrar información sobre la preferencia de bases del ARN en el lugar de escisión".
Jan Borggräfe, investigador doctoral del grupo de trabajo de Etzkorn y autor principal del estudio, explica por qué las ADNzimas no funcionan bien en las células: "Establecimos que el magnesio, como cofactor clave, desempeña varias funciones esenciales en el mecanismo, pero que se une relativamente poco y sólo brevemente a la DNAzima. Hay otros componentes en la célula con mayor afinidad por el magnesio que "roban" el magnesio a la DNAzima, por así decirlo".
El siguiente paso es realizar investigaciones estructurales en cultivos celulares y organoides. El objetivo de las aplicaciones terapéuticas es mejorar la afinidad por el magnesio de las DNAzimas mediante modificaciones específicas para aumentar su actividad en los tejidos biológicos.
El Dr. Etzkorn señala otro campo de aplicación: "Nuestro Instituto se centra en la investigación de las enfermedades neurodegenerativas, donde también vemos un buen potencial para las DNAzimas. En el caso de la enfermedad de Parkinson, en determinadas circunstancias pueden ser capaces de destruir la secuencia de ARNm que impulsa la producción de alfa-sinucleína que, en grandes cantidades, puede promover procesos neurotóxicos". Las ADNzimas también podrían dar lugar a una nueva clase de antibióticos.
El Dr. Dieter Willbold, Director del Instituto de Biología Física de la HHU y del Instituto de Bioquímica Estructural de la FZJ, añade: "El estudio es un ejemplo más de cómo la investigación básica en biología estructural puede aportar contribuciones esenciales a avances biomédicos revolucionarios". El nuevo buque insignia del centro de RMN biomolecular, un dispositivo de RMN de 1,2 GHz, ya ha contribuido a este éxito". El dispositivo del Centro de RMN Biomolecular, gestionado conjuntamente por la HHU y la FZJ, es uno de los sistemas más potentes del mundo y proporciona una visión única de la estructura y el funcionamiento de los componentes básicos de la vida.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.
Publicación original
Jan Borggräfe, Julian Victor, Hannah Rosenbach, Aldino Viegas, Christoph G.W. Gertzen, Christine Wuebben, Helena Kovacs, Mohanraj Gopalswamy, Detlev Riesner, Gerhard Steger, Olav Schiemann, Holger Gohlke, Ingrid Span, Manuel Etzkorn; "Time-resolved structural analysis of an RNA-cleaving DNA catalyst"; Nature; 2021