La fuente de luz de rayos X del DESY identifica candidatos prometedores para los fármacos COVID
Las sustancias activas existentes se unen a la proteína clave del virus
Un equipo de investigadores ha identificado varios candidatos a fármacos contra el coronavirus SARS-CoV-2 en la fuente de luz de rayos X de alto brillo PETRA III del DESY. Se unen a una importante proteína del virus y, por tanto, podrían ser la base de un fármaco contra el Covid-19. En el llamado cribado de rayos X, los investigadores, bajo la dirección del DESY, probaron en poco tiempo casi 6000 sustancias activas conocidas que ya existen para el tratamiento de otras enfermedades. Tras medir unas 7.000 muestras, el equipo pudo identificar un total de 37 sustancias que se unen a la proteasa principal (Mpro) del virus del SARS-CoV-2, según informan los científicos en la revista Science. Siete de estas sustancias inhiben la actividad de la proteína y, por tanto, frenan la multiplicación del virus. Dos de ellas son tan prometedoras que se están investigando en estudios preclínicos. Este cribado de fármacos -probablemente el mayor de su clase- también reveló un nuevo sitio de unión en la principal proteasa del virus al que pueden acoplarse los fármacos.
En un cribado masivo de rayos X, los científicos identificaron candidatos prometedores a fármacos contra el SARS-CoV-2 (impresión artística).
DESY, Science Communication Lab
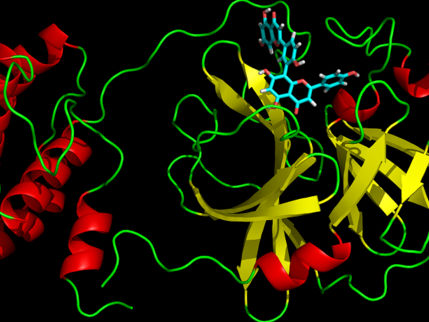
A diferencia de las vacunas, que ayudan a las personas sanas a defenderse del virus, la investigación farmacológica busca fármacos que ralenticen o detengan la reproducción del virus en el organismo de las personas ya infectadas. Los virus no pueden reproducirse por sí mismos. En su lugar, introducen su propio material genético en las células de su huésped y las hacen producir nuevos virus. Las proteínas, como la principal proteasa del virus, desempeñan un papel importante en este proceso. La proteasa corta las cadenas de proteínas producidas por la célula huésped según el plano del material genético del virus en partes más pequeñas que son necesarias para la reproducción del virus. Si se bloquea la proteasa principal, es posible que se interrumpa el ciclo; el virus ya no puede reproducirse y la infección es derrotada.
La línea de luz P11 de la fuente de luz de investigación PETRA III del DESY está especializada en estudios de biología estructural. Aquí, la estructura tridimensional de las proteínas puede visualizarse con precisión atómica. El equipo de investigación dirigido por el físico del DESY Alke Meents utilizó esta capacidad especial para examinar varios miles de sustancias activas de una biblioteca del Instituto Fraunhofer de Medicina Traslacional y Farmacología y de otra biblioteca de la empresa italiana Dompé Farmaceutici SpA para ver si se "acoplan" a la proteasa principal y cómo lo hacen, el primer paso importante para bloquearla. Como una llave en una cerradura, la molécula del fármaco encaja en un centro de unión de la proteasa. La ventaja de la biblioteca de fármacos es que contiene sustancias activas que ya han sido aprobadas para el tratamiento de seres humanos o las que están actualmente en diversas fases de prueba. De este modo, los candidatos adecuados para combatir el SARS-CoV-2 podrían utilizarse en ensayos clínicos con mucha más rapidez, ahorrando meses o años de desarrollo de fármacos.
El equipo técnico especial de la estación P11 de PETRA III incluye cambios de muestras totalmente automatizados con un brazo robótico, de modo que cada una de las más de 7.000 mediciones sólo requirió unos tres minutos. Con la ayuda del análisis de datos automatizado, el equipo pudo separar rápidamente el grano de la paja. "Utilizando un método de alto rendimiento, pudimos encontrar un total de 37 sustancias activas que se unen a la proteasa principal", dice Meents, que inició los experimentos.
En el siguiente paso, los investigadores del Instituto de Medicina Tropical Bernhard Nocht investigaron si estas sustancias activas inhiben o incluso impiden la replicación del virus en cultivos celulares y su compatibilidad con las células huésped. Esto redujo el número de sustancias activas adecuadas a siete, de las cuales dos destacaron especialmente. "Las sustancias activas Calpeptin y Pelitinib mostraron claramente la mayor antiviralidad con una buena compatibilidad celular. Por ello, nuestros socios de cooperación ya han iniciado investigaciones preclínicas con estas dos sustancias", explica el investigador del DESY Sebastian Günther, primer autor de la publicación de Science.
En su cribado de fármacos mediante cristalografía de proteínas, los investigadores no examinaron fragmentos de fármacos potenciales, como es habitual, sino moléculas completas del fármaco. En el proceso, sin embargo, el equipo de más de 100 científicos también descubrió algo completamente inesperado: encontraron un sitio de unión en la proteasa principal que había sido completamente desconocido hasta entonces. "No sólo fue una grata sorpresa que pudiéramos descubrir un nuevo sitio de unión a fármacos en la proteasa principal -un resultado que realmente sólo puede lograrse en una fuente de luz de sincrotrón como PETRA III-, sino que incluso uno de los dos prometedores candidatos a fármacos se une precisamente a este sitio", afirma Christian Betzel, del grupo de excelencia CUI de la Universidad de Hamburgo, co-iniciador del estudio.
"Un punto fuerte de nuestro método de cribado por rayos X, en comparación con otros métodos de cribado, es que obtenemos como resultado la estructura tridimensional de los complejos proteína-fármaco y podemos así identificar la unión de los fármacos a la proteína a nivel atómico. Aunque los dos candidatos más prometedores no lleguen a los ensayos clínicos, las 37 sustancias que se unen a la proteasa principal forman una valiosa base de datos para el desarrollo de fármacos basados en ellas", explica Patrick Reinke, investigador del DESY y coautor de la publicación.
"Las investigaciones realizadas en PETRA III demuestran de forma impresionante la relevancia de las fuentes de luz de sincrotrón de alto brillo para el desarrollo de futuros medicamentos y para la investigación sanitaria en su conjunto", subraya Helmut Dosch, Presidente de la Dirección del DESY. "Debemos y queremos ampliar aún más nuestras infraestructuras en el futuro para hacer frente a crisis sanitarias como la actual".
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.