Se revela la estructura de la nano turbina de proteínas
Los científicos determinan la primera estructura del motor rotativo de una célula utilizando microscopía de última generación
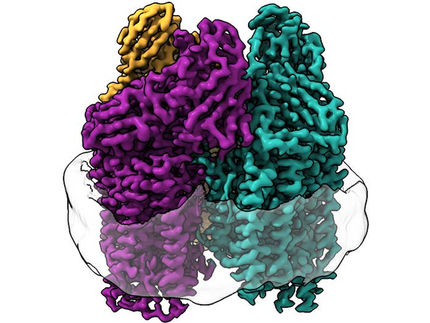
Las células dependen de complejos proteicos conocidos como sintasas de ATP o ATPasas para sus necesidades energéticas - las moléculas de trifosfato de adenosina (ATP) impulsan la mayoría de los procesos que sostienen la vida. El biólogo estructural Leonid Sazanov y su grupo de investigación del Instituto de Ciencia y Tecnología de Austria (IST Austria) en Klosterneuburg, Austria, han determinado la primera estructura atómica del representante de la familia V/A-ATPase, llenando el vacío en el árbol evolutivo de estas máquinas moleculares esenciales. Estos resultados obtenidos utilizando los últimos métodos de microscopía crioelectrónica revelaron una estructura de la enzima similar a la de una turbina o molino de agua.
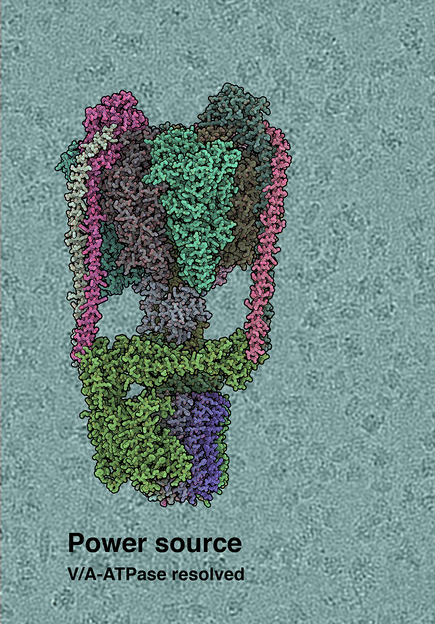
Estructura criogénica de la T. thermophilus V/A-tipo ATP sintasa. El fondo muestra la micrografía crio-EM en bruto, con moléculas individuales de ATPasa visibles.
(c) by IST Austria, 2019
Poder rotatorio
Las ATP sintasas/ATPases son grandes complejos de proteínas de membrana que comparten planes generales de construcción y mecanismos de catálisis rotativa. Esta familia de proteínas incluye la enzima de tipo F que se encuentra en las mitocondrias (fábricas de energía de la célula), cloroplastos (orgánulos que conducen la fotosíntesis en las plantas) y bacterias; el tipo V (vacuolar) que se encuentra en los compartimentos intracelulares de los eucariontes (organismos superiores con un núcleo) y el tipo A (arqueológico) que se encuentra en los procariotas - archaea (microorganismos antiguos) y algunas bacterias. Las proteínas consisten en un dominio globular F1/V1/A1 que sintetiza/hidroliza ATP y un dominio Fo/Vo/Ao embebido en membrana que transloca protones. Las enzimas de tipo F están bien estudiadas y sus estructuras atómicas son conocidas. Los dominios F1 y Fo están conectados por el tallo central o el eje/rotor, que gira dentro de la síntesis ATP de conducción F1. El rotor está unido al llamado anillo en C que es impulsado por el flujo de protones entre el anillo y el estator embebido en membrana. La rotación de la propia F1 se ve impedida por el tallo periférico único, que mantiene la F1 anclada al estator. La arquitectura general de esta máquina molecular única se asemeja a la de una turbina o un molino de agua, impulsado por el flujo de protones en lugar de agua.
Diferentes sabores de ATPasa
Las enzimas de tipo F y A generalmente funcionan para producir ATP, impulsadas por el flujo de protones a través de la membrana. Las enzimas de tipo V usualmente trabajan al revés, usando ATP para bombear protones. Los ATPasos V y A son similares estructuralmente pero se diferencian del tipo F por tener dos o tres tallos periféricos y subunidades adicionales de proteína de conexión entre V1 y Vo. Las enzimas de tipo V probablemente evolucionaron del tipo A y debido a estas similitudes el tipo A también se denomina V/A-ATPasa. Algunas bacterias, incluyendo Thermus thermophilus, adquirieron una enzima tipo A. Long Zhou, postdoctorado en el grupo de investigación Sazanov de IST Austria, ha purificado y estudiado esta enzima (ThV1Vo) por cryo-EM. En contraste con el tipo F, para las ATPasasas de tipo V sólo se determinaron previamente las estructuras de los dominios V1 y Vo aislados. Por lo tanto, no se sabía cómo está acoplado V1 a Vo, y se carecía del conocimiento sobre el ciclo catalítico completo.
Plasticidad y competencia
Los científicos determinaron no una, sino un total de cinco estructuras de toda la enzima ThV1Vo, utilizando métodos de microscopía crioelectrónica desarrollados recientemente en la llamada "revolución de resolución" de esta técnica. Las estructuras representan varios estados conformacionales de la enzima que difieren por la posición del rotor dentro del estator. La plasticidad conformacional global del ThV1Vo se revela como un sustancial bamboleo de V1 en el espacio en transición de un estado a otro. Es el resultado de la competencia mecánica entre la rotación del rotor central doblado y la rigidez del estator. El acoplamiento V1-Vo se consigue mediante una estrecha correspondencia estructural y electrostática entre el eje y la subunidad específica del tipo V que lo une al anillo en C. La visualización del trayecto protónico reveló diferencias significativas en la distribución de los residuos de proteína cargada con respecto a la de las F-ATPases, con un "punto de control" más estricto que impide el "deslizamiento" de la enzima.
¿Por qué complejidad adicional?
En lugar de un solo tallo periférico de enzimas de tipo F, los tipos A como el ThV1Vo tienen dos tallo periférico, mientras que los tipos V eucariotas tienen tres. Pero, ¿cuál es la ventaja de la complejidad adicional en el ya muy grande ensamblaje de proteínas, junto con subunidades adicionales que unen V1 y Vo? El dominio F1/V1 tiene una simetría triple, por lo que se produce (o consume) una molécula de ATP por cada rotación de 120° del estator dentro de F1/V1. El profesor Leonid Sazanov dice: "En las fases V/A-ATP, este paso es una rotación única de 120°, en contraste con la sintetasa F-ATP, donde se divide en varias subetapas. Por lo tanto, puede ser necesaria una mayor plasticidad en el ThV1Vo para vincular estos pasos de 120° en V1 a pasos de subunidad más pequeños por unidad c en el anillo Vo c12. Esta flexibilidad adicional puede conseguirse en los tipos en V gracias a los tallos periféricos adicionales y a las subunidades de conexión. Nuestras nuevas estructuras muestran cómo se logra esto, proporcionando un marco para toda la familia V-ATPase".
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.
Publicación original
Zhou, L. and Sazanov, L.A.; "Structure and Conformational Plasticity of the Intact Thermus thermophilus V/A-type ATPase"; Science; 2019, in press.