Neutronen machen Proteinbewegungen sichtbar
Jülicher Physiker entwickeln Methode zur Beobachtung von Proteinbewegung
Physiker des Forschungszentrums Jülich haben eine neue Methode entwickelt, die erstmals großräumige innere Bewegungen von Proteinen in Raum und Zeit bestimmen kann. Anders als bekannte Methoden ermöglicht diese Methode die direkte Messung nicht nur der Geschwindigkeit von inneren Bewegungen, sondern auch der räumlichen Ausdehnung und der dabei auftretenden inneren Kräfte.

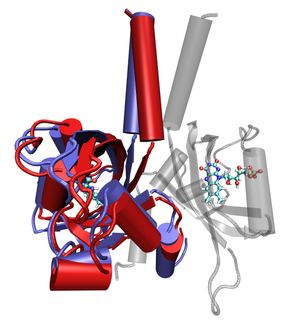
Beim Hefeprotein Alkoholdehydrogenase befindet sich das aktive Zentrum in einem Spalt, der die beiden Hauptdomänen trennt. In jedem Spalt der vier Monomere (z. B. in blau) sitzt ein Zinkatom (grün), welches als Katalysator bei der Umwandlung von Acetaldehyd in Ethanol (kleines Molekül) oder umgekehrt zur Entgiftung dient. Um diese Stoffe und den Cofaktor Nicotinamid-Adenin-Dinukleotid (NAD, großes Molekül vor dem Spalt) zum aktiven Zentrum zu lassen, muss die äußere Domäne großräumige Bewegungen machen, die den Spalt öffnet und wieder schließt.
Forschungszentrum Jülich
Proteine sind die molekularen "Maschinen" des Lebens. Als "Motoren" des Stoffwechsels sind sie in jeder Zelle unseres Körpers unermüdlich aktiv, transportieren, synthetisieren, spalten und wandeln Stoffe um. Fehlfunktionen von Proteinen können schwerwiegende gesundheitliche Folgen haben. Das ist nur einer der Gründe dafür, dass die "Nanomaschinen" im Zentrum des Interesses vieler Wissenschaftler stehen. Auch ihre Eignung als Biokatalysatoren für die Produktion von Rohstoffen, etwa für die Lebensmittelindustrie, macht sie für Industrie und Forschung interessant. Nachdem in den vergangenen Jahren die Zusammensetzung vieler Proteine, d.h. die Reihenfolge ihrer Aminosäurebausteine, aufgeklärt wurde, konzentrieren sich die Bemühungen vieler Forscher nunmehr darauf, ihre Funktionsweise genauer zu verstehen. Die bloße Reihenfolge der Bausteine sagt nämlich wenig aus; die dreidimensionale Struktur ist viel mehr entscheidend. Und diese ist nicht etwa starr, sondern passt sich dynamisch den Erfordernissen an. So katalysieren viele Proteine Umwandlungsprozesse in charakteristisch geformte Taschen, in die nur bestimmte Stoffe wie ein Schlüssel ins Schloss passen. Manchmal sorgt erst die Bindung so genannter Cofaktoren dafür, dass sich die richtige Form ausbildet.
Das ist bisher vor allem durch Strukturuntersuchungen von kristallisierten Proteinen nachgewiesen worden. Solche Untersuchungen stellen gewissermaßen "Schnappschüsse" dar. Direkt beobachteten Forscher innere Bewegungen, indem sie jeweils zwei Punkte eines Proteins mit molekularbiologischen Methoden fluoreszierend markierten. So lassen sich zwar Abstandsänderungen verfolgen, aber nur mit großem Aufwand dreidimensionale Aussagen erzielen. Mangels weiterer geeigneter experimenteller Methoden gründeten viele Erkenntnisse bisher auf Computersimulationen.
Physiker des Jülicher Instituts für Festkörperforschung und des Jülich Centre for Neutron Science haben nun erstmals die inneren Bewegungen eines Proteins in Raum und Zeit nachweisen können. Neutronenstreuung ermöglichte die Beobachtung in einer wässrigen Lösung, die den natürlichen Gegebenheiten in einer Zelle nahe kommt. Als Forschungsobjekt diente ein Protein namens Alkoholdehydrogenase, kurz ADH, das aus Bäckerhefe gewonnen wurde. Die ADH ist eines der Schlüsselenzyme bei der Produktion von Alkohol wie auch bei dessen Abbau.
"Bei der ADH konnten wir mit unserem Vorgehen erstmals das Ausmaß und die Schnelligkeit der räumlichen Bewegung ihrer großen Untereinheiten, genannt Domänen, nachweisen.", freut sich Prof. Dieter Richter, Direktor des Bereichs Neutronenstreuung. Eine zeitliche Auflösung von 100 Nanosekunden ermöglicht einen Blick auf das Auf- und Zuklappen einer Spalte zwischen jeweils zwei Untereinheiten, in der der Cofaktor gebunden wird. "Sogar die dabei herrschenden Kräfte konnten wir bestimmen", erläutert der Physiker. "Damit steht erstmals eine Methode zur Verfügung, mit der direkt zeitaufgelöste dreidimensionale Bewegungen in Proteinen beobachtet werden können." Die Methode eignet sich für Proteine mit einem Gewicht von 40 000 bis 200 000 u (1u = 1,66x10-27 kg).
Neutronen sind elektrisch neutrale Bausteine der Atomkerne, die in Forschungsreaktoren oder Spallationsquellen erzeugt und auf die zu untersuchenden Proben gelenkt werden. An den Atomen und Molekülen der Proben "prallen" sie ab; dabei können sie ihre Richtung und Geschwindigkeit ändern. Die Art dieser Streuung gibt Auskunft über die Anordnung und Bewegung der Atome in der Probe - ohne diese dabei zu zerstören. Für ihre Untersuchungen kombinierten die Jülicher Forscher zwei bekannte Methoden: Mittels so genannter Kleinwinkelstreuung bestimmten sie die Struktur, mittels Neutronenspinechospektroskopie die Bewegung. Die Kombination und ergänzende Simulationen ermöglichten es, störende Faktoren herauszurechnen, so dass im Ergebnis die Bewegungen innerhalb eines Einzelproteins herauskamen.
Originalveröffentlichung: Ralf Biehl et al.; "Direct observation of correlated interdomain motion in alcohol dehydrogenase"; Physical Review Letters 2008, Vol.101, No.13.