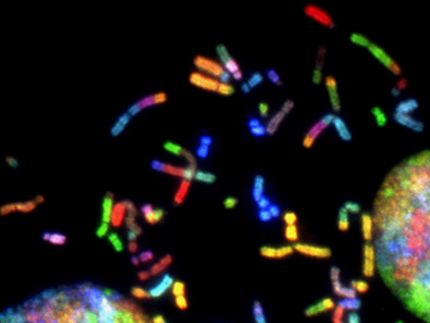
Genomlandschaften eines Tumors kartiert
Wissenschaftler haben eine hochauflösende Landkarte genetischer Veränderungen bei einem Lungenkarzinom erstellt
Die Entschlüsselung der Genome menschlicher Tumore ebnet nach der vollständigen Sequenzierung des menschlichen Erbguts den Weg, die Genomforschung medizinisch nutzbar zu machen. Eine erste vollständige Kartierung hat ein internationales Forscherteam nun abgeschlossen, dem auch ein Wissenschaftler vom Max-Planck-Institut für neurologische Forschung in Köln angehört. In einem hochauflösenden Genomscan analysierten sie nahezu 400 Proben des menschlichen Adenokarzinoms der Lunge. Dadurch konnten sie einen Katalog genetischer Veränderungen erstellen, der erstmalig die genomischen Landschaften dieses Tumortyps definiert. Sie fanden auch eine bisher unbekannte genetische Veränderung, die einen therapeutischen Ansatzpunkt bieten könnte.
Krebs ist eine Erkrankung des Erbguts. Veränderungen in der Struktur und der Sequenz von Genen können dazu führen, dass einige Signale in der Zelle beeinträchtigt werden. Spielen diese Signale eine Rolle bei der Zellteilung, hört die Zelle möglicherweise nicht mehr auf, sich zu vermehren. Die Sequenzierung des menschlichen Genoms und die begleitende technologische Revolution in der DNA-Analytik haben die Vorraussetzungen geschaffen, die Genome humaner Tumore systematisch zu analysieren. Aber Tumore eines bestimmten Typs wie zum Beispiel Lungenkrebs sind genetisch sehr inhomogen. Da nur ein kleiner Anteil der Tumorzellen jeweils die gleiche genetische Veränderung trägt, ist eine vollständige Sequenzierung sehr aufwendig.
In einem Kraftakt haben Wissenschaftler um Matthew Meyerson vom Broad-Institut des Massachusetts Institute of Technology (MIT) und der Harvard Universität in Cambridge nun erstmalig die genetischen Veränderungen eines humanen Tumors kartiert. Sie verglichen dabei die Anzahl der Genkopien auf den Chromosomen von normalen und entarteten Zellen. Durch die Beteiligung von drei der weltweit größten Genomforschungszentren, zahlreichen Cancer Centers in den USA, Deutschland und Japan und weiteren Forschungseinrichtungen ähnelt das Projekt in seiner Größe dem humanen Genomprojekt. Mitgestaltet und finanziert wurde es durch das National Human Genome Research Institute (NHGRI) und durch das National Cancer Institute (NCI) der USA.
Die Wissenschaftler sammelten weit über 500 Biopsieproben von Patienten mit einem Adenokarzinom der Lunge und unterzogen sie einer systematischen Qualitätskontrolle. In die Auswertung wurden schließlich 371 Proben aufgenommen. Untersucht wurden die Tumore mit sogenannten SNP-arrays. Diese Methode ermöglichte es, mithilfe von Genomsonden fast 250.000 Positionen auf dem Genom abzutasten. Aufgrund dieser Vielzahl von Messpunkten musste ein neues statistisches Verfahren entwickelt werden, das in der Lage war, eine solche Flut von Informationen auf einzelne, statistisch bedeutsame genetische Veränderungen herunterzubrechen. Das Verfahren heißt "Genomic Identification of Significant Targets in Cancer" (GISTIC), und erst seine Anwendung ermöglichte die Auswertung des Datensatzes in seiner ganzen Tiefe und Breite.
Neben bereits bekannten genetischen Veränderungen fanden die Wissenschaftler auch zahlreiche neue. Unter diesen befindet sich die hochgradige Vervielfältigung eines Gens, das unter dem Namen TITF1 bekannt ist. Dieses Gen kodiert für einen Lungen-Entwicklungsfaktor. Mäuse ohne TITF1 weisen schwere Störungen in der Lungenentwicklung auf. Um die Funktion des Gens genauer zu verstehen, schalteten die Wissenschaftler um Matthew Meyerson es in den Tumoren mit einer erhöhten Anzahl an Kopien aus. Als Folge wuchsen die Tumorzellen schlechter und bildeten keine Kolonien mehr. Die Vervielfältigung des TITF1 Gens im Tumor begünstigt offenbar das Tumorwachstum. Da diese Mutation in etwa zwölf Prozent der untersuchten Tumorproben vorhanden war, könnten einige Patienten mit einem Adenokarzinom der Lunge von einer therapeutischen Hemmung des TITF1-Gens profitieren.
Originalveröffentlichung: Barbara A. Weir, Michele S.Woo, Gad Getz, Sven Perner, Li Ding, Rameen Beroukhim, William M. Lin, Michael A. Province, Aldi Kraja, Laura A. Johnson, Kinjal Shah, Mitsuo Sato, Roman K. Thomas, Justine A. Barletta, Ingrid B. Borecki, Stephen Broderick, Andrew C. Chang, Derek Y. Chiang, Lucian R. Chirieac, Jeonghee Cho, Yoshitaka Fujii, Adi F. Gazdar, Thomas Giordano, Heidi Greulich, Megan Hanna, Bruce E. Johnson, Mark G. Kris, Alex Lash, Ling Lin, Neal Lindeman, Elaine R. Mardis, John D. McPherson, John D. Minna, Margaret B. Morgan, Mark Nadel, Mark B. Orringer, John R. Osborne, Brad Ozenberger, Alex H. Ramos, James Robinson, Jack A. Roth, Valerie Rusch, Hidefumi Sasaki, Frances Shepherd, Carrie Sougnez, Margaret R. Spitz, Ming-Sound Tsao, David Twomey, Roel G. W. Verhaak, George M. Weinstock, David A. Wheeler, Wendy Winckler, Akihiko Yoshizawa, Soyoung Yu, Maureen F. Zakowski, Qunyuan Zhang, David G. Beer, Ignacio I. Wistuba, Mark A. Watson, Levi A. Garraway, Marc Ladanyi, William D. Travis, William Pao, Mark A. Rubin, Stacey B. Gabriel, Richard A. Gibbs, Harold E. Varmus, Richard K. Wilson, Eric S. Lander & Matthew Meyerson; "Characterizing the cancer genome in lung adenocarcinoma"; Nature 2007.