Proteinnetzwerke auf dem Prüfstand für die medizinische Forschung
Saarbrücker Max-Planck-Wissenschaftler erstellen umfangreiche Qualitätsstudie zu menschlichen Proteininteraktionen
Proteine sind der molekulare Maschinenpark der Zelle. Oft interagieren Hunderte von ihnen miteinander, um die zahlreichen biologischen Aufgaben zu erfüllen - vom Stofftransport über die Signalleitung bis hin zur Krankheitsabwehr. Um diese Prozesse in der Zelle zu verstehen, ist es notwendig, das Netzwerk der Proteine und ihre Wechselwirkungen untereinander - das sogenannte Interaktom - zu kennen. Wissenschaftler vom Max-Planck-Institut für Informatik haben jetzt mehrere weltweit verfügbare Datensätze von Proteininteraktionen einer Qualitätsprüfung unterzogen. Ihr Ziel war es, die Verlässlichkeit dieser Interaktionsdaten zu ermitteln. Nur wirklich zuverlässige Interaktionen, die tatsächlich in der Zelle stattfinden, sollten für weitere biologische und medizinische Forschungsarbeiten herangezogen werden. Denn das Verständnis der Interaktionen zwischen menschlichen Proteinen kann bei der Suche nach neuen Ansätzen für die Behandlung von Krankheiten eine wichtige Rolle spielen.
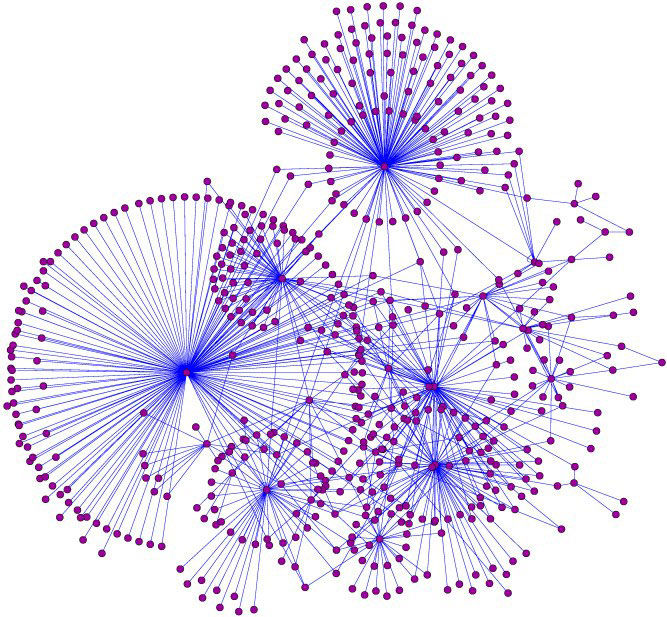
Dargestellt ist ein kleiner Teil eines molekularen Netzwerkes von menschlichen Proteinen (violette Punkte), die miteinander interagieren. Die Interaktionen der Proteine zur Erfüllung ihrer vielfältigen biologischen Aufgaben sind durch blaue Linien gekennzeichnet.
MPI für Informatik
Der Mensch hat schätzungsweise etwa 25.000 Gene, welche die Informationen für den Bau von mehreren Zehntausend Proteinen liefern. Proteine sind an nahezu allen Lebensvorgängen in der Zelle beteiligt und interagieren oft miteinander, um ihre biologischen Funktionen zu erfüllen. Sie bilden dabei molekulare Maschinen, die beispielsweise Stoffe transportieren, Signale weiter leiten, biochemische Vorgänge beschleunigen oder Krankheitserreger abwehren. Um die entsprechenden biologischen Prozesse in den Zellen zu verstehen, ist es notwendig, die verschiedenen molekularen Wechselwirkungen zwischen Proteinen zu kennen. Die Gesamtheit aller Proteininteraktionen, Interaktom genannt, wird auf 300.000 bis 400.000 geschätzt, von denen bisher nur wenig mehr als zehn Prozent experimentell ermittelt wurden.
Störungen von Interaktionen zwischen Proteinen und die damit verbundenen Beeinträchtigungen von zellulären Vorgängen können zum Auftreten verschiedener Krankheiten führen. Fachleute hoffen daher, dass die Aufklärung aller Proteininteraktionen und die detaillierte Analyse des menschlichen Interaktoms neue Ansätze zur Behandlung zahlreicher Erkrankungen liefern. Ein Beispiel dafür sind Infektionen mit Viren wie dem AIDS verursachenden HI-Virus oder dem Hepatitis C Virus. Hier ist es von besonderer Bedeutung, Interaktionen zwischen viralen und menschlichen Proteinen zu identifizieren, um die molekularen Mechanismen der Infektion besser zu verstehen und somit neue Therapien und Medikamente gegen Viren entwickeln zu können.
Es gibt verschiedene experimentelle Techniken, um Interaktionen zwischen Proteinen im Labor zu bestimmen. Da die Experimente aber sehr zeit- und kostenintensiv sind, werden auch rechnerische Methoden entwickelt, um Proteininteraktionen vorherzusagen. Jedoch sind bisher weder die experimentellen noch die computergestützten Methoden ganz ausgereift. Zum einen werden viele Proteininteraktionen noch nicht entdeckt, zum anderen sind einige der beschriebenen Interaktionen Artefakte, die in der Realität nicht vorkommen. Deswegen ist eine genaue Analyse der Interaktionsdaten und ihrer Verlässlichkeit notwendig, bevor sie in der biologischen und medizinischen Forschung verwendet werden können.
In einer umfangreichen Vergleichsstudie untersuchte die Forschungsgruppe von Mario Albrecht in der Bioinformatik-Abteilung von Thomas Lengauer am Max-Planck-Institut für Informatik mehrere weltweit verfügbare Datensätze von Interaktionen zwischen menschlichen Proteinen, die mit unterschiedlichen Methoden vorhergesagt oder experimentell bestimmt worden waren. Die Analyse umfasste verschiedene Aspekte der Proteinstruktur und -funktion und zeigte unter anderem, dass viele Interaktionen nur jeweils in einem Datensatz vorhanden sind. "Wir führen das darauf zurück, dass die unterschiedlichen Methoden verschiedene Teilbereiche des gesamten humanen Interaktoms abdecken", erklärt der am Forschungsprojekt beteiligte Bioinformatiker Fidel Ramírez. In anderen Fällen wurden bestimmte Interaktionen von mehreren Methoden zugleich vorhergesagt, was die einzelne Interaktion glaubwürdiger macht. Der Anteil dieser zuverlässigen Interaktionen an der gesamten bis jetzt vorliegenden Datenmenge von Proteininteraktionen ist allerdings noch gering.
Ein weiterer Schritt war die Analyse der molekularen Funktion der an Interaktionen beteiligten Proteine, weil Proteininteraktionen meist der Ausführung einer gemeinsamen Aufgabe dienen. Mit Hilfe des von Andreas Schlicker am Max-Planck-Institut entwickelten funktionellen Ähnlichkeitsmaßes erkennt man bei Interaktionen mit bekannter hoher Verlässlichkeit, dass die beteiligten Proteine auch eine hohe funktionelle Ähnlichkeit aufweisen. Anhand bereits bekannter Funktionen von Proteinen kann man so im Umkehrschluss überprüfen, ob eine gefundene Interaktion zwischen zwei Proteinen funktionell sinnvoll ist und sie vermutlich tatsächlich in der lebenden Zelle stattfindet - oder aber, ob die Interaktion auf einen technischen Fehler der angewandten Methode zurückzuführen ist.
"Unsere Qualitätsstudie zu menschlichen Proteininteraktionen hilft, die verfügbaren experimentellen und vorhergesagten Interaktionsdaten zu filtern, um nur wirklich zuverlässige Informationen in nachfolgenden biologischen und medizinischen Untersuchungen weiter zu verwenden. Hierdurch können die Erfolgschancen von Experimenten zu molekularen Ursachen vieler Erkrankungen erheblich verbessert werden", resümiert Mario Albrecht.
Originalveröffentlichung: Fidel Ramírez, Andreas Schlicker, Yassen Assenov, Thomas Lengauer, Mario Albrecht, "Computational analysis of human protein interaction networks"; Proteomics 2007, Volume 7, Number 15, August 2007.