Wie Schalter in Bakterien funktionieren
Struktur und Dynamik von Riboschaltern in lichtoptischen Einzelmolekülexperimenten untersucht
Viele Bakterien besitzen molekulare Kontrollelemente, über die sie Gene an- und abschalten können. Diese Riboschalter eröffnen neue Möglichkeiten bei der Entwicklung von Antibiotika oder auch zum Aufspüren und Abbauen von Umweltgiften. Wie die Riboschalter funktionieren, haben Forscher des Karlsruher Instituts für Technologie (KIT), der Universität Heidelberg und der Freien Universität Berlin nun anhand von lichtoptischer Mikroskopie an Einzelmolekülen grundlegend untersucht.
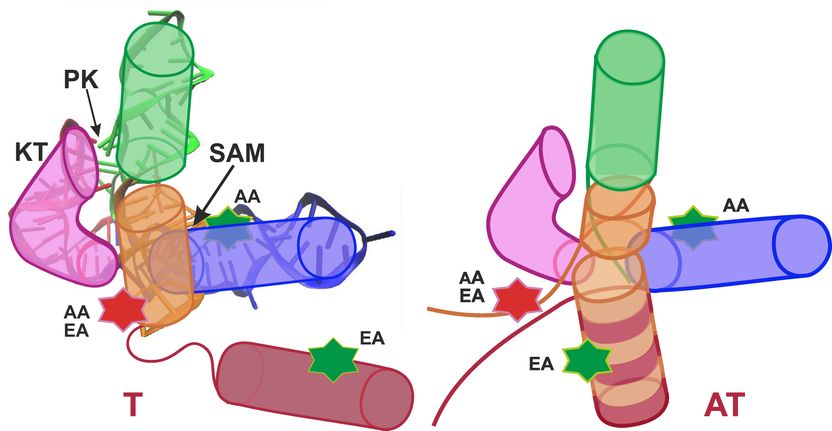
Schematische Darstellung des SAM-I Riboschalters in der Terminator-Konformation (Schalter aus; links) und der Antiterminator-Konformation (Schalter ein; rechts).
APH/KIT
Riboschalter (Riboswitches) liegen auf der Boten-Ribonukleinsäure (mRNA), die genetische Information zum Ort der Proteinbiosynthese transportiert. Ein Riboschalter besteht aus einem Sensor, der die Konzentration eines kleinen Stoffwechselmoleküls misst, und einem Effektor, der die Genexpression und damit die Synthese eines Proteins steuert. Weil Riboschalter in vielen bakteriellen Krankheitserregern vorkommen, stellen sie bei der Entwicklung neuer Antibiotika wichtige Angriffsziele dar. Weitere Anwendungen eröffnen sie in der synthetischen Biologie. Zum Beispiel lassen sich Bakterien mit Riboschaltern gentechnisch so modifizieren, dass sie niedermolekulare Umweltgifte, wie Herbizide, aufspüren und abbauen können. Voraussetzung ist allerdings ein grundlegendes Verständnis der Prozesse, auf denen die Funktion der Riboswitches beruht. Dazu trägt die nun im Magazin Nature Chemical Biology vorgestellte Arbeit wesentlich bei.
Die von der Heidelberg Karlsruhe Research Partnership (HEiKA) geförderte Arbeit entstand in Kooperation der Forschergruppen um Professor Gerd Ulrich Nienhaus am Institut für Angewandte Physik (APH), Institut für Nanotechnologie (INT) und Institut für Toxikologie und Genetik (ITG) des KIT sowie um Professor Andres Jäschke am Institut für Pharmazie und Molekulare Biotechnologie (IPMB) der Universität Heidelberg. Im Fokus der Arbeit steht der S-Adenosyl-L-Methionin (SAM)-I Riboschalter. „Bei diesem Riboschalter bewirkt die Anbindung des SAM-Moleküls, dass sich die Konformation, das heißt die räumliche Anordnung der Atome, von der Antiterminator (AT)-Struktur zur Terminator (T)-Struktur hin verändert“, erklärt Nienhaus. „Dadurch wird die Genexpression abgeschaltet.“
Zunächst synthetisierten die Wissenschaftler in Heidelberg die SAM-I Riboschalter und markierten sie an verschiedenen Stellen gezielt mit je zwei unterschiedlichen Fluoreszenzfarbstoffen. Die Forscher am KIT untersuchten diese RNA-Moleküle dann in hoher räumlicher und zeitlicher Auflösung mit höchst sensitiven Lichtmikroskopen, welche die Fluoreszenzemission einzelner Farbstoffmoleküle registrierten. In Förster-Resonanzenergietransfer-Experimenten (FRET) ließ sich die Konformationsdynamik direkt erfassen: Ein grüner Farbstoff wird dazu mit Laserlicht zur Lichtemission angeregt. Befindet sich in der Nähe ein roter Farbstoff, kann dieser die Anregungsenergie des grünen Farbstoffs übernehmen und selbst Licht aussenden. Die Wahrscheinlichkeit, dass ein Energietransfer stattfindet, ist stark abhängig vom Abstand der Farbstoffe zueinander, sodass sich Strukturänderungen eines Moleküls, an das die Farbstoffe gezielt angebracht sind, direkt über die Emission des roten Farbstoffs beobachten lassen. Da die Lichtemission extrem schwach ist, bedurfte es aufwendiger Datenanalyseverfahren, basierend auf Hidden Markov Modeling. Professorin Bettina Keller vom Institut für Chemie und Biochemie der Freien Universität Berlin entwickelte die Verfahren speziell für diese Art von Experimenten, um die zeitabhängigen Lichtemissionssignale aus dem Rauschen herauszuheben.
Bei der Analyse gelang es den Forschern, nicht nur zwei Konformationen (T und AT) des SAM-I Riboschalters zu unterscheiden, sondern insgesamt vier (T1, T2, AT1 und AT2). Überraschend war auch, dass der Riboschalter in An- und Abwesenheit von SAM nicht, wie eigentlich zu erwarten war, vollständig zwischen T- und AT-Strukturen hin und her schaltete, sondern ständig zwischen allen Zuständen hin und her fluktuierte; lediglich deren Gewichtungen waren verschoben. Ein für die biologische Funktion besonders wichtiges Ergebnis war, dass die beobachteten Strukturfluktuationen mit angedocktem SAM deutlich schneller waren als ohne SAM. Da die Riboschalter-Sequenz auf der Boten-RNA direkt vor dem zu steuernden Gen liegt, muss das RNA-Molekül nach der Synthese – bei Anwesenheit von SAM – schnellstmöglich eine T-Struktur (Schalter aus) einstellen können, um die anschließende Transkription des zu steuernden Gens zu verhindern. Die Beschleunigung der Strukturfluktuationen durch SAM-Bindung stellt demnach sicher, dass die T-Struktur schnell genug eingestellt werden kann. „Mithin spielt die Dynamik des SAM-I Riboschalters eine entscheidende Rolle für seine Funktion“, resümiert Nienhaus vom KIT. „Diese detaillierten Einblicke in die Funktionsweise eines Biomoleküls verdanken wir einem fächerübergreifenden Forschungsansatz mit Beiträgen aus der Physik, der Biotechnologie und der theoretischen Chemie.“























































