Achillesferse von Bakterien entdeckt
Salmonellen sind besonders widerstandsfähig gegen Antibiotika, denn sie besitzen gleich zwei Membranen, die sie vor äußeren Einflüssen schützen. Damit gehören sie zu den im Fachjargon als gramnegativ bezeichneten Bakterien. Da Infektionen mit diesen Erregern immer schwerer mit Antibiotika zu behandeln sind, suchen Wissenschaftler nach alternativen Wirkstoffen. Ein Ansatzpunkt dafür sind die bakteriellen Antriebe: Viele gramnegative Bakterien bilden lange Filamente aus – sogenannte Flagellen, die sie in Rotation versetzen und sich damit gezielt fortbewegen. Ohne intakte Flagellen könnten die Bakterien keine Nahrungsquellen ansteuern und könnten auch nicht in fremde Zellen eindringen – sie wären nicht mehr infektiös. Wissenschaftler des Helmholtz-Zentrums für Infektionsforschung (HZI) in Braunschweig haben gemeinsam mit Partnern herausgefunden, dass ein bestimmtes Protein die ersten Schritte des Flagellenaufbaus organisiert. War dieses Protein ausgeschaltet, konnten die Bakterien keine Flagellen mehr bilden. So wären sie auch in ihrer Fähigkeit, einen Wirt zu infizieren, stark eingeschränkt.
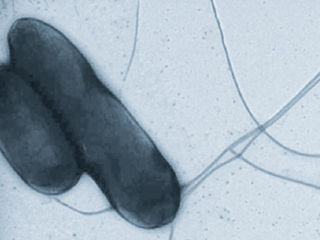
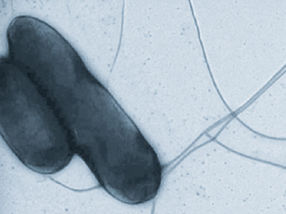
Salmonellen
© HZI/Manfred Rohde
Salmonellen nutzen wie viele andere Bakterien sogenannte Flagellen zur gezielten Bewegung. Diese langen Filamente sind in einem komplexen Basalkörper aus einer Vielzahl verschiedener Proteine in der Bakterienmembran verankert
© Marc Erhardt
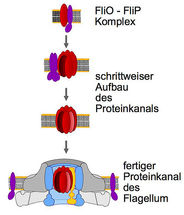
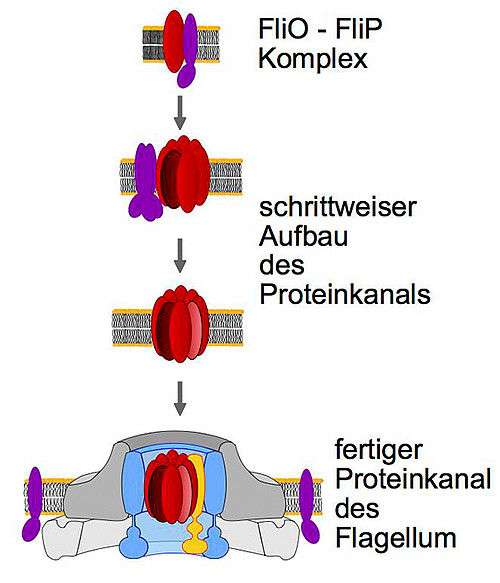
Auf der Suche nach Nahrung oder einem geeigneten Wirt bilden viele Bakterien Flagellen – das sind lange Filamente aus tausenden Proteinen, die die Bakterien wie einen Propeller einsetzen, um sich fortzubewegen. Bei der Bildung eines neuen Flagellums lagern sich zunächst verschiedene Bausteine in der Zellmembran zu einer Pore zusammen, aus der das Flagellum wächst. Diese Pore ist ein Kanal für die Bausteine des Flagellums und zugleich der zentrale Bestandteil eines komplizierten, weit verbreiteten Proteintransportsystems – des sogenannten Typ III-Sekretionssystems. Dieses komplexe Gebilde ähnelt im Aufbau einer anderen Struktur vieler Bakterien: einer molekularen Spritze, mit der krankheitserregende Bakterien während einer Infektion zum Beispiel Giftstoffe in Wirtszellen schießen und sie so zum Absterben bringen. Forscher vermuten, dass sich der Bauplan des Spritzenapparates im Laufe der Evolution aus dem der Flagellen entwickelt hat.
Sowohl Flagellen als auch die molekularen Spritzen sind notwendige Werkzeuge, die Bakterien für eine Infektion benötigen. „Gelänge es, diese Werkzeuge auszuschalten, wären die Bakterien für den Menschen harmlos“, sagt Dr. Marc Erhardt, der am HZI die Nachwuchsgruppe „Infektionsbiologie von Salmonellen“ leitet. Gemeinsam mit Kooperationspartnern der Universität Osnabrück, des Max-Planck-Instituts für Infektionsbiologie, der Universität Tübingen und des Deutschen Zentrums für Infektionsforschung hat das Team um Erhardt den Aufbauprozess von Flagellen am Bakterium Salmonella enterica im Detail untersucht. „Viele verschiedene Proteine sind am Aufbau der initialen Pore beteiligt, bislang waren ihre individuellen Rollen aber nicht bekannt“, sagt Erhardt.
Mithilfe von Fluoreszenzfarbstoffen haben die Forscher zunächst sichtbar machen können, wo welches Protein lokalisiert ist. So fiel ihnen auf, dass eines der beteiligten Proteine, abgekürzt mit FliO, gar nicht dauerhaft an der Verankerung der Flagellen sitzt. Es bewegt sich frei in der Zellmembran und wandert nur während der Initiierung einer neuen Pore an die jeweilige Stelle. Dort bildet es einen Komplex mit dem Protein FliP, welches hauptsächlich die Pore aufbaut. In einem Experiment schalteten die Forscher die genetische Information für FliO in den Salmonellen aus. Das Ergebnis: Die Bakterien konnten keine funktionsfähigen Poren und damit kein Flagellum mehr bilden. „Da das Protein FliO selbst kein Bestandteil der Pore ist, arbeitet es offenbar wie ein Organisator: Es hilft anderen Proteinen, sich korrekt aneinanderzulagern und so die Pore aufzubauen“, sagt Marc Erhardt.
In weiteren Untersuchungen konnten die Forscher zeigen, dass das Protein FliP zwar auch ohne den Organisator FliO Komplexe bildete. Diese waren jedoch ungeordnet, sodass sich keine weiteren Bausteine der Pore anlagern konnten – die Komplexe wurden schließlich wieder entsorgt. Da FliO den einzelnen FliP-Proteinen bei der korrekten Komplexbildung hilft, fungiert es als sogenanntes Chaperon. „Diese Funktion von FliO war bislang nicht bekannt“, sagt Erhardt. Sie eröffne neue Ansatzpunkte für künftige Wirkstoffe, die gleich gegen eine ganze Reihe krankheitserregender Bakterien eingesetzt werden könnten.
„Ein Wirkstoff, der zum Beispiel die Porenbildung verhindert, wäre gleich ein doppelter Erfolg“, sagt Marc Erhardt. „Er würde Bakterien treffen, die Flagellen ausbilden, und auch solche, die molekulare Spritzen einsetzen.“ So sind Salmonellen, Escherichia coli, Yersinien und Pseudomonaden typische gramnegative Krankheitserreger, die ihre Wirte mithilfe eines Spritzenapparates infizieren. Dagegen besitzen zum Beispiel Bakterien der Gattungen Campylobacter und Helicobacter, die Darm- beziehungsweise Magenerkrankungen hervorrufen, keine molekularen Spritzen. Sie bilden jedoch Flagellen aus und liefern somit ebenfalls einen Angriffspunkt für einen alternativen Wirkstoff. „Ein solcher Wirkstoff hätte gegenüber einem Antibiotikum auch den Vorteil, dass die Erreger am Leben blieben und so nicht dem Druck ausgesetzt wären, Resistenzen gegen die Substanz entwickeln zu müssen“, sagt Erhardt. „Außerdem würden die nützlichen Bakterien im Körper ebenfalls überleben.“
Originalveröffentlichung
Florian D. Fabiani, Thibaud T. Renault, Britta Peters, Tobias Dietsche, Eric J.C. Gálvez, Alina Guse, Karen Freier, Emmanuelle Charpentier, Till Strowig, Mirita Franz-Wachtel, Boris Macek, Samuel Wagner, Michael Hensel, Marc Erhardt; "A flagellum-specific chaperone facilitates assembly of the core type III export apparatus of the bacterial flagellum"; PLOS Biology; 2017

























































