Hop hält „springende Gene“ in Schach
Biologen entwickeln bioinformatische Algorithmen zur Auswertung riesiger Datensätze
Die Erbinformation höherer Lebewesen wird als genetischer Code in Form von DNA-Bausteinen auf den Chromosomen kodiert. In nahezu allen bis heute untersuchten Organismen findet man neben den funktionell relevanten Genen aber auch „parasitische DNA“, sogenannte springende Gene oder Transposons. Diese springenden Gene können sich im Genom ihres jeweiligen Wirts ausbreiten, indem sie sich selbst kopieren oder ausschneiden und an anderer Stelle eines Chromosoms wieder einfügen. Transposons haben in der Regel keinen direkten Nutzen für ihren Wirtsorganismus, sondern stellen eine Gefahr für die Integrität des Genoms dar. Es ist daher nicht überraschend, dass man innerhalb einer Zelle eine Vielzahl molekularer Mechanismen findet, die an der Kontrolle solcher Transposons beteiligt sind und deren Ausbreitung verhindern. Einer dieser Mechanismen, der speziell Keimbahnzellen schützt, beruht auf bestimmten Proteinen der Argonauten-Klasse, den Piwi-Proteinen, sowie auf kleinen RNA-Molekülen, die an diese Proteine gebundene sind, den piRNAs. Piwi-Proteine können gezielt Transposons deaktivieren, da ihre piRNAs die Transposons nach dem Schlüssel-Schloss-Prinzip erkennen.
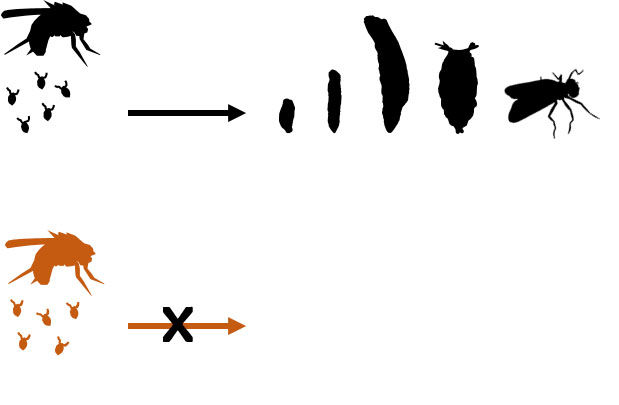
Aus den Eiern von Fruchtfliegen schlüpfen Larven, aus denen sich über drei Larvenstadien und ein Puppen-Stadium die adulten Fliegen der nächsten Generation entwickeln (schwarz). Fliegen mit deaktiviertem Hop-Gen (orange) legen zwar Eier, jedoch schlüpfen aus diesen keine Larven.
©: David Rosenkranz, JGU
Dank intensiver Forschung verstehen Mikrobiologen heute recht gut, wie diese Piwi-basierten Mechanismen funktionieren und welche Proteine bei der Kontrolle von Transposons beteiligt sind, auch wenn viele Details noch immer rätselhaft bleiben. Nun konnte ein Team um Vamsi Gangaraju von der Medizinischen Universität South Carolina, Charleston, USA, in Kooperation mit Dr. David Rosenkranz von der Johannes Gutenberg-Universität Mainz (JGU) ein weiteres essentielles Puzzleteil, ein Gen namens Hop, identifizieren.
Studien dieser Art beruhen auf der Analyse von Millionen verschiedener kleiner RNA-Moleküle. Zwar kann heute dank neuer Hochdurchsatz-Sequenziermethoden prinzipiell jede Arbeitsgruppe mit wenig Aufwand riesige Sequenzdatensätze erzeugen, allerdings bleibt die bioinformatische Auswertung dieser Daten eine Herausforderung. Die Arbeitsgruppe um David Rosenkranz vom Institut für Organismische und Molekulare Evolutionsbiologie hat sich in den letzten Jahren auf die Auswertung solcher Datensätze spezialisiert und eine Reihe bioinformatischer Algorithmen entwickelt, die zur Auswertung der Daten der vorliegenden Studie angewendet wurden.
Die Ergebnisse zeigen, dass die Produktion von piRNAs entscheidend gestört ist, wenn das Hop-Gen in weiblichen Fliegen gezielt ausgeschaltet wird. Dies hat zur Folge, dass Transposons aktiviert werden, was wiederum zum programmierten Zelltod führt. Fliegen mit deaktiviertem Hop-Gen legen zwar in gleichem Maße Eier wie ihre Wildtyp-Pendants, jedoch entwickeln sich diese nicht zu Larven weiter, sondern sterben ab.
Originalveröffentlichung
Weitere News aus dem Ressort Wissenschaft

Holen Sie sich die Life-Science-Branche in Ihren Posteingang
Mit dem Absenden des Formulars willigen Sie ein, dass Ihnen die LUMITOS AG den oder die oben ausgewählten Newsletter per E-Mail zusendet. Ihre Daten werden nicht an Dritte weitergegeben. Die Speicherung und Verarbeitung Ihrer Daten durch die LUMITOS AG erfolgt auf Basis unserer Datenschutzerklärung. LUMITOS darf Sie zum Zwecke der Werbung oder der Markt- und Meinungsforschung per E-Mail kontaktieren. Ihre Einwilligung können Sie jederzeit ohne Angabe von Gründen gegenüber der LUMITOS AG, Ernst-Augustin-Str. 2, 12489 Berlin oder per E-Mail unter widerruf@lumitos.com mit Wirkung für die Zukunft widerrufen. Zudem ist in jeder E-Mail ein Link zur Abbestellung des entsprechenden Newsletters enthalten.






















































