DNA-Reparatur: Ein neuer Buchstabe im Zellalphabet
Studie offenbart, wie „blinde Flecken“ in der Wissenschaft entstehen können
Das Erbgut in unseren Zellen muss ständig repariert werden, um die Entstehung von Krankheiten, wie zum Beispiel Krebs, zu verhindern. Dafür schickt die Zelle „Reparatur-Proteine“ zu den geschädigten Bereichen der DNA. Um diese zu steuern, haben Zellen ein präzises Alphabet entwickelt. Jetzt haben Wissenschaftler vom Max-Planck-Institut für Biologie des Alterns einen neuen Weg entdeckt, wie ein Buchstabe dieses Zellalphabets mit Proteinen verknüpft wird. Diese neue Proteinmodifikation, genannt Serin-ADP-Ribosylierung, wurde jahrzehntelang von Wissenschaftlern übersehen. Daher offenbart diese Studie auch, wie in der Wissenschaft „blinde Flecken“ entstehen können.
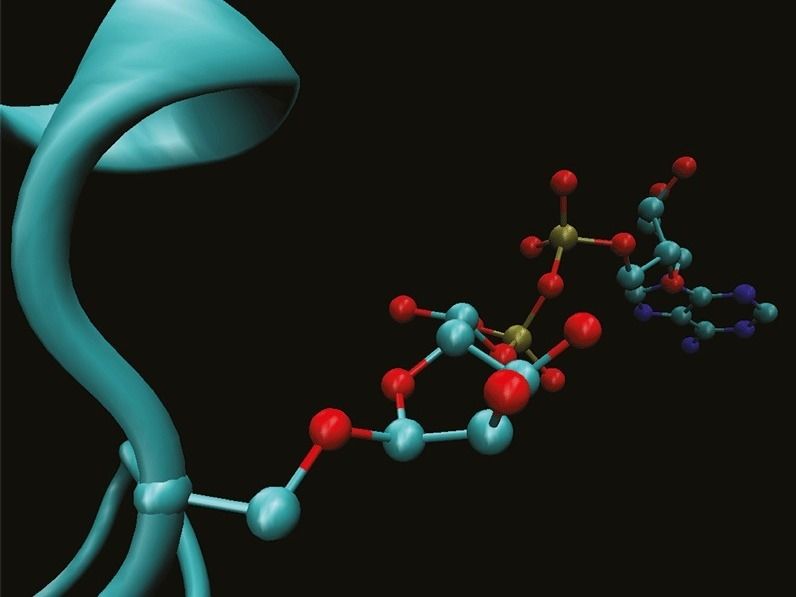
Komplexe Markierung für die DNA-Reparatur: Die 3D-Darstellung zeigt die Anheftung der ADP-Ribosylierung an die Aminosäure Serin im Protein (türkis).
© MPI f. Biologie des Alterns
In der Grundlagenforschung beginnen neue Forschungsprojekte häufig damit, bestehende Ergebnisse zu reproduzieren, um auf diesen dann die eigene Studie aufzubauen. Das war auch genau der Plan einer jungen Forschungsgruppe unter der Leitung von Ivan Matic am Max-Planck-Institut für Biologie des Alterns in Köln. Am Ende fand die Forschungsgruppe in Kollaboration mit Ivan Ahel von der Universität in Oxford einen völlig neuen Mechanismus, der einige der alten Erkenntnisse auf den Kopf stellt.
Die Forschungsgruppe untersucht, wie Zellen das Schicksal von Proteinen bestimmen, in dem sie präzise verschiedene Markierungen, genannt „posttranslationale Modifikationen“, an Proteine anheftet. Dieses sind kleine chemische Etiketten, die Proteine aktivieren und funktionstüchtig werden lassen. Der Zelle steht dabei ein ganzes Repertoire an Markierungen zur Verfügung, welches sie wie ein Alphabet nutzen kann, um in etwa Proteine in den Zellkern zu schicken, die dort dann beschädigtes Erbgut reparieren. „Wir haben eine der komplexesten Markierungen untersucht – die Serin-Adenosin-Diphosphat-Ribosylierung. Es wurde in diesem Forschungsgebiet seit vielen Jahren angenommen, dass diese Markierung an einen bestimmten Teil im Protein angeheftet wird, nämlich an die Aminosäuren Arginin, Lysin, Glutaminsäure und Asparaginsäure. Als wir uns unsere Ergebnisse jedoch im Detail angesehen haben, fiel uns auf, dass die Aminosäure Serin immer ganz in der Nähe des angenommenen Anheftungspunkts auftauchte. Das hat uns skeptisch gemacht. Nach vielen weiteren Untersuchungen waren wir uns dann sicher, dass tatsächlich die Aminosäure Serin der Anknüpfungspunkt für die Markierung des Proteins ist“, erzählt Matic.
Für Nicht-Wissenschaftler mag sich das wie ein kleines Detail anhören. Aber in der Zelle macht es einen großen Unterschied. Die Forscher konnten zeigen, dass die Modifikation einer Schlüsselrolle bei der Reparatur des Erbguts spielt. Diese Reparatur ist lebensnotwendig für alle Organismen - auch für den Menschen. Mit der Entdeckung des neuen Buchstabens haben die Wissenschaftler jetzt die Grundlage für eine weitere Entschlüsselung der DNA-Reparatur gelegt. „Unsere Forschung eröffnet neue Möglichkeiten für eine Verbesserung der Effizienz der DNA-Reparatur Maschinerie“, kommentiert Juan José Bonfiglio, Forscher in der Matic Gruppe.
Blinder Fleck
Aber wie kann es sein, dass diese Modifikation so viele Jahre übersehen wurde? Tom Colby, Wissenschaftler in der Matic Gruppe, versucht es zu erklären: „Von Wissenschaftlern wird heutzutage verlangt, dass sie große Datenmengen produzieren und analysieren. Das bedeutet, dass man auf schon entwickelte Werkzeuge zurückgreifen muss und diese dann auf das eigene biologische System anwendet. Das Problem dabei ist aber, dass diese Werkzeuge häufig auf alten Erkenntnissen basieren. Werden diese nicht überarbeitet und überprüft, können „blinde Flecken“ entstehen. Die interessantesten Ergebnisse können sich also in diesen „blinden Flecken“ verstecken“, Matic fügt hinzu:“ Ich bin altmodisch. Ich trete gerne einen Schritt zurück und sehe mir die Originalergebnisse im Detail an. Wenn ich das nicht gemacht hätte, hätten wir diese Modifikation genauso übersehen wie die Wissenschaftler in den Jahren zuvor.“
Originalveröffentlichung
Juan José Bonfiglio, Pietro Fontana, Qi Zhang, Thomas Colby, Ian Gibbs-Seymour, Ilian Atanassov, Edward Bartlett, Roko Zaja, Ivan Ahel and Ivan Matic; "Serine ADP-ribosylation depends on HPF1"; Molecular Cell; February, 2017
Orsolya Leidecker, Juan José Bonfiglio, Thomas Colby, Qi Zhang, Ilian Atanassov, Roko Zaja, Luca Palazzo, Anna Stockum, Ivan Ahel and Ivan Matic; "Serine is a new target residue for endogenous ADP-ribosylation on histones"; Nature Chemical Biology; December, 2016