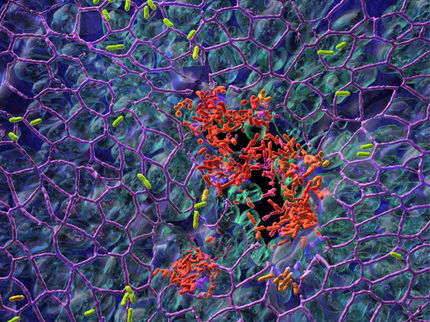
Strategien multiresistenter Erreger in der Nase verstehen
Staphylococcus aureus durch Adaption seines Stoffwechsels sehr gut an Stress angepasst
Staphylococcus aureus ist ein oft multiresistenter Erreger von Wundinfektionen und kommt bei 20 bis 30 Prozent der Menschen als natürlicher Begleiter hauptsächlich in der Nasenflora vor. Um sein Verhalten in dieser Umweltnische besser zu verstehen, führten Wissenschaftler des Helmholtz-Zentrums für Infektionsforschung (HZI) in Braunschweig jetzt erstmals eine sogenannte Transkriptom-Studie durch, eine Analyse aller in der Zelle aktiven Gene. Das Ergebnis: Der Keim zeigt eine sehr gute Stressanpassung, indem er seinen Stoffwechsel adaptiert. Dadurch ist er in der Lage, seine Zellwandzusammensetzung zu verändern oder niedermolekulare organische Verbindungen aufzunehmen oder zu synthetisieren und dadurch erhöhte Salzkonzentrationen in der Nase zu überleben. Die Erkenntnisse aus den genetischen Analysen des Keims im Wirt und die Gemeinsamkeiten zwischen unterschiedlichen Stapyhlococcus aureus-Arten könnten zukünftig dabei helfen, multiresistente Krankheitserreger in der Nase von Patienten in Krankenhäusern zu bekämpfen.
Unsere Nasenhöhle ist natürlicherweise durch eine Vielzahl von Mikroorganismen besiedelt, deren Interaktionen untereinander und mit dem menschlichen Wirt von bisher unverstandener Komplexität und Dynamik sind. Sie ist auch das Habitat von Staphylococcus aureus, einem gefürchteten, oft multiresistenten Erreger schwerer Infektionen. Auch wenn das Tragen des Keims meist symptomlos verläuft, spielt die Besiedelung der Nase eine wichtige Rolle als kritische Infektionsquelle in der Bevölkerung und in Krankenhäusern. Wenn der Erreger in offene Wunden eindringt, kann das sehr gefährlich werden. Seit einigen Jahren wird es zunehmend zum Problem, weil immer häufiger MRSA-Bakterien auftreten – methicillin-resistente Staphylococcus aureus-Stämme, gegen die viele gängige Antibiotika unwirksam sind. Der Grund für das gehäufte Auftreten dieser MRSA-Keime ist bisher noch unklar und kann nach Ansicht der Forscher nicht allein mit dem Selektionsdruck durch Antibiotika erklärt werden. Die Wissenschaftler am Helmholtz-Zentrum für Infektionsforschung versuchen nun, mit modernsten Sequenziermethoden und umfangreichen bioinformatischen Analysen aufzuklären, wie sich S. aureus in seiner ökologischen Nische, der Nase mit ihren Umweltbedingungen und der mikrobiellen Nachbarschaft, durchsetzt.
„Staphylococcus aureus gehört als Erreger von Wundinfektionen beim Menschen zu den meist studierten bakteriellen Arten und sein Verhalten unter Laborbedingungen, als in vitro bezeichnet, wurde schon im Detail untersucht. Mit dem Aufkommen der Sequenzierung im Hochdurchsatz steigt nun aber das Interesse daran, den Keim in vivo, also sein Verhalten im Menschen, zu erforschen“, erklärt HZI-Wissenschaftler Prof. Dietmar Pieper, Leiter der Arbeitsgruppe „Mikrobielle Interaktionen und Prozesse“. Die derzeitige Sequenziertechnik ermöglicht es unter anderem, die gesamte mRNA, also die Abschriften aller aktiven Gene, von Staphylococcus zu analysieren und so zum Beispiel Rückschlüsse auf den Stoffwechsel der Bakterien in der menschlichen Nase zu ziehen. „Um besser zu verstehen, wie Staphylococcus aureus in seiner ökologischen Nische überlebt, müssen wir den Keim im lebenden Wirt analysieren.“ Das sei im Labor nur begrenzt möglich, weil man die natürliche Umgebung in der Nase und die Interaktionen mit dem menschlichen Wirt nicht exakt simulieren könne. Zudem vernachlässige der Laborversuch die komplexe Bakteriengemeinschaft, die in der Nase lebt, dort um die Nährstoffe konkurriert und mit Staphylococcus aureus interagiert. „Unsere ersten Experimente mit bioinformatischer Auswertung auf Transkriptomebene zeigen, dass sich das Verhalten von Staphylococcus aureus signifikant von dem in vitro beobachteten Verhalten unterscheidet“, sagt Pieper.
Für die Pilotstudie entnahmen die Wissenschaftler drei freiwilligen Personen, die Träger des Keims sind, zu zwei unterschiedlichen Zeitpunkten Proben aus den vorderen Nasenhöhlen. „Es war eine sehr große Herausforderung, genug Probenmaterial für eine ausreichend gute RNA-Sequenzierung aus der Nase zu bekommen, da man viel mehr menschliche Zellen als Bakterien isoliert“, sagt der HZI-Wissenschaftler Dr. Diego Chaves-Moreno, Mikrobiologe und Bioinformatiker im Team von Dietmar Pieper. „Außerdem mussten für diese ersten Versuche bioinformatische Methoden weiterentwickelt werden, um eindeutig zwischen S. aureus und anderen Staphylococcus-Stämmen in der Nase unterscheiden zu können. Erste Trends sind aus diesen Analysen schon sehr gut erkennbar.“
Um einen Überblick über das Verhalten und die Interaktionen von Staphylococcus aureus in der menschlichen Nase zu erhalten, analysierten die Wissenschaftler die Expression sämtlicher ca. 3000 S. aureus- Gene. Die dabei erhaltenen Transkriptomprofile wurden mit denen von S. aureus-Stämmen, die unter Laborbedingungen kultiviert wurden, verglichen. Die Resultate enthüllten sehr clevere Anpassungsstrategien des Keims an seine ökologische Nische in der menschlichen Nase.
„Das innere Milieu der Nase ist feucht, salzig und ein sehr stressreiches Milieu für Mikroorganismen. Staphylococcus aureus hat sich an diese Umweltnische angepasst“, sagt Dietmar Pieper. „Der Keim zeigt eine sehr gute Stressanpassung, indem er seinen biosynthetischen Stoffwechsel adaptiert.“ Dadurch sei er in der Lage, seine Zellwandzusammensetzung zu verändern oder sogenannte „kompatible Solute“ aufzunehmen oder zu synthetisieren – kleine organische Moleküle, die der Zelle helfen können, den osmotischen Stress zu überwinden.
„Durch die Analyse der allgemeinen Genexpressionsmuster und der Gemeinsamkeiten zwischen den Staphylococcus aureus-Stämmen in unterschiedlichen menschlichen Wirten können wir nun neue Erkenntnisse gewinnen, um solche Organismen in der Nase zu bekämpfen“, sagt Dietmar Pieper. „Die Wirtsumgebung in der menschlichen Nase hat einen starken Einfluss auf die exprimierten Gene. Um sowohl die Besiedlung und die Balance zwischen opportunistischen Krankheitserregern und harmlosen Begleitern als auch Infektionsprozesse besser zu verstehen, sind in Zukunft weitere in vivo-Studien mit mehr Probanden nötig.“
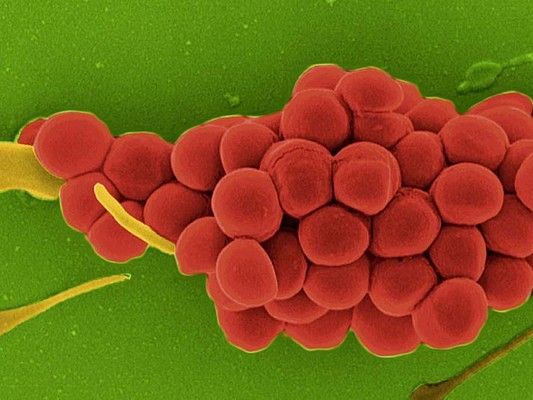
Rasterelektronenmikroskopische Aufnahme von Zellen des Eiter-Erregers Staphylococcus aureus
HZI/ Manfred Rohde