Neue Methode gibt "lebensnahe" Einblicke in zelluläre Prozesse
Proteine sind die Grundbausteine allen Lebens auf der Erde und spielen eine entscheidende Rolle bei fast allen Prozessen, die in den Zellen von Organismen stattfinden. Ein internationales Forscherteam unter Leitung von Dr. Manuel Etzkorn am Institut für Physikalische Biologie der Heinrich-Heine-Universität Düsseldorf hat nun ein Verfahren entwickelt, mit denen die Strukturen und Interaktionen der Bio-Moleküle unter wesentlich natürlicheren Bedingungen als bisher sichtbar gemacht werden können. Damit ermöglicht die Methode neuartige Einblicke in biologische Systeme. Die Forscher nutzten hierfür ein neuartiges NMR-Spektrometer des Biomolekularen NMR-Zentrums auf dem Campus des Forschungszentrums Jülich.
Mithilfe eines neuartigen DNP-verstärkten Hochfeld-NMR-Spektrometers (Bild oben) gelang es den Forschern zum ersten Mal, die Signale eines ausgewählten Proteins direkt in einer aus Bakterienzellen gewonnener Lösung zu messen. DNP steht für Dynamic Nuclear Polarization. Das Gerät wurde durch Prof. Henrike Heise (HHU, Institut für Physikalische Biologie) bei der Deutschen Forschungsgemeinschaft eingeworben und 2014 im Biomolekularen NMR-Zentrum der HHU Düsseldorf und des Forschungszentrums Jülich in Betrieb genommen.
Institut für Physikalische Biologie
Winzige Proteinmoleküle verrichten so gut wie alle Aufgaben in unseren Zellen, können bei Fehlfunktionen aber auch schwere Krankheiten wie Alzheimer oder Krebs auslösen. Um die komplexen dreidimensionalen Strukturen dieser Moleküle in atomarem Detail aufzuklären, nutzen Forscher Verfahren wie die Nuklearmagnetische Resonanzspektroskopie (NMR). Dabei werden die Atomkerne in einem starken Magnetfeld so angeregt, dass sie ein schwach messbares Signal aussenden, aus denen sich die Position jedes einzelnen Atoms im Molekül ableiten lässt.
Das Verfahren ist eigentlich prädestiniert dazu, das Verhalten von Proteinen unter natürlichen Bedingungen zu beobachten. Eine Limitierung führte bis jetzt allerdings dazu, dass NMR-Messungen so ganz "lebensnah" dann doch nicht waren: Denn im Plasma lebender Zellen sind Proteine von zahllosen anderen Molekülen umgeben. Im Spektrometer erzeugen diese jedoch ein Hintergrundrauschen, in dem die feinen Signale des Zielproteins untergehen. Bisher benötigen NMR-Forscher deshalb möglichst pure Proben aus künstlich gereinigtem und hochkonzentriertem Protein.
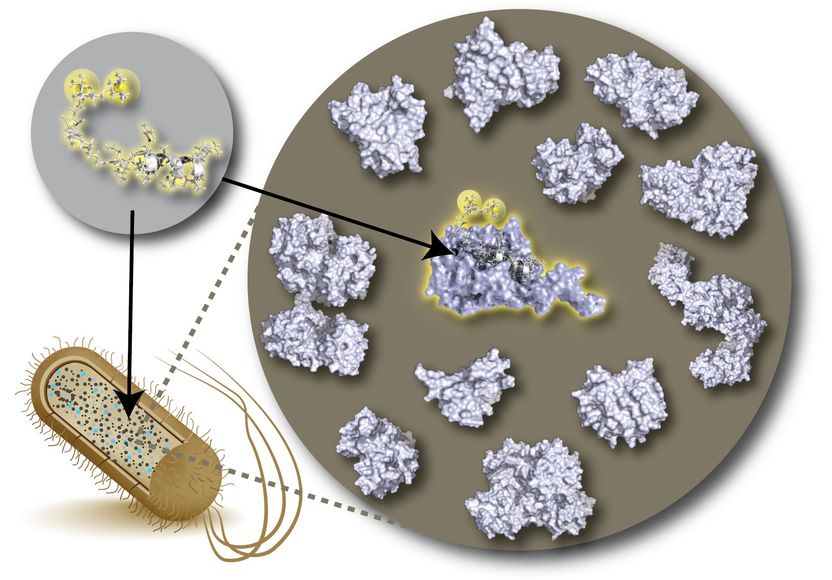
"Die Proteine werden in aller Regel aus ihrer natürlichen Umgebung herausgenommen, was nicht nur ein aufwendiger Prozess ist, sondern auch zu verfälschten Einblicken führen kann", sagt Dr. Manuel Etzkorn. Der Leiter einer Emmy-Noether-Nachwuchsgruppe am Düsseldorfer Institut für Physikalische Biologie arbeitet mit seinem Team an neuen Möglichkeiten, um das Potenzial der NMR-Methode für die biologische Forschung noch besser nutzbar zu machen. Gemeinsam mit Kollegen des Max-Planck-Instituts für Molekulare Physiologie in Dortmund sowie der Universitäten Amsterdam und Sofia haben die Forscher nun ein neues Verfahren entwickelt, das das Problem der verrauschten Signale elegant löst. Es gelang ihnen, das NMR-Signal eines Zielproteins in einer direkt aus einer Bakterienzellkultur gewonnenen Lösung um ein Vielfaches und dazu noch sehr selektiv zu verstärken.
Der Großteil der Forschung fand dabei am Biomolekularen NMR-Zentrum auf dem Gelände des Forschungszentrums Jülich statt, einem der führenden NMR-Zentren in Deutschland. Es wird gemeinsam durch das Jülicher Institute of Complex Systems (ICS-6) und das Düsseldorfer Institut für Physikalische Biologie betrieben. Die Wissenschaftler konnten dort ein neuartiges DNP-verstärktes NMR-Spektrometer einsetzen bei dem das Spektrometer mit einem Mikrowellengenerator verbunden ist. Dieser regt Elektronen von speziellen, von den Forschern zu diesem Zweck modifizierten Molekülen in der Probe an, die ausschließlich an das Zielprotein binden. Die Anregung der Elektronen überträgt sich dabei auf die Atomkerne des Proteins, und führt zu einer enormen Verstärkung des Signals.
Damit lassen sich Daten selbst aus Proben gewinnen, in denen nur geringe Konzentrationen des Proteins in Mischung mit anderen Molekülen vorliegen. Ausgewählte Proteine können also unter Bedingungen, die denen in lebenden Zellen deutlich besser entsprechen, mit atomarer Auflösung untersucht werden. Das vereinfacht nicht nur die Probenherstellung, da aufwändige Schritte zur Protein-Aufreinigung entfallen, sondern ermöglicht auch, die Einflüsse der natürlichen Umgebung besser zu verstehen. "Die zielgerichtete Verstärkung von Proteinsignalen bietet eine Vielzahl spannender Anwendungen im Bereich der zellulären Strukturbiologie und hat das Potenzial, neuartige Einblicke in komplexe biologische Prozesse zu ermöglich", sagt Dr. Etzkorn.
"Die Entwicklung neuer Methoden für die Strukturbiologie ist Schwerpunkt der Forschung des Biomolekularen NMR-Zentrums", erklärt Prof. Dieter Willbold, Leiter des Zentrums und Direktor des Düsseldorfer Instituts für Physikalische Biologie und des Jülicher ICS-6. "Das neue Verfahren ist hierzu ein wichtiger Beitrag, der viele neue Möglichkeiten eröffnet."
Originalveröffentlichung
Thibault Viennet, Aldino Viegas, Arne Kuepper, Sabine Arens, Vladimir Gelev, Ognyan Petrov, Tom N. Grossmann, Henrike Heise, Manuel Etzkorn; "Selective Protein Hyperpolarization in Cell Lysates Using Targeted Dynamic Nuclear Polarization"; Angew Chem Int Ed; 2016 June 28.