Faszinierender Blick in den Zellkern
Vor jeder Zellteilung muss die Erbsubstanz kopiert werden. Die Startpunkte der DNA-Verdoppelung in Zellen von Menschen und Mäusen haben Wissenschaftler um Biologieprofessorin M. Cristina Cardoso von der TU Darmstadt jetzt erstmals unter dem Mikroskop verfolgt. Zusammen mit Forschern um Physikprofessorin Barbara Drossel entwickelten sie ein Computermodell, das die räumliche und zeitliche Verteilung der Replikationsorte beschreibt und erstaunlich gut mit der mikroskopischen Beobachtung übereinstimmt.

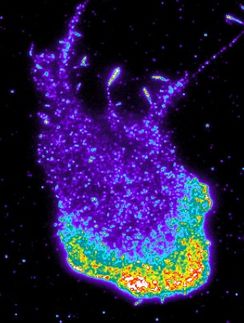
Die Startpunkte der DNA-Verdoppelung haben Wissenschaftlerinnen der TU Darmstadt untersucht.
FG Zellbiologie und Epigenetik
Bei Menschen und anderen höheren Lebewesen befindet sich die Erbsubstanz gut verpackt mit Proteinen im Zellkern. Vor der Zellteilung muss die Zelle daher eine komplexe Entpackungs- und Kopiermaschinerie anwerfen, damit die Tochterzellen dieselbe genetische Ausstattung erhalten wie ihr Vorläufer. Milliarden Basenpaare der DNA müssen dann verdoppelt werden – und zwar möglichst präzise, denn Kopierfehler können zu Krebs und anderen Krankheiten führen. Forscher vermuten seit mehreren Jahrzehnten, dass die Zelle die Replikation des Erbguts an tausenden Stellen auf der DNA gleichzeitig startet. Ansonsten würde der Prozess viel zu lange dauern.
„Mit neuen Methoden der super-auflösenden Mikroskopie ist es uns erstmals gelungen, die vielen Replikationsorte im Zellkern zu visualisieren und quantifizieren“, berichtet M. Cristina Cardoso, Professorin für Zellbiologie und Epigenetik an der TU Darmstadt. Zusammen mit Kollegen aus München und Berlin sowie aus Russland, England und Frankreich gelang Cardoso der faszinierende Blick in den Zellkern, der die Replikationsorte farbig und in 3D zeigt. Die Wissenschaftler haben sowohl menschliche Zellen als auch Mauszellen über einen kompletten Zellzyklus betrachtet. Die super-auflösende Mikroskopie kombinierten sie dafür mit einer computergesteuerten Bildanalyse. Ihre Studie veröffentlichten sie kürzlich in Nature Communications.
In einem zweiten, in derselben Ausgabe von Nature Communications erschienenen Artikel präsentiert Cardosos Gruppe zusammen mit Forschenden um TU-Physikprofessorin Barbara Drossel zudem ein Computermodell, das die räumliche sowie zeitliche Verteilung der Replikationsorte im Zellkern beschreibt und gut mit der mikroskopischen Beobachtung übereinstimmt. Dem Modell zufolge sind die Startpunkte der Replikation anfangs rein zufällig über die Erbsubstanz verstreut. Anschließend breitet sich die DNA-Verdoppelung nach dem Dominoprinzip aus: Neue Replikationsorte entstehen in der Nähe von DNA-Stellen, die bereits kopiert wurden. Diesen Dominoeffekt hat Cardoso schon vor über zehn Jahren postuliert. Jetzt endlich wurde ihre Annahme bestätigt und damit eine grundlegende Frage der DNA-Replikation geklärt.
Originalveröffentlichung
Chagin, V. O. et al.; "4D Visualization of replication foci in mammalian cells corresponding to individual replicons"; Nat. Commun.; 7:11231, (2016)
Löb, D. et al.; "3D replicon distributions arise from stochastic initiation and domino-like DNA replication progression"; Nat. Commun.; 7:11207, (2016)
Meistgelesene News
Originalveröffentlichung
Chagin, V. O. et al.; "4D Visualization of replication foci in mammalian cells corresponding to individual replicons"; Nat. Commun.; 7:11231, (2016)
Löb, D. et al.; "3D replicon distributions arise from stochastic initiation and domino-like DNA replication progression"; Nat. Commun.; 7:11207, (2016)
Themen
Organisationen
Weitere News aus dem Ressort Wissenschaft

Holen Sie sich die Life-Science-Branche in Ihren Posteingang
Mit dem Absenden des Formulars willigen Sie ein, dass Ihnen die LUMITOS AG den oder die oben ausgewählten Newsletter per E-Mail zusendet. Ihre Daten werden nicht an Dritte weitergegeben. Die Speicherung und Verarbeitung Ihrer Daten durch die LUMITOS AG erfolgt auf Basis unserer Datenschutzerklärung. LUMITOS darf Sie zum Zwecke der Werbung oder der Markt- und Meinungsforschung per E-Mail kontaktieren. Ihre Einwilligung können Sie jederzeit ohne Angabe von Gründen gegenüber der LUMITOS AG, Ernst-Augustin-Str. 2, 12489 Berlin oder per E-Mail unter widerruf@lumitos.com mit Wirkung für die Zukunft widerrufen. Zudem ist in jeder E-Mail ein Link zur Abbestellung des entsprechenden Newsletters enthalten.