Studie läutet neues Kapitel der Infektionsforschung ein
Eine Studie an der Universität Würzburg könnte die Infektionsforschung einen großen Schritt voran bringen. Die Wissenschaftler konnten erstmals detailliert zeigen, welche Gene im Verlauf einer Infektion in Erreger und Wirtszelle aktiv werden.
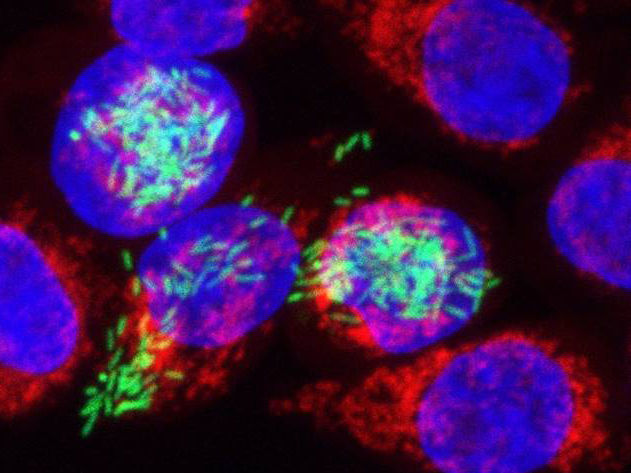
Mit einer neu entwickelten neuen Methode können Forscher nachvollziehen, was bei Infektionen in Krankheitserregern und den von ihnen befallenen Zellen vor sich geht.
IMIB
An der von Professor Jörg Vogel, Direktor des Würzburger Instituts für Molekulare Infektionsbiologie, geleiteten Studie waren auch Forscher aus Leipzig und Köln beteiligt. Die Wissenschaftler untersuchen darin die Abläufe in den ersten Stunden nach einer Salmonellen-Infektion. Diese Bakterien können schwere Lebensmittelvergiftungen auslösen. Sie werden mit der Nahrung aufgenommen und vermehren sich im Darm.
Was dabei in befallenen Wirtszellen genau passiert, ist nur zu einem kleinen Teil bekannt. Die Forscher konnten nun mit einer von ihnen neu entwickelten Methode, der Dualen RNA-Sequenzierung, Licht ins Dunkel bringen. Dazu infizierten sie zunächst Kulturen menschlicher Zellen mit dem Erreger Salmonella Typhimurium. Zu verschiedenen Zeitpunkten nach der Infektion untersuchten sie dann eine bestimmte Molekülgruppe in den befallenen Zellen, die RNA.
RNA kommt in allen Lebewesen vor und übernimmt dort unterschiedliche Aufgaben. Wenn Zellen beispielsweise ein bestimmtes Protein herstellen müssen, benötigen sie dazu eine Bauanleitung, ein Gen. Dieses Gen wird bei Bedarf vielfach kopiert; jede dieser Kopien besteht dabei aus RNA.
Die Wissenschaftler isolierten die komplette RNA aus den befallenen Zellen, also die vom Bakterium und dem Wirt zusammen. Sie konnten so im Detail zeigen, welche seiner rund 5.000 Gene Salmonella zu verschiedenen Phasen der Infektion an- oder abschaltet. Gleichzeitig konnten sie nachweisen, wie die mehr als 40.000 Gene der Wirtszelle auf den Eindringling reagieren.
Kleines Molekül, große Wirkung
Bei ihrer Analyse fiel den Forschern ein bakterielles RNA-Molekül namens PinT auf, von dem Salmonella während einer Infektion mehr als einhundertmal soviel produziert wie normalerweise. Dabei enthält PinT gar keine Protein-Bauanleitung, sondern gehört zu einer speziellen Gruppe bakterieller RNAs, den so genannten sRNAs.
sRNAs sind auffällig kleine RNA-Moleküle (das „s“ steht für „small“), die für das Feintuning der Genaktivität zuständig sind: Sie sorgen beispielsweise dafür, dass die kopierten Protein-Bauanleitungen schnell wieder vernichtet werden können. Ihre Rolle während Infektionen war bislang weitgehend unbekannt. „Wir haben eine Salmonella-Mutante hergestellt, die kein PinT produzieren kann“, erklärt Dr. Alexander Westermann vom Würzburger Institut für Molekulare Infektionsbiologie. „Dann haben wir untersucht, wie sich diese Mutante bei einer Infektion verhält.“
Das Ergebnis war frappierend: Das Mini-Molekül beeinflusst augenscheinlich eine ganze Latte bakterieller Gene, vor allem so genannte Virulenzfaktoren. Diese entscheiden darüber, wie aggressiv sich das Bakterium bei der Infektion durchsetzt. So gibt es beispielsweise Virulenz-Gene, die für die Invasion des Bakteriums in die Wirtszelle nötig sind. Weil es viel Energie kostet, produzieren Bakterien ihre Virulenzfaktoren nur dann, wenn sie sie wirklich benötigen. Auch minimieren die Erreger damit ihr Risiko, vorzeitig vom Immunsystem entdeckt zu werden.
Der Taktstock für das richtige Timing
PinT ist der Taktstock, der dabei für das richtige Timing sorgt. Ohne das Mini-Molekül kommt die fein orchestrierte Abstimmung der Virulenzfaktoren durcheinander. Diese Verschiebung hat wiederum massive Auswirkungen auf die Wirtszelle. „In unserer Studie waren fast ein Zehntel aller Wirtsgene betroffen, die nun – im Vergleich zu einer normalen Infektion – entweder vermehrt oder seltener abgelesen wurden“, erläutert Westermann. „So wurden bestimmte Immungene deutlich stärker aktiviert als normalerweise.“
Die simultane RNA-Sequenzierung von Krankheitserreger und Wirtszelle erlaubt es erstmals, derart komplexe Kausalketten im zeitlichen Verlauf einer Infektion nachzuvollziehen. „Bei vergleichsweise geringem Aufwand verspricht die Methode daher einen enormen Erkenntnisgewinn“, erklärt Professor Jörg Vogel. „Bislang war es bei vielen bakteriellen Genen kaum möglich, ihren Beitrag zur Infektion aufzuklären – dazu fehlten einfach die passenden Methoden. Jetzt haben wir endlich ein sensitives Werkzeug, um diese Gene zu untersuchen. Die Duale RNA-Sequenzierung eröffnet daher der Infektionsforschung eine neue Dimension.“ Die anfallende Datenmenge ist allerdings enorm. Bioinformatiker der Universitäten Würzburg und Leipzig entwickelten eigens für die Studie neue Algorithmen, mit denen sich die RNA-Sequenzen automatisiert und in ausreichender Geschwindigkeit verarbeiten lassen.
Originalveröffentlichung
Alexander J. Westermann, Konrad U. Förstner, Fabian Amman, Lars Barquist, Yanjie Chao, Leon N. Schulte, Lydia Müller, Richard Reinhardt, Peter F. Stadler & Jörg Vogel; "Dual RNA-seq unveils noncoding RNA functions in host–pathogen interactions"; Nature