Zelltracking in Mikroskopiebildern
Heidelberger Wissenschaftler entwickeln hochleistungsfähiges automatisches Bildanalyseverfahren
Um biologische Partikel in einer Zelle in ihrer Bewegung verfolgen zu können, haben Wissenschaftler der Universität Heidelberg und des Deutschen Krebsforschungszentrums ein hochleistungsfähiges Analyseverfahren für Lebendzell-Mikroskopiebilder entwickelt. Dieses sogenannte probabilistische Partikel Tracking-Verfahren, das automatisch und computergestützt arbeitet, kann für zeitaufgelöste zwei- und dreidimensionale Mikroskopiebilddaten eingesetzt werden. In einem internationalen Leistungsvergleich unterschiedlicher Bildanalysemethoden konnte das Heidelberger Verfahren das beste Gesamtergebnis erzielen. Die Wettbewerbsergebnisse wurden jetzt in der Fachzeitschrift „Nature Methods“ publiziert.
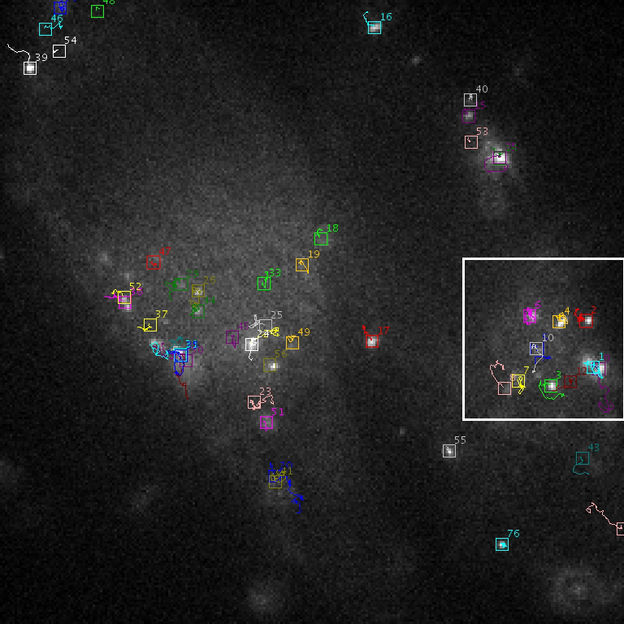
Tracking-Ergebnis für-Virus Partikel
Ruprecht-Karls-Universität Heidelberg

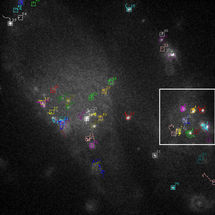
Tracking-Ergebnis für Virus-Partikel – vergrößerter Auschnitt
Ruprecht-Karls-Universität Heidelberg

Die Frage, wie sich die Bewegung von biologischen Partikeln, etwa Viren, Zellvesikeln oder Zellrezeptoren, automatisch verfolgen lässt, ist von zentraler Bedeutung in biomedizinischen Anwendungen für die quantitative Analyse von intrazellulären dynamischen Prozessen. Eine manuelle Auswertung von zeitaufgelösten Mikroskopiebildern, die hunderte oder tausende sich bewegender Objekte zeigen, ist dabei nicht durchführbar. Daher wurden in den vergangenen Jahren verstärkt automatische Bildanalyseverfahren zum sogenannten Partikel Tracking entwickelt. Sie ermitteln computergestützt die Partikelpositionen über die Zeit hinweg. Um die Leistungsfähigkeit dieser Methoden objektiv vergleichend zu untersuchen, wurde 2012 erstmals ein internationaler Wettbewerb durchgeführt.
Unter den insgesamt 14 teilnehmenden Forschungsgruppen dieser „Partikel Tracking Challenge“ waren mit Dr. William J. Godinez und Privatdozent Dr. Karl Rohr auch Wissenschaftler der Universität Heidelberg und des Deutschen Krebsforschungszentrums (DKFZ) vertreten. Im Rahmen des Wettbewerbs wurden die unterschiedlichen Bildanalyseverfahren auf ein großes Spektrum an zwei- und dreidimensionalen Bilddaten angewendet, wobei die Leistungsfähigkeit durch verschiedene Maße quantifiziert wurde. Für jede Kategorie der verwendeten Daten wurden die jeweils drei besten Verfahren ermittelt. Die Heidelberger Wissenschaftler konnten mit insgesamt 150 „Top 3-Rängen“ das beste Gesamtergebnis erzielen.
Das von Dr. Godinez und Dr. Rohr entwickelte Partikel Tracking-Verfahren zeichnet sich dadurch aus, dass Unsicherheiten in den Bilddaten, beispielsweise aufgrund von Bildrauschen, und Wissen über den Anwendungsbereich durch eine mathematisch fundierte Methode der Wahrscheinlichkeitstheorie berücksichtigt werden. „Im Vergleich zu deterministischen Methoden lässt sich mit unserem probabilistischen Verfahren eine hohe Genauigkeit insbesondere bei schwierigen Bilddaten mit einer großen Objektanzahl, einer hohen Objektdichte und starkem Rauschen erzielen“, sagt Dr. Rohr. Dabei ist es möglich, Bewegungspfade von Objekten zu bestimmen und relevante Parameter, etwa Geschwindigkeit, Weglänge, Bewegungstyp oder Objektgröße, zu quantifizieren. Zusätzlich werden wichtige dynamische Ereignisse wie beispielsweise Virus-Zell-Fusionen automatisch erkannt.