Weltkarten des Lebens in Echtzeit
Max-Planck-Forscher in Dresden verarbeiten Mikroskopie-Bilder in Echtzeit und projizieren Zebrafische auf Weltkarten
Heutige Mikroskope liefern immer schneller immer höher aufgelöste Bilder. Das bringt ein Problem mit sich: Bei jedem Experiment häufen sich Unmengen von Daten an, die irgendwo gespeichert und später ausgewertet werden müssen – bei den neuesten Geräten können es täglich schnell 25 Terabyte sein. Forscher des Max-Planck-Instituts für molekulare Zellbiologie und Genetik in Dresden haben ihre Mikroskope so weiterentwickelt, dass diese die Bilder in Echtzeit verarbeiten und nur noch das Ergebnis speichern – das reduziert die Datenmengen ungemein und vereinfacht die weitere Auswertung der Bilder.

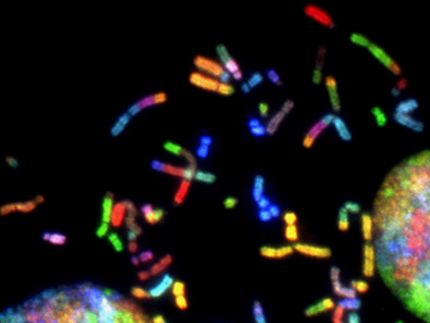
In dem neuartigen Mikroskop werden alle fluoreszierenden Zellen des kugelförmigen Zebrafischembryos auf eine Karte projiziert - wie Länder auf einer Weltkarte. Die Zellen werden automatisch erkannt und farblich hervorgehoben (obere Hälfte). Ihre Wege durch den Embryo lassen sich über mehrere Stunden verfolgen (untere Hälfte) und liefern so wichtige Erkenntnisse zum Verständnis der Organentwicklung.
© MPI f. molekulare Zellbiologie u. Genetik
„Mikroskope sind leider erst mal dumm“, sagt Jan Huisken vom Planck-Institut für molekulare Zellbiologie und Genetik. Stur nehmen die Geräte große rechteckige Bilder auf, unabhängig davon, ob und wo die Bilder relevante Informationen enthalten. Am Ende entsteht so zwar ein Abbild der Probe. Aber ein Großteil dieses dreidimensionalen Datenblocks ist unnötige Information, die die Datenmengen aufbläht und gigantische Computerleistung in Anspruch nimmt.
Benjamin Schmid in Huiskens Team hat genau hier angesetzt und dem Mikroskop vor dem Experiment beigebracht, was wichtig ist und was nicht: Nun rastert es nur die relevanten Daten ab, in diesem Fall die Zellen auf der Oberfläche eines sich entwickelnden kugelförmigen Zebrafisch-Embryos. In Echtzeit werden die Daten verarbeitet und ihr Volumen damit extrem reduziert.
Gespeichert wird also nun das Ergebnis, nicht mehr die Rohdaten. Die Forscher konnten damit die angehäufte Datenmenge drastisch senken: Sie erreichten eine 240-fache Reduktion und schafften so den entscheidenden Schritt von Terabytes zu Gigabytes. „So kann man nun mehrere Experimente parallel machen – das ist es, was Biologen jetzt dringend brauchen und was die Systembiologie vorantreiben wird“, so Huisken. In seiner Arbeitsgruppe wurde die Verteilung und Bewegung von Zellen parallel in zwölf wachsenden Zebrafisch-Embryonen mit der sogenannten Lichtscheibenmikroksopie (SPIM) beobachtet. Dazu wird Laserlicht zu einer hauchdünnen Ebene gebündelt, mit der man die Probe durchleuchtet. Innerhalb von nur zehn Sekunden wird aus den Daten der mikroskopierten Embryo-Kugel eine Karte aller markierten Zellen erstellt – mittels Projektion wird aus dem Globus eine Weltkarte des Lebens. Die Information aus allen zwölf Experimenten wird schließlich in einer Darstellung der Bewegungsabläufe in allen Embryonen verschmolzen.
Originalveröffentlichung
Benjamin Schmid, Gopi Shah, Nico Scherf, Michael Weber, Konstantin Thierbach, Citlali Pérez Campos, Ingo Roeder, Pia Aanstad, Jan Huisken High-speed panoramic light-sheet microscopy reveals global endodermal cell dynamics Nature Communications, 25. Juli 2013
Meistgelesene News
Originalveröffentlichung
Benjamin Schmid, Gopi Shah, Nico Scherf, Michael Weber, Konstantin Thierbach, Citlali Pérez Campos, Ingo Roeder, Pia Aanstad, Jan Huisken High-speed panoramic light-sheet microscopy reveals global endodermal cell dynamics Nature Communications, 25. Juli 2013
Organisationen
Weitere News aus dem Ressort Wissenschaft
Diese Produkte könnten Sie interessieren

alpha300 R von WITec
3D Raman Mikroskope mit unerreichter Geschwindigkeit, Sensitivität und Auflösung
Jedes chemische Detail der Probe wird sichtbar

JEOL CRYO ARM von JEOL
Kryo-TEM: Schnelle und stabile Datenerfassung für Bioproben
Effizienzsteigerung in der Strukturbiologie mit automatisiertem Probenladesystem

FLUOVIEW FV4000 von EVIDENT
Revolutionäre Bildgebung mit FLUOVIEW FV4000: Konfokales Laser-Scanning
Nutzen Sie KI-gestützte Bildverarbeitung und innovative Detektortechnologie

Holen Sie sich die Life-Science-Branche in Ihren Posteingang
Mit dem Absenden des Formulars willigen Sie ein, dass Ihnen die LUMITOS AG den oder die oben ausgewählten Newsletter per E-Mail zusendet. Ihre Daten werden nicht an Dritte weitergegeben. Die Speicherung und Verarbeitung Ihrer Daten durch die LUMITOS AG erfolgt auf Basis unserer Datenschutzerklärung. LUMITOS darf Sie zum Zwecke der Werbung oder der Markt- und Meinungsforschung per E-Mail kontaktieren. Ihre Einwilligung können Sie jederzeit ohne Angabe von Gründen gegenüber der LUMITOS AG, Ernst-Augustin-Str. 2, 12489 Berlin oder per E-Mail unter widerruf@lumitos.com mit Wirkung für die Zukunft widerrufen. Zudem ist in jeder E-Mail ein Link zur Abbestellung des entsprechenden Newsletters enthalten.
Meistgelesene News
Weitere News von unseren anderen Portalen
Zuletzt betrachtete Inhalte
Alt aber oho: Regeneration von Nierenschäden bei erwachsenen Zebrafischen
Endosymbiontentheorie
Magisches_Denken

Genetiker enthüllen, wie Mutation Kinderkrebs verursacht - Verwenden Sie das Medikament, um seine Wirkung umzukehren
Tumornekrosefaktor
Johann_Friedrich_Eschscholtz