Personalisierte Medizin: Fortschritte in der Erkennung genomischer Veränderungen in klonalen Tumorpopulationen
Tumoren entstehen meistens über längere Zeiträume und durchleben dabei eine Evolution. Als Folge davon bestehen viele aus separaten, sogenannten klonalen Zellenpopulationen, die über verschiedene irreversible genomische Veränderungen (Aberrationen) verfügen und deshalb unterschiedlich auf Medikamente reagieren. Forschende der Universität Basel und des Translational Genomics Research Institute (TGen, Arizona) haben nun ein Verfahren entwickelt, das die verschiedenen klonalen Populationen im Patientengewebe erkennt, trennt und entschlüsselt. Das neue Verfahren verbessert das Verständnis über das Ansprechen der Tumorpopulationen auf Krebsmedikamente.
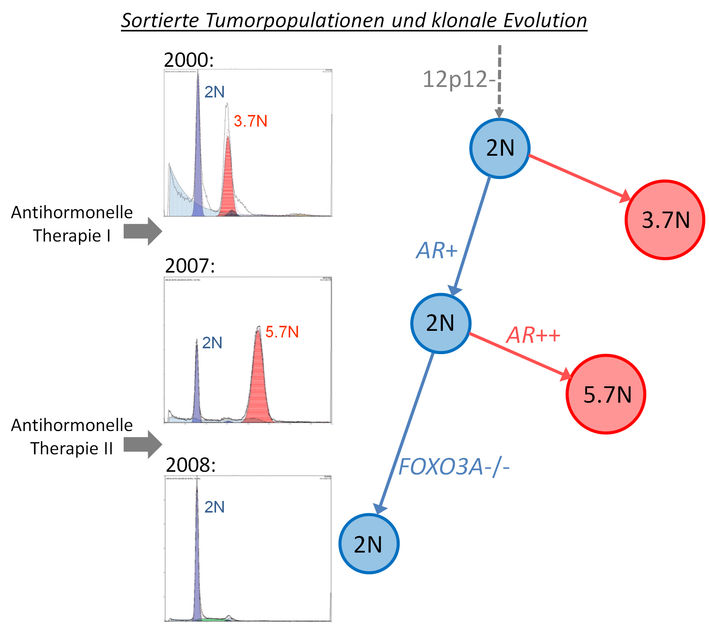
Untersuchung der genomischen Evolution eines Prostatakarzinoms über einen Zeitraum von acht Jahren. Die diploiden Tumorpopulationen (2N) überstanden die antihormonelle Behandlung und entwickelten sich zu neuen, therapieresistenten Populationen. Die aneuploiden Populationen (3.7N, 5.7N) hingegen wurden durch die Therapie ausgelöscht.
Bei der Entstehung von Tumoren geschehen genomische Veränderungen wie zum Beispiel eine Vermehrung, ein Verlust, eine Ortsveränderung oder eine Mutation von genetischem Material. Diese Aberrationen sind spezifisch für den Tumor und können das Tumorwachstum fördern. Die Detektion dieser Veränderungen in Tumoren ist zu einem wichtigen Bestandteil der personalisierten Medizin geworden, da die daraus resultierenden Proteinprodukte Angriffsziele von zielgerichteten Krebsmedikamenten sein können. Die Suche nach neuen Aberrationen wird aber oft durch die Heterogenität des Tumors stark beeinträchtigt, da Tumoren sich über längere Zeit entwickeln und als Folge davon aus klonalen Populationen bestehen, die über verschiedene genomische Aberrationen verfügen und deshalb unterschiedlich auf Medikamente reagieren können. Einem Forscherteam um Prof. Dr. Lukas Bubendorf und Dr. Christian Ruiz vom Institut für Pathologie der Universität Basel ist es nun in Zusammenarbeit mit Kollegen des Translational Genomics Research Institute (TGen, Arizona) gelungen, ein Verfahren zu entwickeln, um die verschiedenen klonalen Populationen in Patientengewebe zu detektieren, zu trennen und mittels Hochdurchsatztechnologien genomisch mit hoher Auflösung zu entschlüsseln.
Das neue Verfahren wurde in den letzten drei Jahren entwickelt. Dabei wurde archiviertes Tumorgewebe von Patienten aufgrund ihres DNA-Gehaltes sortiert und die sortierten Tumorpopulationen mittels Array-CGH und neuen Sequenziertechnologien untersucht. Die Anwendung des neuen Verfahrens an einer Gruppe von Pankreas- und Prostatakarzinomen hat gezeigt, dass die Tumoren in vielen Fällen aus mehreren klonalen Populationen mit unterschiedlichen klinisch-relevanten Aberrationen zusammengesetzt sind. Die Forschenden verfolgten außerdem die Entwicklung eines Prostatakarzinoms über einen Zeitraum von mehr als acht Jahren. Damit konnte zum ersten Mal gezeigt werden, wie die verschiedenen klonalen Tumorpopulationen auf den Selektionsdruck der Therapie reagierten: Nur Tumorpopulationen mit einem wenig verändertem Genom waren in der Lage, die Therapie zu überleben, indem sie therapieresistente Aberrationen akquirierten und neue spezialisierte Tumorpopulationen generierten.
Die Forschenden sind überzeugt, dass die neue Technologie helfen wird, das Ansprechen auf Krebsmedikamente besser zu verstehen. Das Verfahren wird nun in Basel im Rahmen von Forschungsprojekten weiter entwickelt und bei verschiedenen Tumortypen angewandt.
Originalveröffentlichung
Christian Ruiz et al.; Advancing a clinically relevant perspective of the clonal nature of cancer; PNAS, publishes online before print, July 5, 2011






















































