Auf der Spur des epigenetischen Codes
Testsystem an Drosophila soll den Schlüssel liefern
Lange Zeit galt die Erbsubstanz DNA als alleiniger Informationsträger der Vererbung. Ihren Verpackungsproteinen, den Histonen, wurde nur eine strukturelle Bedeutung zugeschrieben. Durch chemische Änderungen an der DNA oder den Histonen kann jedoch zusätzliche Information gespeichert und an nachfolgende Generationen weitergegeben werden. Wissenschaftlern vom Max-Planck-Institut für Biophysikalische Chemie in Göttingen ist es gelungen, ein experimentelles System zu etablieren, um die Funktion solcher chemischen Histon-Modifikationen und deren Einfluss auf den Organismus auszutesten. Chemische Änderungen an den Histonen stellen möglicherweise einen "epigenetischen Histon-Code" dar, der den genetischen Code ergänzt und darüber entscheidet, ob die Information bestimmter DNA-Regionen genutzt oder unterdrückt wird.
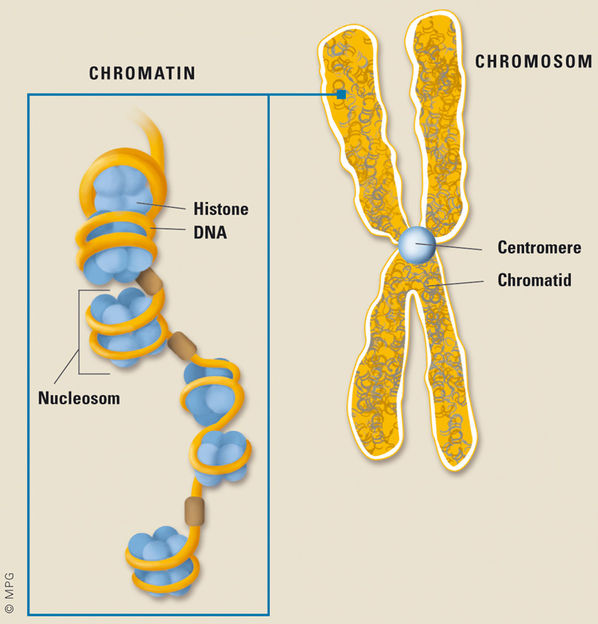
Nur durch geschickte Verpackung ist es überhaupt möglich, den zwei Meter langen DNA-Faden im Zellkern unterzubringen. Dazu wird der Faden wie bei einer Spule um Millionen von Proteinen, die Histone, herumgewickelt und diese dann perlschnurartig aneinander gereiht.
Max-Planck-Gesellschaft
Wie bekommt man einen zwei Meter langen DNA-Faden in den Zellkern? Indem man ihn zusammenknäuelt! Dabei wird die DNA um Proteine, die Histone, gewickelt und somit bis um das 50.000-fache verkürzt. Weitere Proteine lagern sich dann an, um das Chromatin und letztlich die Chromosomen zu bilden. Sie sind das Ergebnis eines genialen Verpackungstricks. Die insgesamt fünf unterschiedlichen Histontypen (H1, H2A, H2B, H3 und H4) haben aber noch weitere Aufgaben - und das macht sie so interessant: Histone können an verschiedenen Stellen mit kleinen chemischen Anhängseln wie Acetyl-, Methyl- oder Phosphatgruppen versehen werden. Diese bewirken zum Beispiel eine Öffnung des Chromatins und ermöglichen erst das Ablesen der genetischen Information. Umgekehrt können durch andere Veränderungen, wie die Bindung von Proteinen, bestimmte Bereiche des DNA-Moleküls stillgelegt werden, sodass sie nicht mehr abgelesen werden: "Gen-Silencing" nennen die Wissenschaftler das. "Die Histon-Modifikationen greifen auf diese Weise in die Aktivitätskontrolle der Gene ein und ergänzen damit den genetischen Code", erklärt Herbert Jäckle, Direktor am Max-Planck-Institut für biophysikalische Chemie in Göttingen.
Dieses Modifikationsmuster der Histone wird bei jeder Zellteilung an die Tochterzellen vererbt. Die Wissenschaftler gehen davon aus, dass diese epigenetische Vererbung durch einen zell- oder organspezifischen "Histon-Code", bestimmt wird: "Er entscheidet darüber, ob die Zellmaschinerie zu den DNA-codierten Genen Zugang hat oder ob der Zugang gesperrt wird", so Jäckle. Diesen Code würden die Wissenschaftler nur zu gerne knacken. Doch dabei gibt es ein nicht unerhebliches Hindernis: Für die Produktion der Histone sind im Genom höherer Organismen bis zu jeweils hunderte Genkopien abgelegt. Bislang schien es daher unmöglich, diese Genkopien auszuschalten und durch gentechnisch veränderte Histon-Varianten zu ersetzen. Erst das würde den Forschern ermöglichen, ein Testsystem zu etablieren: Denn wenn diesen Varianten bestimmte Andockstellen, z.B. für chemische Gruppen fehlen, ließen sich bestimmte Modifikationen an den Histonen verhindern und untersuchen, inwieweit das Fehlen dieser Modifikationen zu diagnostizierbaren Störungen im Organismus führt.
Doch nun haben die Göttinger Max-Planck-Forscher eine neue Methode entwickelt, um die Funktion aller Histon-Modifikationen zu untersuchen. Die Zellbiologen entfernten alle Histon-Gene aus dem Genom der Fruchtfliege Drosophila melanogaster, was bewirkt, dass sich Zellen nicht mehr weiterteilen. Ihr Genom wird zwar noch wie bei einer normalen Zellteilung durch DNA-Synthese verdoppelt, danach allerdings bleibt die Zelle ungeteilt im Teilungszyklus stehen und der Organismus stirbt. Mit steigender Zahl an Kopien von den jeweils vier Histon-Genen normalisiert sich die Situation zunehmend: "Fliegen mit zwölf Kopien des Histon-Gen-Clusters überleben schließlich und sind fortpflanzungsfähig", erzählt Jäckles Mitarbeiter Alf Herzig, der das Projekt leitet.
Dass mehrere Kopien der Histon-Gene, notwendig sind, damit der Organismus überlebt, war bei mehrzelligen Organismen schon allgemein festgestellt, worden. Die Ergebnisse liefern darüber hinaus Hinweise dafür, dass die Zelle während der Teilung erkennt, dass Histone fehlen und daraufhin die Teilung der Zelle unterbleibt, obwohl die DNA - wie vor jeder Zellteilung - bereits verdoppelt wurde. "Offenbar gibt es also Kommunikationswege zwischen der Histon-Produktion und der Zellteilungsmaschinerie", sagt Günesdogan, Doktorand in der Abteilung. Das Entscheidende aber ist: Die Forscher haben nun ein Analyse-System zur Verfügung, in das Histon-Varianten eingeschleust werden können, um schrittweise die Funktion der Histonmodifikationen, und damit letztlich den Histon-Code im Organismus experimentell zu überprüfen. Mal sehen, wann der Code geknackt wird.
Originalveröffentlichung: Ufuk Günesdogan, Herbert Jäckle & Alf Herzig; "A genetic system to assess in vivo the functions of histones and histone modifications in higher eukaryotes"; EMBO reports (2010) 11, 772 - 776; online veröffentlicht am 1. Oktober 2010