Was löst den programmierten Zelltodmechanismus aus?
Entwicklung und Kombination verschiedener mikroskopischer und spektroskopischer Techniken
Wie lassen sich molekulare Strukturen analysieren, wenn die Auflösung der Techniken eigentlich nicht ausreicht? Forschende der Heinrich-Heine-Universität Düsseldorf (HHU) aus der Physik, Chemie und Medizin haben verschiedene mikroskopische und spektroskopische Techniken kombiniert und weiterentwickelt, um eine für den „programmierten Zelltod“ wichtige Proteinanordnung in der Zellmembran zu untersuchen. Unter welchen Umständen der für den Zelltod verantwortliche CD95-Rezeptor reagiert, beschreiben sie in der Fachzeitschrift Science Advances.
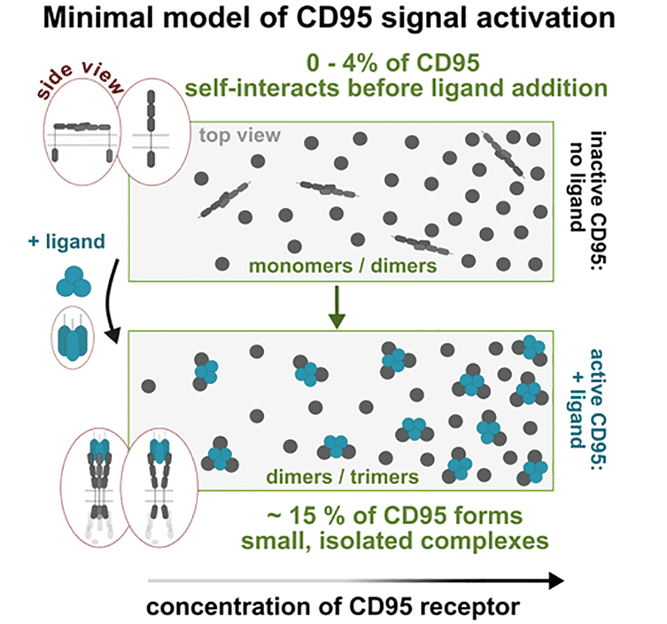
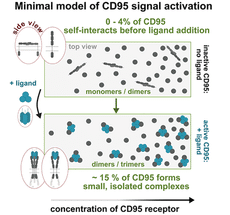
Aus den Messungen resultierendes Modell der Signalaktivierung: Der CD95-Rezeptor verändert nach Wechselwirkung mit dem CD95-Liganden seine Anordnung und Konformation. Es bilden sich kleine Proteinkomplexe.
HHU / Cornelia Monzel
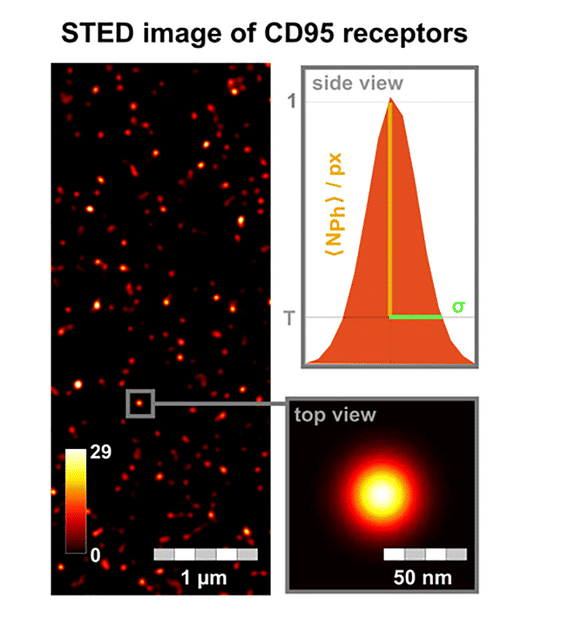
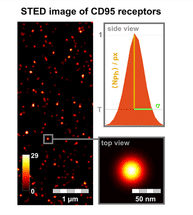
STED-Aufnahme von CD95-Rezeptoren auf der Zellmembran. Von jedem Leuchtpunkt werden die Helligkeit (Anzahl Photonen pro Pixel) und die Standardabweichung der Punktgröße (σ) analysiert. Neben der Anwendung der STED-Mikroskopie wurden die FRET-Einzelmolekülspektroskopie und eine Analyse von Photobleichschritten weiterentwickelt.
HHU / Cornelia Monzel
In biologischen Zellen sind die allermeisten funktionalen Strukturen aus Eiweißmolekülen, sogenannten Proteinen, aufgebaut. Um die Funktion von Proteinen zu verstehen, muss zum einen deren dreidimensionale Struktur bekannt sein. Wichtig ist ferner, ihre räumliche Anordnung und die dynamische Interaktion mit sich selbst und Proteinen in ihrer unmittelbaren und weiteren Umgebung zu erfassen. Denn diese Faktoren lösen häufig erst ihre Funktion aus.
Diese Informationen zu erlangen ist sehr komplex und ihre Messung stößt in Teilen an die Grenzen des technisch Machbaren. So sind die relevanten Proteinstrukturen oft nur einen bis 100 Nanometer (nm; ein Milliardstel Teil eines Meters) klein. Sie können deshalb nicht mit herkömmlichen optischen Mikroskopen aufgelöst werden. Auch moderne, hochauflösende Techniken wie die 2014 mit dem Chemienobelpreis ausgezeichnete „Stimulated Emission Depletion-Mikroskopie“ (kurz STED-Mikroskopie) reichen hier alleine nicht aus.
Ein Forschungsteam um Prof. Dr. Cornelia Monzel (Institut für Experimentelle Medizinische Physik) und Prof. Dr. Claus A. M. Seidel (Lehrstuhl für Molekulare Physikalische Chemie) hat verschiedene Techniken verknüpft, um die Wirkungsweise des sogenannten CD95-Rezeptors auf der Oberfläche von Zellmembranen zu verstehen. Es ging darum, die molekulare Anordnung und Interaktion des Rezeptors zu bestimmen, die insgesamt benötigt werden, um ein Zellsignal auszulösen.
Der CD95-Rezeptor hat für die Zellen eine elementare Bedeutung: Über dieses Protein wird das Signal eingeleitet, dass letztlich zum kontrollierten oder programmierten Zelltod, zur „Apoptose“, führt. Dazu Prof. Monzel, eine der beiden Korrespondenzautorinnen der Studie: „Dieser Signalweg ist in unserem alltäglichen Dasein dafür verantwortlich, dass wir nicht ständig wachsen, sondern dass gleich viele Zelle absterben, wie neue entstehen. Er spielt darüber hinaus bei vielen anderen Prozessen wie der Entwicklung eines Lebewesens, der Wundheilung und der Krebstherapie eine wichtige Rolle.“
Dieser Rezeptor ist nur rund 20 nm groß. Dr. Nina Bartels, eine der beiden Erstautorinnen der jetzt bei Science Advances erschienenen Studie: „Da wir diese kleinen Strukturen mit hochauflösenden Techniken nicht direkt und eindeutig abbilden können, mussten wir verschiedene mikroskopische und spektroskopische Techniken weiterentwickeln und miteinander kombinieren.“
Neben der STED-Mikroskopie, die Strukturen bis hinunter zu 40nm auflösen kann, haben die Düsseldorfer Forschenden die sogenannte FRET-Einzelmolekülspektroskopie und eine Analyse von Photobleichschritten weiterentwickelt. Im Ergebnis können sie bis hinab zu Abständen von einigen Nanometern die Anzahl, Verteilung und Interaktion des Rezeptors auf der Membran messen.
Dr. Nicolaas van der Voort, der zweite Erstautor der Studie: „Wir konnten aus unseren Messungen ein eindeutiges Modell entwickeln, welche Prozesse zur Aktivierung des CD95-Rezeptors und somit des kontrollierten Zelltodes notwendig sind. Damit das Zelltodsignal angeschaltet wird, müssen sich von allen CD95-Rezeptoren auf der Zellmembran nur 15 Prozent je zu zweit oder zu dritt um ein weiteres Protein, den sogenannten CD95-Liganden, anlagern.“
Prof. Seidel, der ebenfalls Korrespondenzautor der Studie ist, weist auf die weiterreichenden Aspekte der Forschungsergebnisse hin: „Neben den Erkenntnissen zur Aktivierung des kontrollierten Zelltods beschreibt diese Arbeit wichtige mikroskopisch-spektroskopische Weiterentwicklungen, die sich auf viele weitere biologisch-medizinische Fragestellungen übertragen lassen.“
Originalveröffentlichung
Nina Bartels, Nicolaas T. M. van der Voort, Oleg Opanasyuk, Suren Felekyan, Annemarie Greife, Xiaoyue Shang, Arthur Bister, Constanze Wiek, Claus A. M. Seidel, Cornelia Monzel; "Advanced multiparametric image spectroscopy and super-resolution microscopy reveal a minimal model of CD95 signal initiation"; Science Advances, Volume 10