Schneller Nachweis von antibiotikaresistenten Bakterien
Eine papierbasierte Plattform, die von Forschern des Indian Institute of Science (IISc) und des Jawaharlal Nehru Centre for Advanced Scientific Research (JNCASR) entwickelt wurde, könnte dazu beitragen, das Vorhandensein von antibiotikaresistenten, krankheitsverursachenden Bakterien schnell zu erkennen.
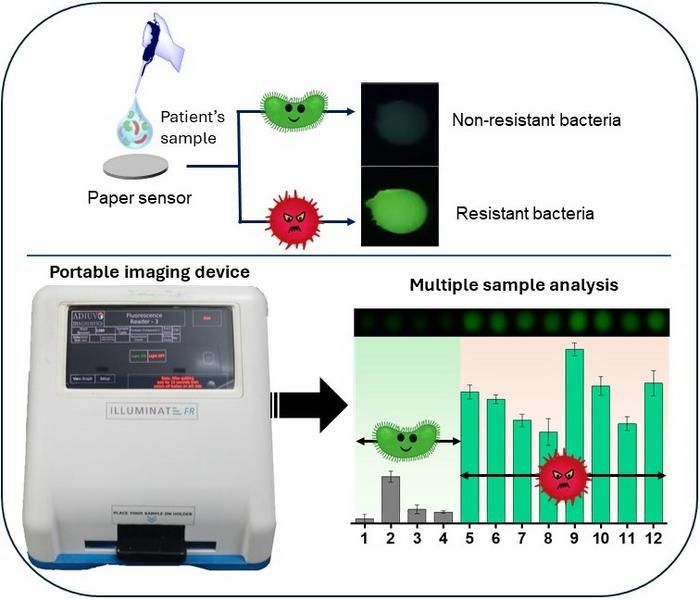
Schematische Darstellung des Nachweises/der Differenzierung von Bakterien mit Antibiotikaresistenz
Arnab Dutta
Eine der größten Herausforderungen für die Menschheit ist das Aufkommen von krankheitsverursachenden Bakterien, die gegen Antibiotika resistent sind. Ihr Aufkommen wurde durch den Missbrauch und die Überbeanspruchung von Antibiotika begünstigt.
Eine Handvoll solcher Bakterien - darunter E. coli und Staphylococcus aureus - haben bereits über eine Million Todesfälle verursacht, und diese Zahl wird nach Angaben der Weltgesundheitsorganisation in den kommenden Jahren voraussichtlich noch steigen. Eine rechtzeitige Diagnose kann die Effizienz der Behandlung verbessern.
"In der Regel stellt der Arzt eine Diagnose und gibt dem Patienten ein Medikament. Der Patient nimmt sie dann 2 bis 3 Tage lang ein, bevor er merkt, dass die Medikamente nicht wirken, und wieder zum Arzt geht. Selbst die Diagnose, dass es sich um antibiotikaresistente Bakterien handelt, die durch Blut- oder Urintests festgestellt wird, braucht Zeit. Wir wollten diese Zeit bis zur Diagnose verkürzen", sagt Uday Maitra, Professor an der Abteilung für organische Chemie des IISc.
In einer in der Zeitschrift ACS Sensors veröffentlichten Arbeit haben Maitras Labor und seine Mitarbeiter diese Herausforderung angenommen. Sie haben ein Schnelldiagnoseprotokoll entwickelt, das eine auf Leuchtpapier basierende Plattform zum Nachweis von antibiotikaresistenten Bakterien verwendet.
Es gibt verschiedene Möglichkeiten, wie ein Bakterium gegen Antibiotika resistent wird. In einem Fall entwickelt sich das Bakterium weiter und kann das Medikament erkennen und aus seiner Zelle ausschleusen. In einem anderen Fall produziert das Bakterium ein Enzym namens β-Lactamase, das den β-Lactamring hydrolysiert - eine wichtige Strukturkomponente gängiger Antibiotika wie Penicillin und Carbapenem - und damit das Medikament unwirksam macht.
Bei dem vom IISc- und JNCASR-Team entwickelten Ansatz wird Biphenyl-4-Carbonsäure (BCA) in eine supramolekulare Hydrogelmatrix eingebaut, die Terbiumcholat (TbCh) enthält. Dieses Hydrogel emittiert normalerweise grüne Fluoreszenz, wenn es mit UV-Licht bestrahlt wird.
"Im Labor haben wir ein Enzym-Substrat synthetisiert, indem wir BCA an den zyklischen [β-Lactam]-Ring gebunden haben, der Teil des Antibiotikums ist. Wenn man dies mit TbCh-Hydrogel mischt, gibt es keine grüne Emission, da der Sensibilisator 'maskiert' wird", erklärt Arnab Dutta, Doktorand in der Abteilung für organische Chemie am IISc und Hauptautor der Arbeit. "In Anwesenheit von β-Lactamase-Enzym erzeugt das Gel eine grüne Emission. Das β-Lactamase-Enzym in den Bakterien ist dasjenige, das den Wirkstoff aufschneidet, zerstört und den Sensibilisator BCA unschädlich macht. Das Vorhandensein von β-Laktamase wird also durch grüne Emission signalisiert." Die Lumineszenz signalisiert das Vorhandensein von antibiotikaresistenten Bakterien, und die Intensität der Lumineszenz zeigt die bakterielle Belastung an. Bei nicht-resistenten Bakterien wurde festgestellt, dass die grüne Intensität extrem niedrig ist, was ihre Unterscheidung von resistenten Bakterien erleichtert.
Der nächste Schritt bestand darin, einen Weg zu finden, die Technologie kostengünstig zu machen. Die derzeit verwendeten Diagnoseinstrumente sind kostspielig, was den Preis für die Tests in die Höhe treibt.
Das Team arbeitete mit einem in Tamil Nadu ansässigen Unternehmen namens Adiuvo Diagnostics zusammen, um ein maßgeschneidertes, tragbares Miniatur-Bildgebungsgerät namens Illuminate Fluorescence Reader zu entwickeln. Durch die Infusion des Hydrogels in ein Blatt Papier als Medium konnten die Kosten erheblich gesenkt werden. Das Gerät ist mit verschiedenen LEDs ausgestattet, die je nach Bedarf UV-Strahlung abgeben. Die grüne Fluoreszenz des Enzyms wird von einer eingebauten Kamera erfasst, und eine spezielle Software-App misst die Intensität, was zur Quantifizierung der Bakterienlast beitragen kann.
Das Team des IISc hat sich mit der Forschungsgruppe von Jayanta Haldar vom JNCASR zusammengetan, um seinen Ansatz an Urinproben zu testen. "Wir verwendeten Proben von gesunden Freiwilligen und fügten pathogene Bakterien hinzu, um Harnwegsinfektionen zu simulieren. Das Ergebnis wurde innerhalb von zwei Stunden erzielt", erklärt Maitra.
Als nächsten Schritt planen die Forscher, mit Krankenhäusern zusammenzuarbeiten, um diese Technologie mit Proben von Patienten zu testen.
Hinweis: Dieser Artikel wurde mit einem Computersystem ohne menschlichen Eingriff übersetzt. LUMITOS bietet diese automatischen Übersetzungen an, um eine größere Bandbreite an aktuellen Nachrichten zu präsentieren. Da dieser Artikel mit automatischer Übersetzung übersetzt wurde, ist es möglich, dass er Fehler im Vokabular, in der Syntax oder in der Grammatik enthält. Den ursprünglichen Artikel in Englisch finden Sie hier.